이번 포스트에서는 실험적으로 세포내의 RNA의 양을 정량할 때 가장 많이 사용되는 방법 중 하나인 qPCR(RT-qPCR)에 대해 알아보도록 할게요.
혹시 qPCR이 아니라 그냥 PCR과 관련된 내용이 궁금하시다면 아래 포스트를 참고해주세요.
2022.03.27 - [전공자를 위한 생물학/실험] - PCR(중합효소 연쇄반응)의 원리, 단계, 응용
PCR(중합효소 연쇄반응)의 원리, 단계, 응용
이번 포스트에서는 대부분의 생물학 실험실에서 다 일상적으로 하는 실험 중 하나인 PCR(polymerase chain reaction)에 대해 알아볼게요. 특히나 요즘은 생물 실험을 하시는 분들뿐만 아니라 코로나 검
unicellular.tistory.com

정량적 역전사 중합효소 연쇄 반응(Quantitative Reverse Transcription Polymerase Chain Reaction), 줄여서 RT-qPCR는 RNA를 검출하고 정량화하는 데 사용되는 실험 방법이에요. 먼저 전체 RNA 또는 mRNA가 상보적 DNA(cDNA)로 전사되며, 이 cDNA가 정량적 PCR(qPCR) 또는 실시간 PCR 반응의 템플릿으로 사용돼요. qPCR에서는 형광을 이용해 각 PCR 사이클마다 증폭된 산물의 양을 측정해요. RT-qPCR은 유전자 발현 분석, RNAi 검증, 마이크로어레이 검증, 병원체 검출, 유전자 검사, 질병 연구 등 다양한 분야에서 사용되죠.
one-step RT-qPCR과 Two-step RT-qPCR

RT-qPCR은 원스텝(one-step) 또는 투스텝(two-step) 방식으로 수행될 수 있어요. 원스텝 실험에서는 역전사와 PCR이 하나의 튜브와 완충액에서 수행되며, 역전사효소(reverse transcriptase)와 DNA 중합효소(DNA polymerase)가 함께 사용돼. 이 방식에서는 특정 서열에 대한 프라이머만을 사용해요.
반면, 투스텝 실험에서는 역전사와 PCR 단계가 별도의 튜브에서 수행되며, 각 단계마다 다른 최적화된 완충액과 반응 조건, 프라이밍 전략을 사용해요.
원스텝 RT-qPCR은 두 반응이 동일한 튜브에서 수행되므로 실험 간 변동이 적고, 피펫팅 단계가 줄어들어 오염 위험도 낮아요. 이 방법은 고처리량 증폭과 스크리닝에 적합하며, 빠르고 높은 재현성을 제공해요. 그러나 역전사와 PCR 반응을 개별적으로 최적화할 수 없고, 두 반응 조건이 절충되어 민감도가 다소 낮아질 수 있어요. 또한, 샘플당 검출 가능한 타겟의 수가 제한되죠.
투스텝 RT-qPCR은 안정적인 cDNA 풀(pool)을 생성하여 장기간 보관이 가능하고, 여러 번의 반응에 활용할 수 있어요. 동일한 cDNA 풀에서 타겟 유전자와 참고 유전자를 멀티플렉싱 없이 증폭할 수 있고, 각 반응에 맞는 최적화된 완충액과 반응 조건을 사용할 수 있어요. 또한, 유연한 프라이밍 옵션을 제공해요. 하지만 여러 튜브와 피펫팅 단계로 인해 DNA 오염의 위험이 커지고, 시간이 더 소요되며, 원스텝보다 최적화가 더 필요하죠.
역전사 (reverse transcriptation)
RT-qPCR 실험 설계 시, 총 RNA(total RNA) 또는 mRNA 중 어느 것을 역전사의 주형으로 사용할지 결정하는 것이 중요해요. mRNA는 약간 더 높은 민감도를 제공할 수 있지만, 총 RNA는 초기 재료로 사용할 때 몇 가지 중요한 장점이 있어요. 첫째, 정제 단계가 적어 주형의 정량 회수가 더 안정적이고, 시작한 세포 수에 대한 결과를 더 잘 정규화할 수 있어요. 둘째, mRNA 농축 단계를 생략함으로써 서로 다른 mRNA의 회수율 차이로 인해 결과가 왜곡될 가능성을 줄일 수 있어. 종합적으로, 대부분의 응용에서는 절대적인 민감도보다 타겟의 상대적 정량화가 더 중요하기 때문에 총 RNA가 더 적합해요.

역전사를 시작하려면 짧은 DNA 올리고뉴클레오타이드(oligonucleotide)인 프라이머가 필요해요. 프라이머는 템플릿 RNA 가닥에 결합하여 역전사효소가 합성을 시작할 수 있는 출발점을 제공하죠. 투스텝 방식의 cDNA 반응에서는 네 가지 프라이밍 방법이 사용될 수 있어요: 올리고(dT) 프라이머, 랜덤 프라이머, 서열 특이적 프라이머(sequence-specific primers), 또는 이들의 혼합(그림 2 및 표 2 참조). 올리고(dT)와 랜덤 프라이머를 혼합하면 cDNA의 잘린 조각(truncated cDNA) 생성을 줄여 역전사 효율과 qPCR 민감도를 높이는 데 도움이 돼요.
oligo dT 프라이머
올리고(dT) 프라이머는 티민(Thymine) 염기들이 연속적으로 배열된 구조로, mRNA의 폴리(A) 꼬리에 결합해요. 앵커드 올리고(dT) 프라이머는 3′ 말단에 하나의 G, C, 또는 A 염기를 추가하여 특정 위치에 결합되도록 보장해 줘요. 이 프라이머는 폴리(A) 꼬리를 가진 mRNA로부터 full length cDNA(모든 정보를 다 함유하고 있는 cDNA)를 생성하는 데 유리하고, 시작 물질이 적을 때 사용하기 좋아요. 앵커는 올리고(dT) 프라이머가 mRNA의 폴리(A) 꼬리 5′ 끝에 결합하게끔 보장해요. 그러나 폴리(A) 꼬리를 가진 유전자만 증폭이 가능하며, 내부 폴리(A) 영역에서 프라이밍이 일어날 경우 cDNA가 잘려 나갈 수 있습니다. 또한 3′ 끝으로 치우친 증폭이 일어날 수 있지만, 앵커드 올리고(dT)를 사용할 경우 이러한 단점이 줄어들어요.
random 프라이머
랜덤 프라이머는 6~9개의 염기로 이루어져 있어 RNA 전사체 곳곳에 결합해요. 모든 RNA(tRNA, rRNA, mRNA)에 결합하기 때문에 시작 물질이 적을 때나, 이차 구조가 있는 전사체에 유용해요. 높은 수율의 cDNA를 생성할 수 있지만, 모든 RNA로부터 cDNA를 생성하기 때문에 특정 mRNA 신호가 희석될 수 있어요. 또한, 잘린 cDNA가 생성될 가능성도 있죠.
서열 특이적 프라이머
서열 특이적 프라이머는 특정 mRNA 서열을 타겟으로 하는 맞춤형 프라이머에요. 이를 사용하면 특정 cDNA 풀을 생성할 수 있어 민감도가 높아져요. qPCR에 사용할 역 프라이머를 활용할 수 있어요. 하지만 원하는 하나의 특정 유전자만 증폭할 수 있다는 제한이 있죠.
역전사효소 (reverse transcriptase enzymes)
역전사효소(reverse transcriptase, RT)는 RNA를 DNA로 전환하는 효소에요. 일부 역전사효소는 전사 후 RNA-DNA 하이브리드에서 RNA 가닥을 분해하는 RNase 활성(RNase activity)을 가지고 있어요. RNase 활성이 없는 효소를 사용할 경우, qPCR 효율을 높이기 위해 RNase H를 추가할 수 있어요. 흔히 사용하는 역전사효소로는 Moloney murine leukemia virus reverse transcriptase와 Avian myeloblastosis virus reverse transcriptase가 있는데요. RT-qPCR을 위한 역전사효소로는 높은 열 안정성을 가진 효소가 이상적이에요. 이는 cDNA 합성을 더 높은 온도에서 수행할 수 있게 하여 이차 구조가 많은 RNA를 성공적으로 전사할 수 있도록 돕고, 반응 동안 효소가 충분히 활성 상태를 유지해 더 높은 수율의 cDNA를 생성할 수 있어요.
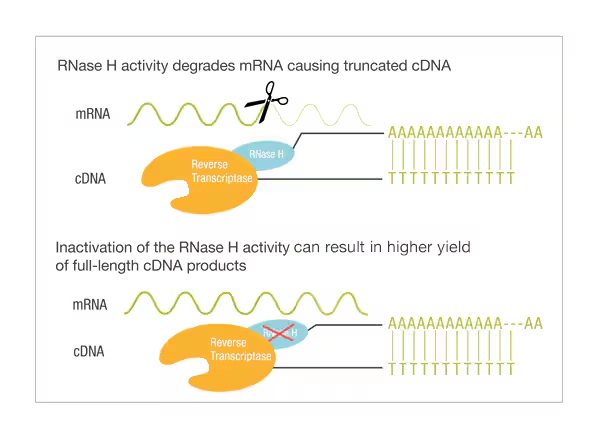
RNase H 활성은 RNA-DNA 복합체에서 RNA를 분해하여 이중가닥 DNA의 효율적인 합성을 가능하게 해요. 그러나 긴 mRNA 템플릿의 경우 RNA가 조기에 분해되어 잘린 cDNA가 생성될 수 있어요. 따라서 긴 전사체를 생성하여 cDNA 클로닝을 목표로 할 때는 RNase H 활성을 최소화하는 것이 유리해요. 반면, qPCR에서는 RNase H 활성을 가진 역전사효소가 RNA-DNA 복합체의 해리를 촉진해 초기 PCR 사이클 동안 효율성을 높이기 때문에 선호되기도 해요. (위 그림 참고)
RT-qPCR 프라이머 설계
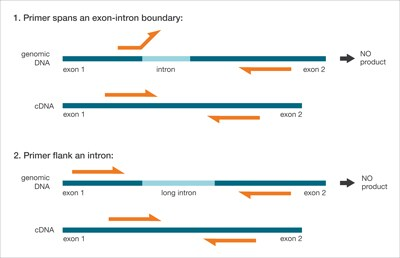
RT-qPCR의 qPCR 단계에서 사용할 PCR 프라이머는 이상적으로 엑손-엑손(exon-exon) 접합 부위를 포함하도록 설계해야 해요. 하나의 증폭 프라이머가 엑손-인트론(exon-intron) 경계를 가로지르는 방식으로 설계할 수도 있어요(위 그림 참고). 이 설계는 염색체 DNA(gDNA) 오염으로 인한 거짓 양성 위험을 줄여줘요. 염색체 DNA에는 인트론이 포함되어 있기 때문에, 이 디자인을 사용하면 오염된 염색체 DNA가 증폭되는 것을 방지할 수 있어요.
모든 RT-qPCR 실험에는 "마이너스 역전사효소 컨트롤(no RT control)"을 포함해야 해요. 이 컨트롤은 오염된 DNA(예: gDNA 또는 이전 실험의 PCR 산물)를 확인하는 데 사용되며, 역전사효소를 제외한 모든 반응 구성 요소를 포함합니다. 이 컨트롤에서는 역전사가 발생하지 않으므로, 만약 PCR 증폭이 관찰된다면 이는 오염된 DNA에서 유래한 가능성이 높아요.
이번 포스트에서는 RT-qPCR에 대해 알아봤어요. 추가적으로 궁금한 점 혹은 디테일한 점들을 질문해주시면 그와 관련된 포스트를 제작해보도록 할게요. 즐거운 실험하세요!
'전공자를 위한 생물학 > 실험' 카테고리의 다른 글
| ELISA의 원리, 단계, 응용 - 2 (0) | 2024.11.01 |
|---|---|
| ELISA의 원리, 단계 응용 - 1 (0) | 2024.10.30 |
| 유세포 분석(flow cytometry)의 원리, 단계, 응용 - 6 (1) | 2024.10.27 |
| 유세포 분석(flow cytometry)의 원리, 단계, 응용 - 5 (0) | 2024.10.27 |
| 유세포 분석(flow cytometry)의 원리, 단계, 응용 - 4 (0) | 2024.10.27 |