이번 포스트부터는 단백질의 기능에 대해 중점적으로 알아보자.
protein이 제대로 된 기능을 하기 위해서는 다른 요소들과의 interaction이 필수적으로 필요함. 따라서 이들 interaction에 대한 regulation도 이루어짐.
protein, 그 중에서도 흔히 관찰되는 globular protein들은 아래와 같이 다양한 function을 가지고 있음.

대표적으로 myoglobin의 경우 O2를 storage하는 역할을, hemoglobin의 경우 O2를 transport하는 역할을 수행함.
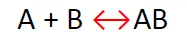
생체 내 대부분의 반응들은 위와 같이 reversible함. (물론 일부는 irreversible하기도 함)
한편 protein에 붙는 molecule을 통칭해서 ligand라 부름. ligand는 일반적으로는 매우 작은 molecule이지만 때때로 엄청 큰 녀석이 ligand로 작용하기도 함. 그리고 protein 중 ligand와 결합하는 결합부위를 일컬어 binding site라 부름.
ligand와 protein간의 binding은 대부분의 경우 noncovalent interaction에 의해 이루어짐(물론 예외는 있음. 그러나 대부분의 ligand-protein interaction 반응은 reversible해야 하므로 너무 강하지도 않고 너무 약하지도 않게 결합하는 것이 유리할 것임).
이제 ligand, protein간의 binding 과정을 조금 더 수식적으로 묘사해보자.
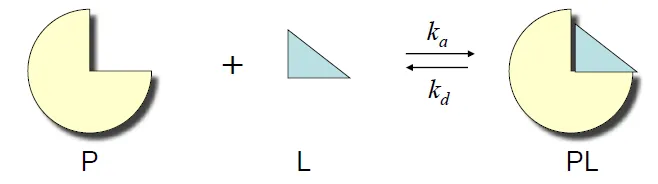
위 그림에서 k로 표시된 것은 rate constant임. 그 중 ka는 complex를 빨리 만드는 정도를 나타내주는 association rate constant이고, kd는 dissociation rate constant임.
한편 시간이 지나서 protein에 딱 적당한 양의 ligand가 붙게 되면 equilibrium에 도달하게 됨. 이 equilibrium 상태에서는 아래와 같은 수식이 성립함.
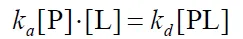
한편 equilibrium 상태에서의 경향을 통해 ligand, protein의 binding 정도, 즉 결합력을 알 수 있음.
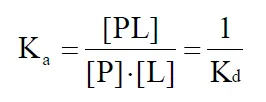
위 수식에서 Ka는 equilibrium association constant이며, Kd는 equilibrium dissociation constant임. 즉 다시말해 Ka는 ligand와 protein이 얼마나 강하게 붙어있는지, Kd는 ligand와 protein이 얼마나 강하게 떨어져 있는지를 나타내줌.
참고로 위 수식과 앞서 유도한 k 관련 수식을 합치면, Ka = ka/kd의 관계식이 성립한다는 사실 또한 알 수 있음.
방금의 개념들을 이용하면 ligand와 실제로 붙어있는 protein의 비율, 즉 fraction of occupied binding site Y를 계산할 수 있음.
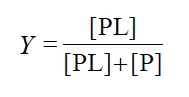
일단 정의에 따라 Y는 위와 같이 표현 가능함. 그런데 이 때 앞서 소개한 Ka 수식을 이용해 위 Y에 관한 수식 내의 [PL]을 다른 식으로 바꾼 뒤 수식을 예쁘게 정리하면 아래와 같이 Y가 정리됨.
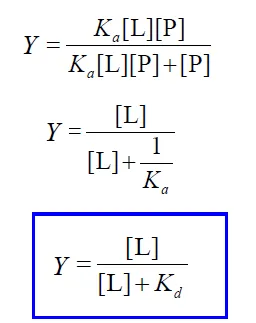
그런데 이 수식에서 만약 [L]이 Kd라면, 이 경우 Y=0.5임. 즉 다시말해 protein 중 절반이 ligand와 결합했을 때의 ligand 농도 [L]은 Kd임.
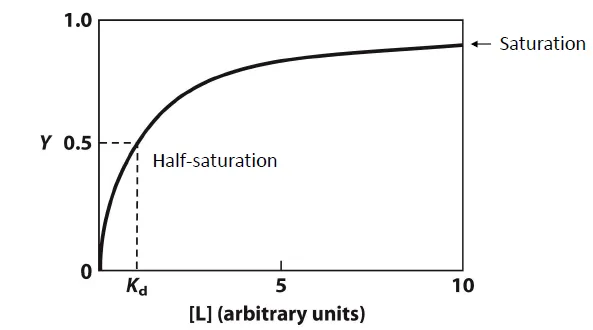
실제로 방금 구한 Y식을 이용해 [L]-Y graph를 그려보면 위와 같음. 이 때 당연히 [L]을 무한정 늘리면 결국 특정 Y 값(1)으로 saturation될 것임. 한편 Y=0.5일 때는 앞서 살펴본 것처럼 [L]이 Kd가 될 것이고, 이 상태를 half-saturation이라 부를 수 있음.
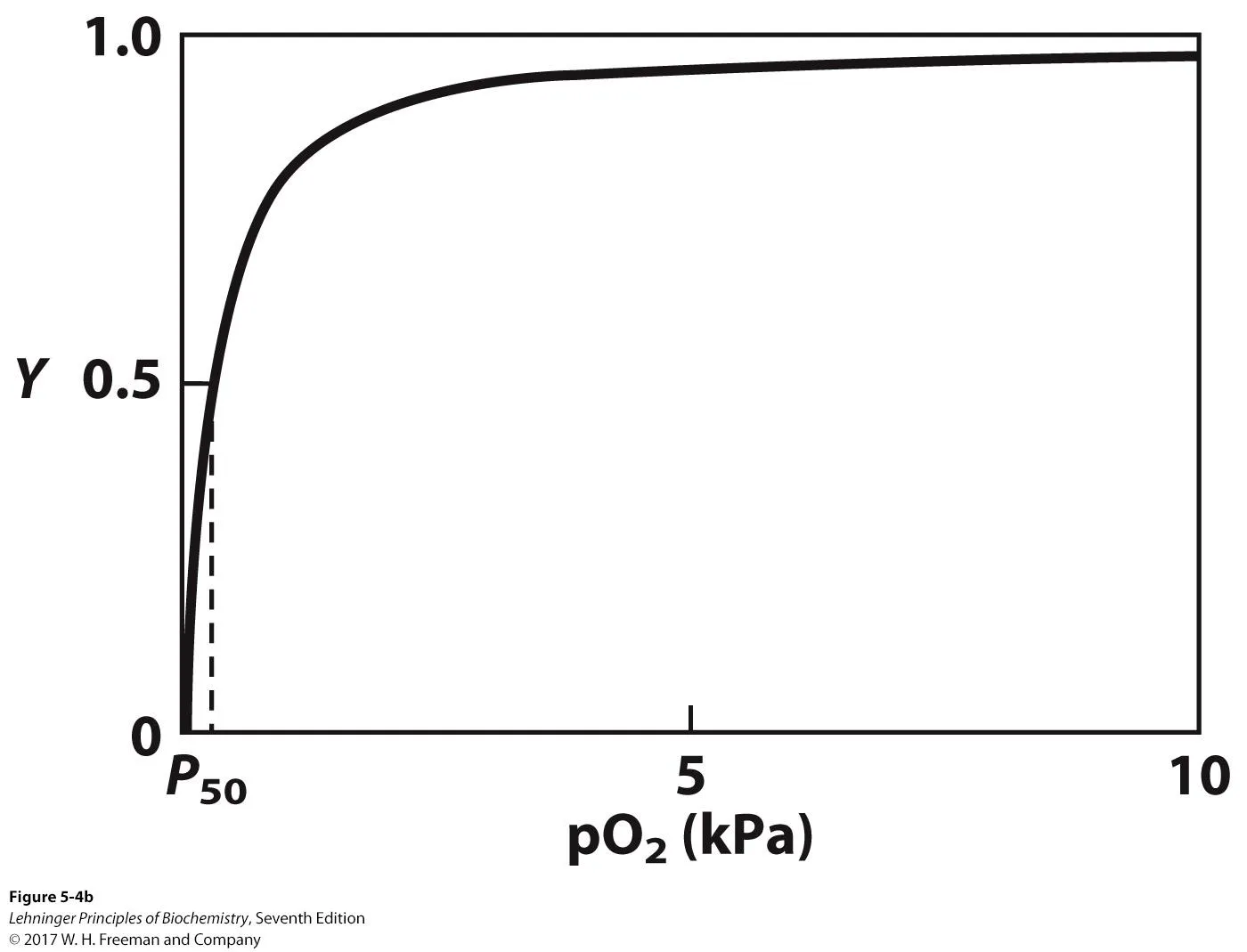
위 graph는 실제로 myoglobin의 oxygen binding 정도를 그려본 것임. 이 때 x축으로 산소의 농도 대신 partial pressure를 사용함. 다만 이는 농도와 비슷하게 생각해도 무방함.
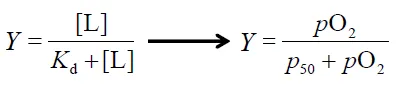
그렇기에 사실 엄밀히 말하면 myoglobin-O2 관계에서의 Y 식은 위와 같이 표현 가능함. (이 때 Kd는 partial pressure 50, 즉 p50으로 표현 가능함)
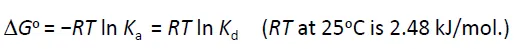
참고로 protein, ligand가 서로 interaction하면서 변화되는 energy는 위와 같은 수식으로 계산될 수 있음.
그 밖에, 잘 생각해보면 Kd가 작을수록(대략 10nM보다 작으면) P-L간의 결합이 strong하고, Kd가 클수록(대략 10μM보다 크면) P-L간의 결합이 weak하다는 것도 알 수 있음.
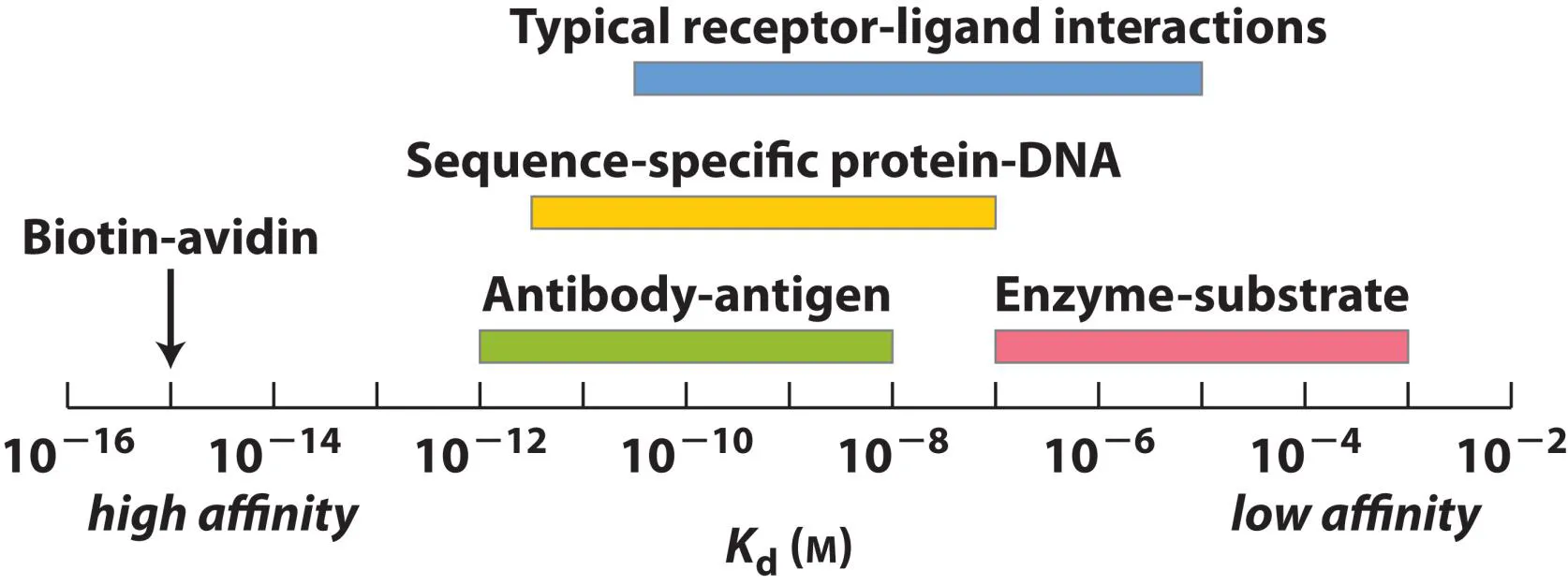
위 그림에는 각종 P-L들 간의 Kd가 나타나 있음. 보면 보통의 enzyme-substrate의 경우 low affinity를 가지고, antibody-antigen의 경우 중간 정도의 affinity를 가지며, biotin, avidin의 경우 상당히 높은 affinity를 가짐을 알 수 있음. (이 때 각각의 range가 넓은 이유는 다시 떨어지는 것이 얼마나 고려되는지에 따라 Kd값이 달라질 수 있기 때문임)
다음으로 protein-ligand에서의 specificity에 대해 알아보자. 이와 관련된 2가지 model이 존재함.
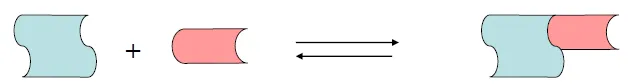
위 그림에는 Emil Fisher가 주장한 lock and key model(열쇠 자물쇠 모델)이 나타나 있음.
이를 이해하기 위해서는 complementarity에 대해 이해해야 함. 쉽게 말해 이 개념은 protein과 ligand 사이에 size, shape, charge, hydrophobicity(hydrophilicity) 측면에서 잘 맞는 정도를 의미함.
lock and key model에서는 사실 protein과 ligand가 결합하기 이전부터 이미 서로 complementary한 구조가 형성되어 있음. 다만 실제로 lock and key model에 의해 설명 가능한 protein-ligand interaction은 극소수임.
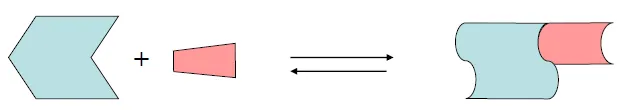
위 그림에 나타난 개념은 induced fit(유도적합모델)임. (Daniel Koshland에 의해 주장) 이 경우 보면 ligand와 protein들이 서로 근처로 와서 결합하는 과정에서(물론 처음에도 일부 region들은 complementary하지만) dynamic하게 구조가 변형되며(induced) energy적으로 더 안정한 interaction을 형성한 결과 서로에게 더더욱 complementary한 구조가 되어 fit한다는 model임. 대부분의 protein은 이 model에 의해 ligand과 결합하는 것으로 보임.
다음 포스트부터는 globin 단백질을 통한 산소의 운반 과정에 대해 단백질 기능의 측면에서 자세히 알아보도록 하자.
'전공자를 위한 생물학 > 생화학' 카테고리의 다른 글
| [생화학] 4.3 : 헤모글로빈(hemoglobin) - 1 (0) | 2024.11.06 |
|---|---|
| [생화학] 4.2 : 미오글로빈(myoglobin) (0) | 2024.11.06 |
| [생화학] 3.6 : 단백질 항상성(proteostasis) - 2 (0) | 2024.11.05 |
| [생화학] 3.6 : 단백질 항상성(proteostasis) - 1 (0) | 2024.11.05 |
| [생화학] 3.5 : 단백질의 구조 결정 방법 (0) | 2024.11.05 |