이번 포스트에서는 spliceosome cycle에 대해 알아보자.
앞서 말한 것처럼 spliceosome은 많은 protein들과 RNA들로 이루어져 있음. 이 component들은 stepwise로 assemble되는데, stepwise assemble이 이루어지기에 각 단계에서의 assemble을 다양한 신호가 regulation하여 단계별로 splicing의 quality, quantity를 control해 gene expression을 조절할 수 있게 됨.
우리가 이러한 regulation에 대해 제대로 이해하려면 결국 spliceosome cycle에 대해 이해해야 함.
이와 관련된 실험을 살펴보자.
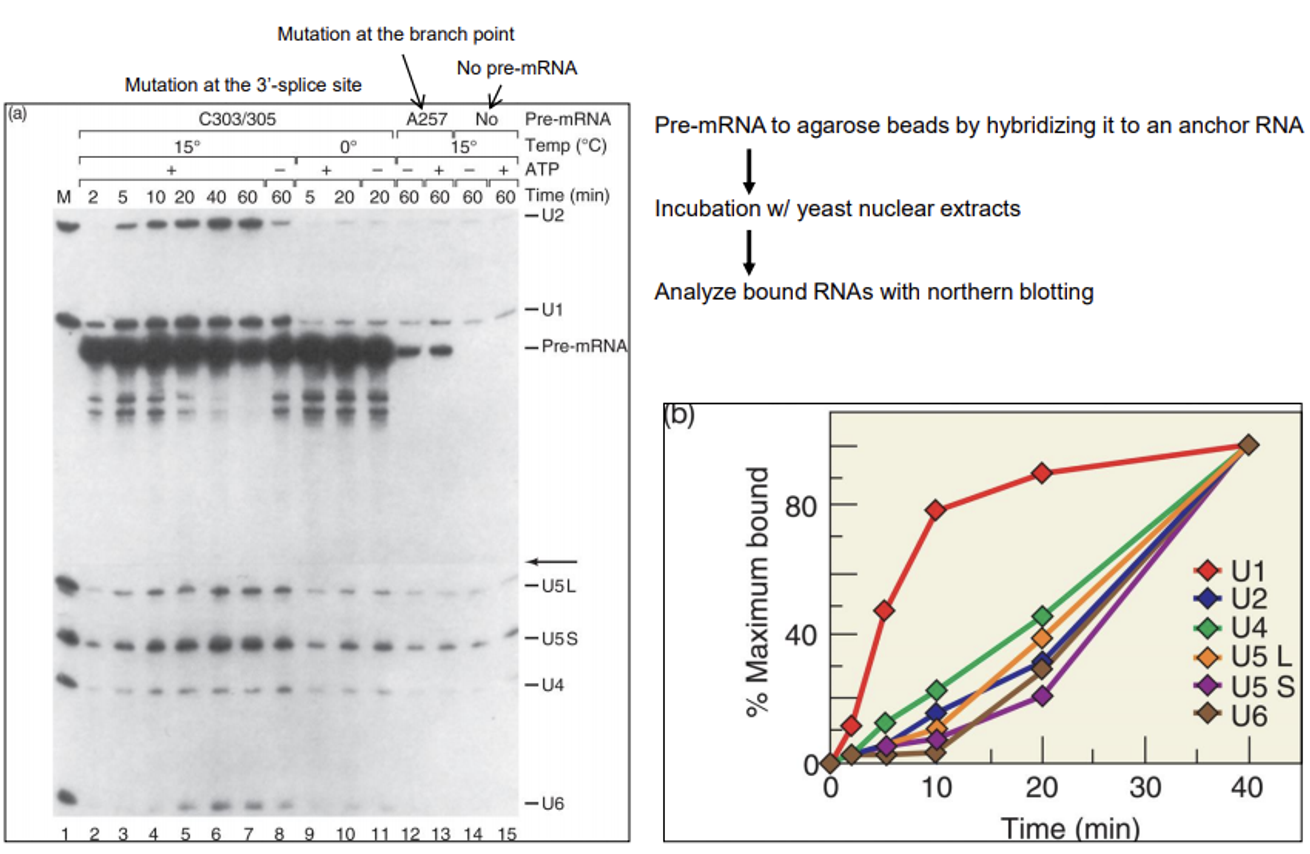
이 경우 pre-mRNA를 붙인 agarose bead에 nuclear extract를 통과시킴. 이 때 bead 표면에 있는 pre-mRNA에 어떤 녀석들이 붙어서 splicing을 시키는지를 nothern blot으로 시간대별로 분석한 결과가 위 그림 왼쪽에 나타나 있음.
참고로 lane 14, 15는 pre-mRNA를 넣어주지 않은 negative control이므로, 이 때의 결과를 바탕으로 나머지 결과들을 비교해보면 됨. 그리고 C303/305의 경우 3' splice site에 mutation이 생겨 있어서 splicing이 끝나더라도 3' intron 부분이 떨어져 나가지 못하게 막아준 상태임. (이게 떨어져버리면 bead에서 RNA가 탈출해버리게 되므로 이런 조작을 해 준 것)
보면 U1에 해당하는 band가 가장 이른 시간(2)부터 관찰되기 시작하며 뒤이어 나머지 component들이 붙게 됨. 이를 graph로 나타낸 것이 위 그림 오른쪽 아래와 같음. (U1이 제일 먼저, 그리고 제일 많이 들어온다는 것이 핵심)
한편 lane 7과 8을 비교해볼 시 이 때는 time이 동일하고 ATP를 넣어주었냐(7) 넣어주지 않았냐(8)만 다름. 그런데 결과를 봤을 때 U2의 band 두께만 얇아지는 것을 확인 가능함. (나머지는 거의 그대로) 이를 통해 U2가 ATP가 있어야 더 잘 assemble되는 요소라는 것을 추정할 수 있음.
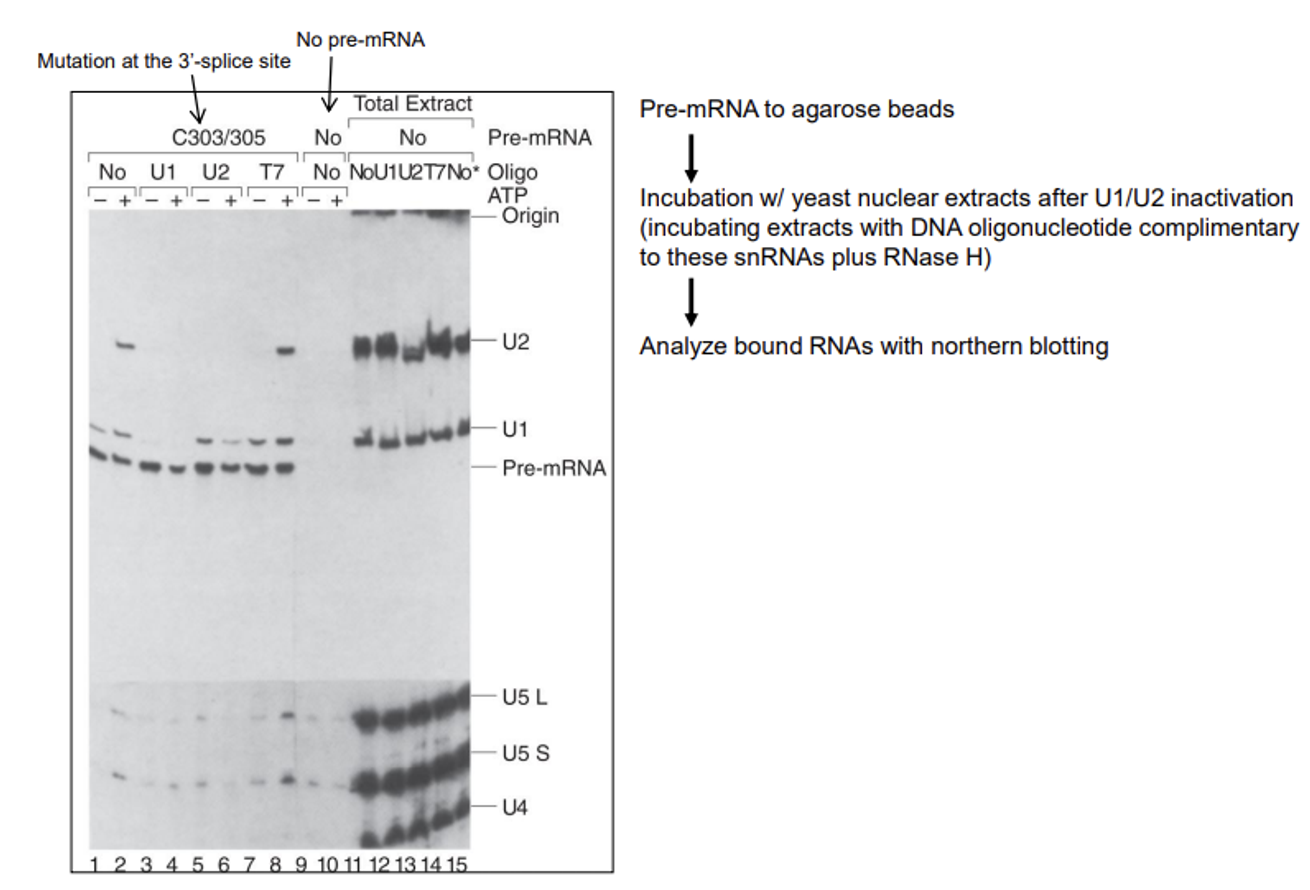
spliceosome의 assembly 과정을 더 잘 보여주는 실험 결과가 위에 나타나 있음. 이 실험의 경우 앞서와 기본 구성은 동일하지만 U1, U2 각각을 없애봤음. (이들을 없애기 위해 앞서 사용했던 방법과 유사하게 U1, U2 snRNA에 상보적으로 결합 가능한 oligonucleotide를 넣어준 뒤 RNase H를 처리해줘 이들을 분해시켜줌)
결과를 살펴보자. lane 11~15까지를 보면 U1의 경우 U1에 상보적인 oligo를 넣어줬을 때(12) band가 아래쪽으로 더 쳐진다는 것을 확인할 수 있고(RNase H에 의해 특정 부위가 분해된 것) U2의 경우에도 U2에 상보적인 oligo를 넣어줬을 때(13) band가 아래쪽으로 더 쳐진다는 것을 확인할 수 있음. 즉, RNase H에 의한 반응이 제대로 일어난 것을 확인 가능함. 한편 lane 9, 10은 negative control이므로 이와 나머지 데이들을 비교해가면서 보면 됨.
lane 3을 보면 U1이 없을 때 U1, U2, U5에 해당하는 band가 다 관찰되지 않음. 한편 lane 5를 보면 U2가 없을 때 U2, U5 band는 관찰되지 않지만 U1 band는 관찰됨. 이 결과를 통해 U1→U2→U5 순으로 assemble되며 이전 component들이 다음 component의 assemble을 도와주는 역할을 한다는 것을 알 수 있음.
뒤이어 다시 살펴보겠지만 맨 처음 splicing substrate에 assemble되는 U1에 의해 형성되는 complex의 이름이 commitment complex(CC)임. 이 때 commitment complex란 일단 특정 단백질(U1)이 특정 RNA에 붙으면 딱 그 RNA를 targeting해서 다른 곳으로 가지 않고 거기서 splicing을 일으키는 complex를 의미함. (다시말해 U1에 의해 protein이 특정 RNA에서 떨어지지 않고 끝까지 splicing을 수행하게 됨)
한편 일단 U1이 붙고 나면 U2와 같은 녀석들이 complex에 오게 되고, 뒤이어 많은 element들이 모임. (참고로 앞서 말했듯 U2 binding에는 ATP가 필요함) 참고로 U6의 경우 U4에서 떨어져 나오고 나면 U1의 자리를 displace하여 5' splice site에 결합함. (이 step은 ATP-dependent함)
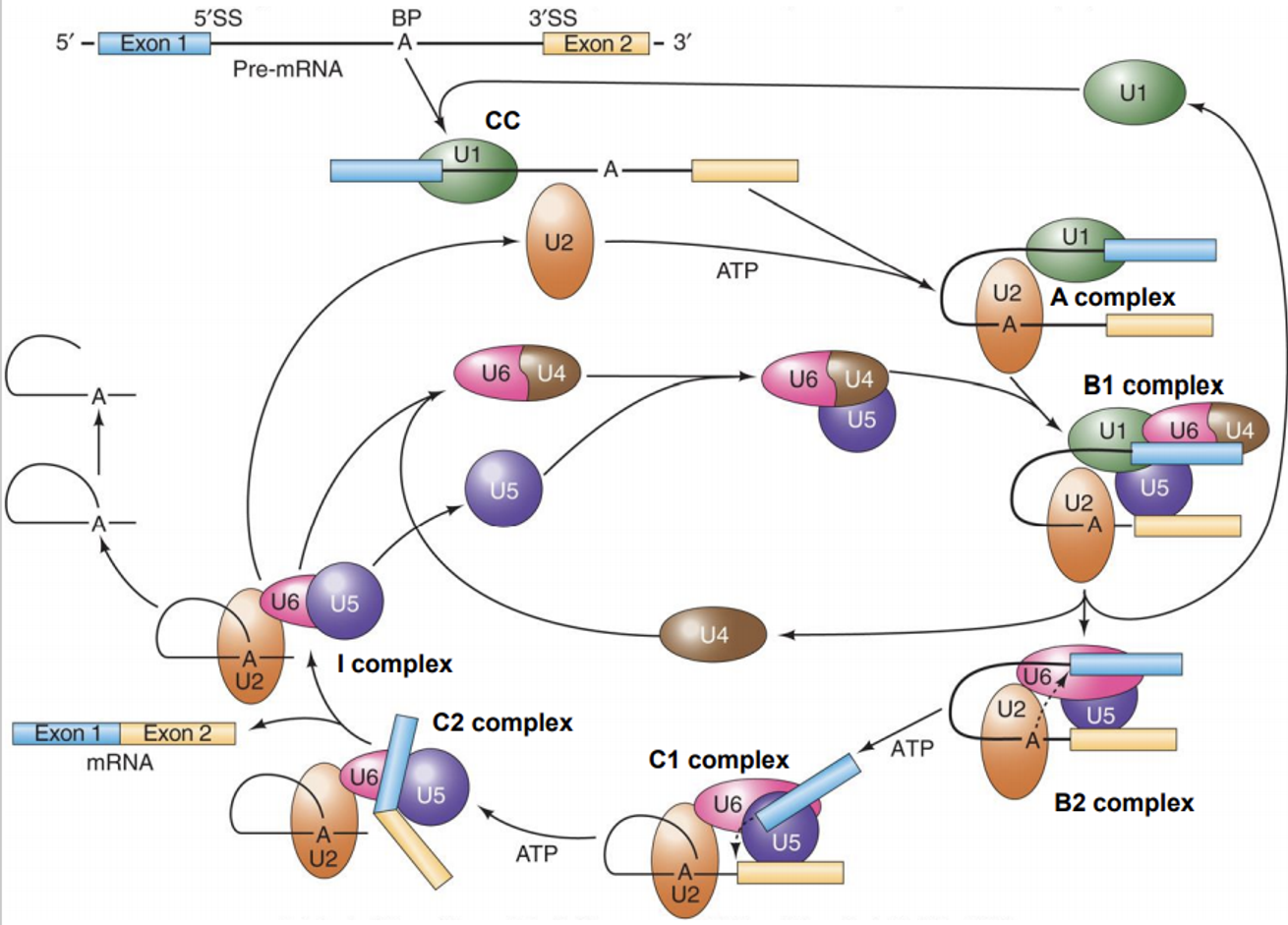
전체 spliceosome cycle을 정리한 것이 위 그림과 같으므로 참고할 것.
모든 snRNP들은 동일한 set의 7개의 Sm protein을 가지고 있음. Sm protein은 snRNA에 있는 AAUUUGUGG로 구성된 common Sm site에 결합할 수 있음.

위 그림에 U1 snRNP의 구조가 나타나 있음. (위 그림에서 Sm protein이 가지는 가운데 구멍 부분에 snRNA가 들어가 결합하게 됨) 참고로 이 구조는 요즘 구조생물학 연구에서 핫한 기술 중 하나인 single particle cryo-EM을 이용해 시각함.
한편 지금까지 배운 common한 spliceosome 이외에 minor한 spliceosome들도 존재하는데, 이 녀석들의 경우 intron sequence가 다양하다 보니 결국 이 다양한 intron sequence에 맞는 다양한 snRNP들로 구성되어 있음.
다음 포스트부터는 splice site selection에 대해 알아보자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 14.6 : RNA polymerase II에 의한 splicing 조절 (0) | 2023.12.19 |
|---|---|
| [분자생물학] 14.5 : splice site selection (0) | 2023.12.19 |
| [분자생물학] 14.3 : splicing에서 snRNA의 역할 (0) | 2023.12.19 |
| [분자생물학] 14.2 : splicing의 분자적 작동원리 - 2 (0) | 2023.12.18 |
| [분자생물학] 14.2 : splicing의 분자적 작동원리 - 1 (0) | 2023.12.18 |