이번 포스트에서는 지난 포스트에 이어 각각의 snRNP가 어떤 역할을 하는지에 대해 알아보도록 하자.
U1 snRNP
U1 snRNP는 splicing 시 가장 먼저 assemble되는 녀석 중 하나임.
U1 snRNP를 구성하는 U1 snRNA를 분리해 sequencing을 진행해본 결과, 5', 3'-splice site consensus sequence와 U1 snRNA간에 상보성이 관찰됨. 이 때문에 처음에는 U1 snRNA가 단순히 두 개의 exon을 직접적으로 이어주는 역할을 하지 않을까 생각했었음. 그러나 branchpoint의 존재가 발견되면서 이 생각은 맞지 않음을 알게 됨.
아래에 나타나 있는 실험은 U1 snRNA의 역할을 알아내기 위해 수행한 실험임.
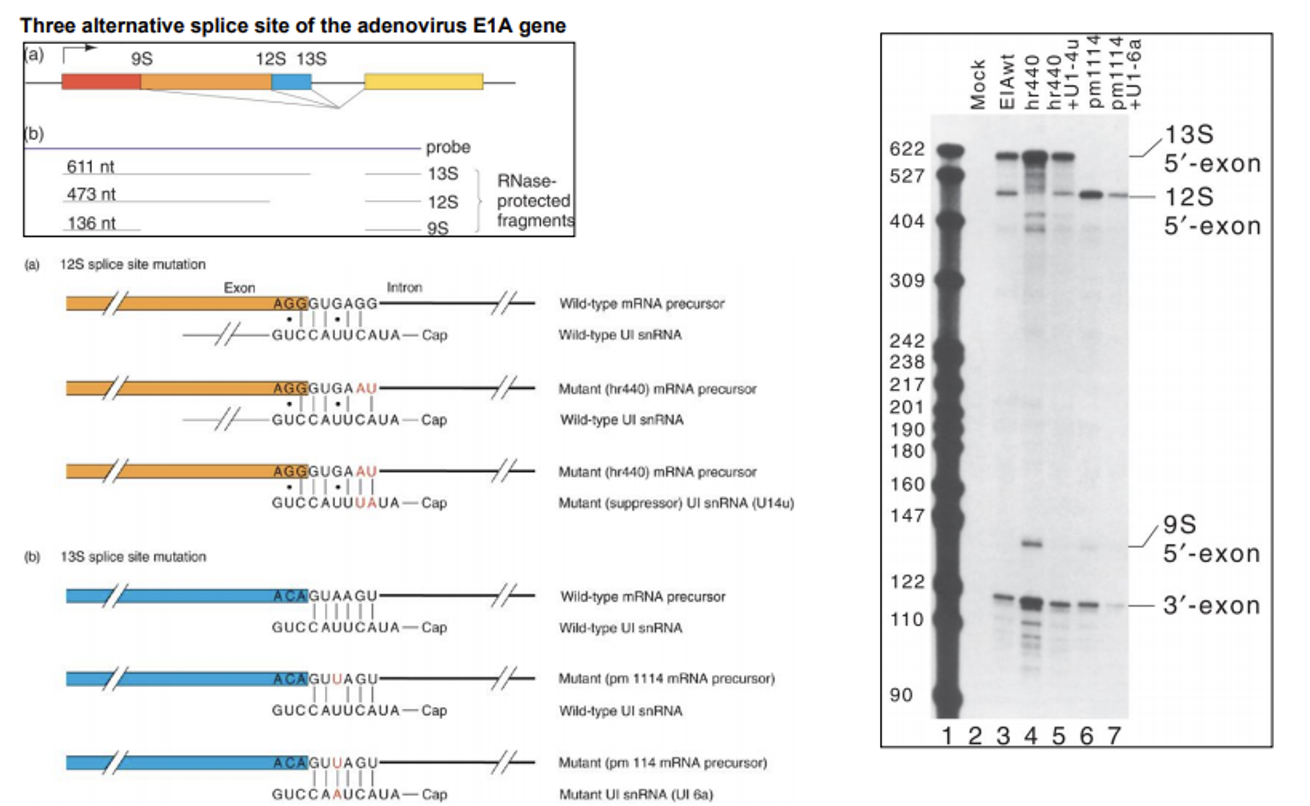
이 실험에서는 특히 U1 snRNA와 5'-splice site(즉, intron의 5' 끝부분)간의 상동성에 대해 알아봄.
이 때 사용한 gene이 adenovirus의 E1A gene인데, 이 gene은 위 그림 왼쪽 위에 나타난 것처럼 alternative splicing될 수 있어서 어떨 때는 빨간색과 노란색이 붙을 수도 있고(9S), 혹은 주황색과 노란색이 붙을 수도 있고(12S), 파란색과 노란색이 붙을 수도 있음(13S).
이제 이 gene으로부터 생합성되고 splicing된 mature mRNA에 대해서 실험을 진행하게 되는데, 이 때 위 그림 왼쪽 위 (b)와 같이 probe와 함께 RNase-protection assay를 수행함.
이 방법은 single strand RNA를 분해하는 RNase를 처리해서 마치 S1 mapping과 유사하게 RNA-RNA hybrid를 형성한 부분만 남겨놓는 method임. 이 방법을 사용하게 되면 결국 얻어지는 fragment의 크기를 통해 어떤 종류의 alternative splicing 생성물이 발생했는지를 확인할 수 있음.
우선 lane 3번(WT)의 결과를 살펴보자. 보면 13S에 해당하는신호가 강하게 관찰되었고, 12S에 해당하는 신호도 관찰되었으며, 9S에 해당하는 신호는 거의 관찰되지 않음. (9S 신호가 거의 관찰되지 않는 것은 그냥 E1A gene 자체의 특성인 것으로 보임)
다음으로 위쪽 그림 왼쪽 아랫부분에 나타나 있는 (a)에 대해 살펴보자. 이 경우 12S(주황색) 5' splice site에 mutation을 유발해봄. (a)의 두 번째에 나타나 있는 mutation의 경우 U1 snRNA는 그대로 두고 mRNA precursor의 5' site 근방의 서열을 GG에서 AU로 바꿔준 것임.
이렇게 바꿔주게 되면 원래 base pairing을 이루던 G-C 조합에서 base pairing이 일어나지 않는 대신 그 뒤쪽의 U-A간에 base pairing이 일어나게 됨. 언뜻 봐서는 둘에 무슨 차이가 있는지 모를 수 있지만, base pairing이 WT처럼 인접해서 연속적으로 이루어져 있는 경우 매우 강한 base pairing이 일어나는 반면 중간에 빈 부분이 있는 경우 상대적으로 base-pairing 세기가 약함.
이렇게 mutation을 유발했을 때의 결과가 lane 4에 나타나 있음. 보면 13S의 신호는 전보다 더 강하게 관찰되는 반면 12S 신호는 거의 관찰되지 않고, 9S 신호가 관찰됨. 이를 통해 12S 근방의 5' splice site에 mutation을 유발할 경우 12S splicing이 잘 안일어나게 된다는 것을 알아냄.
한편 (a)의 3번째에 나와있는 것처럼 이번에는 앞서 유발했던 mutant mRNA precursor와 추가적인 base pairing을 할 수 있게 변형된 U1 snRNA를 사용해줌. (추가적으로 A-U pairing이 일어나도록 해줌) 그랬더니 lane 5에 나타난 것과 같이 WT과 거의 유사한 결과가 나타남. 즉, U1 snRNA의 서열 그 자체보다는 5' splice site와의 상보적인 결합이 splicing을 촉진하는 것처럼 보임.
그러나 위 그림 왼쪽 아래에 나타난 (b) mutation 결과를 보면 그렇지만도 않음. 물론 이 경우에도 13S 부분 5' splice site에 mutation을 유발한 경우 lane 6에 나타난 것처럼 이번에는 13S 신호가 잘 관찰되지 않고 12S, 9S 신호는 관찰됨.
그러나 이번에도 앞서와 마찬가지로 U1 snRNA를 변형시켜 mutated된 5' splice site와 상보적으로 base pairing을 할 수 있게 해줬는데도 lane 7처럼 별로 달라진 점이 없이 여전히 13S 신호가 관찰되지 않음.
이를 통해 U1 snRNA와 hnRNA 사이의 complementary한 base pairing도 물론 중요하지만, 어떤 경우에는 U1 snRNA의 '서열' 그 자체가 중요하게 작용하기도 한다는 것을 알 수 있음.
이런 결과 때문에 intron의 5' 말단을 인지하는 다른 녀석이 있어서 U1의 작용을 도와주는 것이 아닌가를 의심하게 되었고, 결과적으로 U6 snRNP가 발견됨.
U6 snRNP
U6 snRNP는 5' splice site를 인지하는 역할을 수행하며, 실제로 5' splice site와의 상보적인 결합이 가능한 U6 snRNA를 가지고 있음. U6 snRNP는 U1에 의해 매개되는 splicing의 first step 이후에, 그러나 lariat intermediate가 형성되기는 전에 작용함.

위 그림에 U6 snRNA가 나타나 있으므로 참고할 것.
한편 U6는 splicing 과정에서 U2와도 interaction을 하게 됨.
U2 snRNP
U2 snRNA는 splicing branchpoint의 conserved sequence와 base-pair를 이룸. 그리고 이 base pairing이 splicing에 있어 매우 핵심적인 역할을 함.
이와 관련된 실험 결과를 살펴보자.
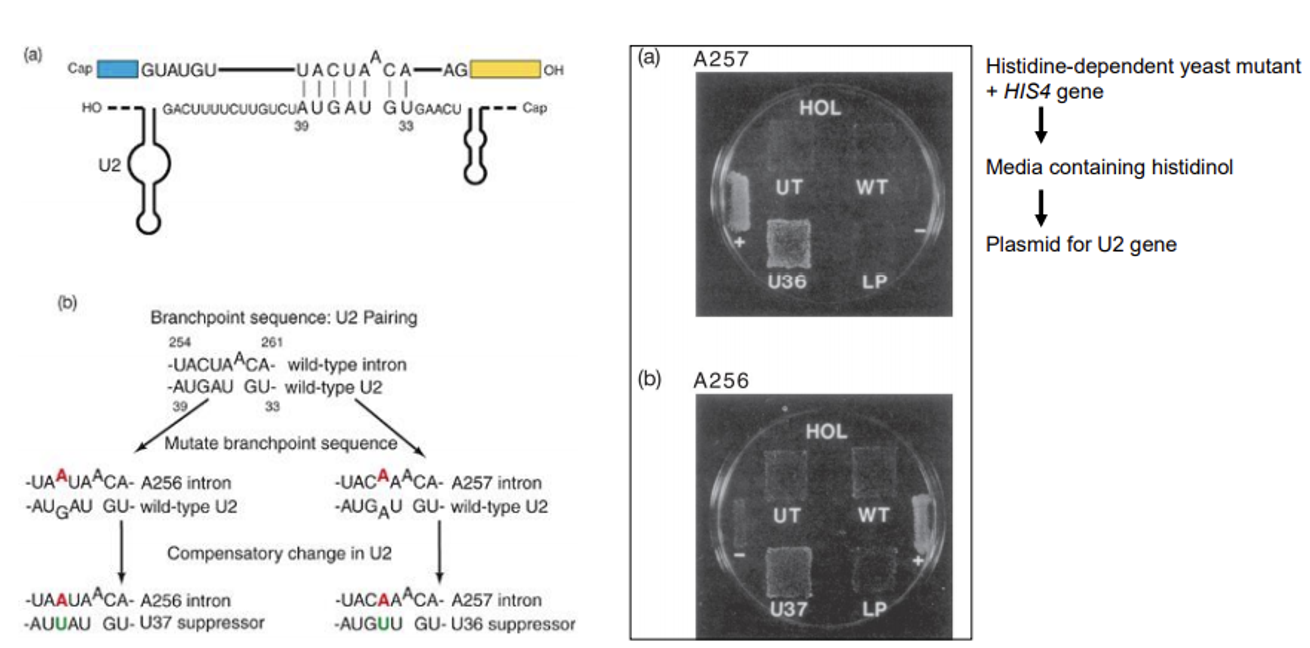
우선 이 실험에 사용한 yeast system에 대해서부터 논해보자. 이 실험에 사용한 yeast는 histidine-dependent yeast mutant로 이 mutant는 histidine이 없는 환경에서 자라지 못하고 죽어버림. 그런데 이 mutant에 HIS4 gene을 삽입해주면 media가 (histidine 없이) histidinol만 가지고 있어도 이 mutant는 살아갈 수 있게 됨.
한편 본 실험의 목적은 splicing이 잘 이루어지는지 여부를 확인하기 위함이므로 HIS4 gene을 잘 design하여, HIS4 gene으로부터 만들어진 mRNA가 잘 splicing되었을 때만 정상적인 기능을 하는 HIS4 단백질이 만들어질 수 있도록 해줌. 즉, 정리하자면 histidinol이 포함된 배지에서 mutant yeast를 배양할 시 제대로 splicing이 잘 일어난 yeast만 살아남게 됨. (위 그림 오른쪽의 실험 결과에서 +(positive control)와 -(negative control)를 보면 이 경향이 그대로 나타남을 확인 가능함)
한편 위 그림 왼쪽 (b)와 같이 branchpoint sequence 중 특히 U2 snRNA와 상보적인 sequence 중 256번 sequence를 A로(왼), 257번 sequence를 A로(오) 각각 바꿔봄. 먼저 이렇게 바뀐 intron 서열과 함께 wild-type U2 snRNA를 넣어줬을 때의 실험 결과를 살펴보자.
보면 위 그림 오른쪽의 (a), (b)에 각각 WT으로 나타나 있는 부분을 봤을 때 거의 splicing이 제대로 일어나지 않았음을 확인할 수 있음. (A257의 경우 splicing이 90%정도 억제되었고, A256의 경우 splicing이 50%정도 억제됨)
다음으로 앞서와 비슷하게 바뀐 intron과 상보적으로 결합할 수 있게끔 U2 snRNA를 변형시킨 채로 넣어봄. 그랬더니 위 그림 오른쪽 (a), (b)에서 각각 U36, U37로 표시된 부분을 보면 알 수 있듯이 WT U2 snRNA를 사용했을 때보다 splicing이 잘 일어남.
이를 통해 U2 snRNA와 branchpoint의 conserved sequence간의 base-pairing이 splicing에 있어 essential하다는 것을 알 수 있음.
한편 별도의 추가실험을 통해 U2는 U6와도 base-pairing을 이룰 수 있다는 것을 알게 됨. (단순히 sequence를 비교하는 작업을 통해 이를 쉽게 알아낼 수 있음) 일반적으로 snRNP가 splicing을 시작할 수 있도록 도와줄 때의 U2, U6의 base-pairing 부분을 helix I이라 부르고, 이 밖에 U2의 5' end 부분과 U6의 3' end와 상호작용해서 mammalian cell(not yeast cell)에서 splicing이 잘 일어나도록 해줄 때 이 부분을 helix II라고 부름. 이 사실은 참고 수준으로만 알아둘 것.
U5 snRNP
U5 snRNA의 sequence와 intron, exon 부분의 sequence를 대조해본 결과 이렇다할 complementarity가 관찰되지 않음. 그럼에도 아래와 같은 실험을 통해 U5가 splice site 주변에서 interaction을 한다는 것은 확인함.
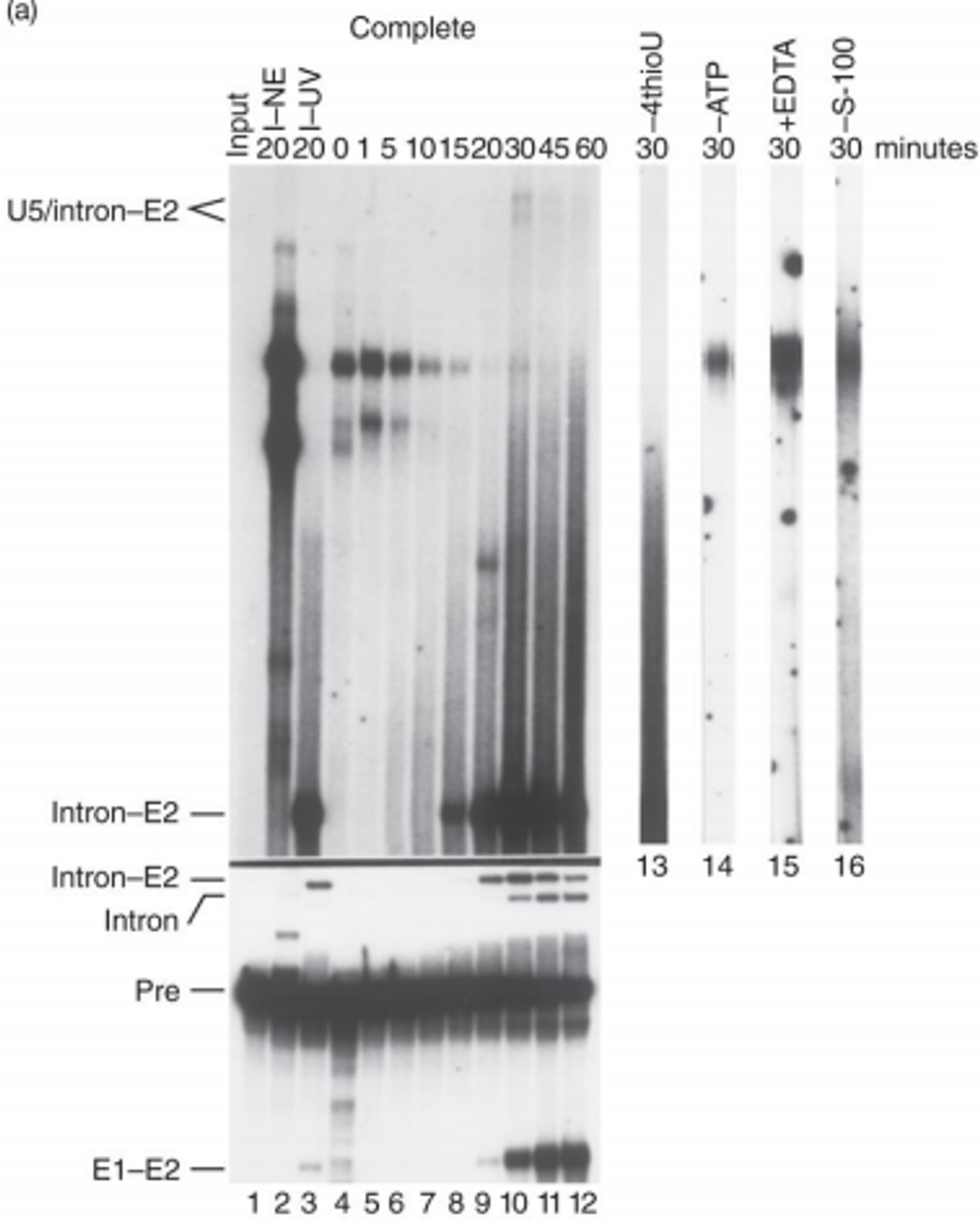
이 실험에서는 second exon의 첫 번째 자리에 4-thioU를 넣어줌. 이 때 thioU는 UV를 비춰줄 시 주변부에 위치한 녀석들을 다 crosslinking시켜주는 효과를 가지고 있음.
실험 결과를 살펴보자. 우선 x축은 시간으로, 점점 시간이 경과함에 따라 위 그림 아래쪽에 Pre로 나타난 부분을 봤을 때 splicing이 진행되어가는 것을 알 수 있음.
시간이 30분 경과했을 때를 보면 thioU에 의해 crosslinking되어 나타난 U5/intron-E(exon)2 band가 연하게나마 관찰됨. 이를 통해 U5 snRNP가 splicing에 관여한다는 것을 알 수 있음. (나머지 실험들은 일종의 control 실험이므로 참고만 할 것)
한편 30분 경과 시 나타난 이 complex에 구체적으로 U5/intron-E2가 들어있다는 사실은 어떻게 알아냈을까. 이를 확인하기 위해 아래와 같은 실험을 수행함.
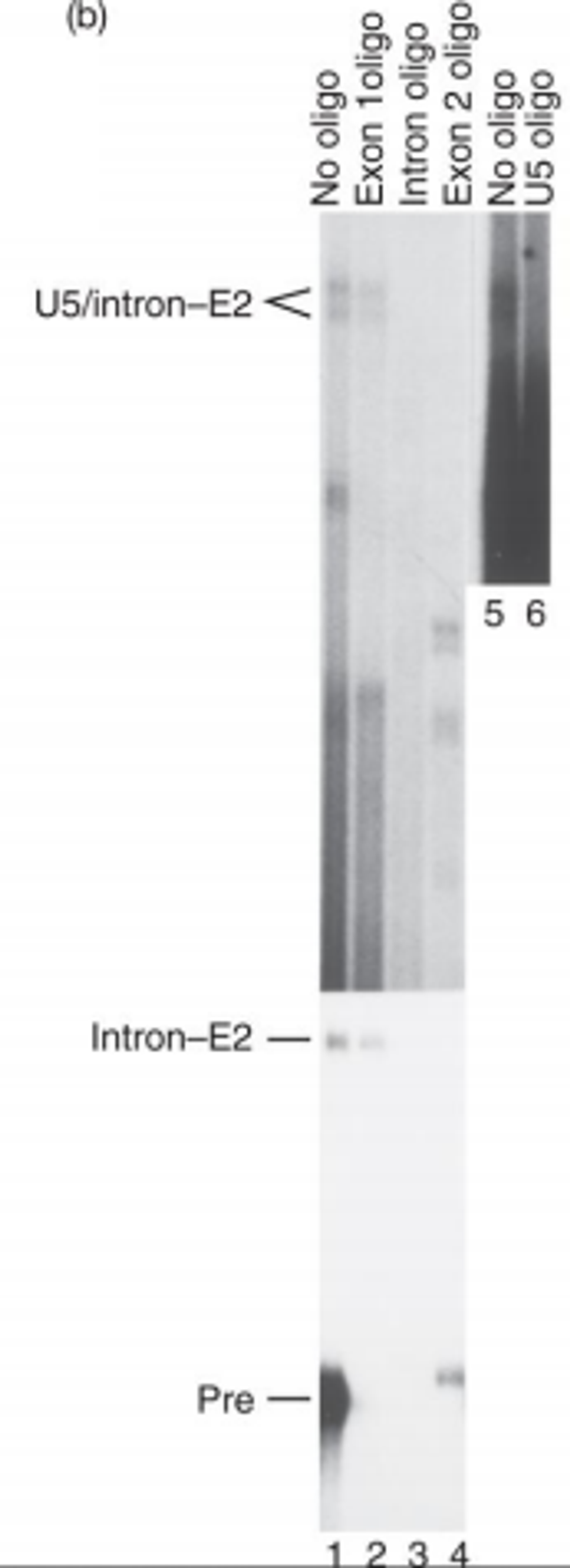
이 때 수행한 실험은 RNase H assay임. RNase H는 double strand를 형성한 hybrid에서 RNA를 분해하는 활성을 가지고 있음.
기본적인 실험 아이디는, 앞서의 실험에서 분리된 complex에 exon 1 oligo, intron oligo, exon 2 oligo, U5 oligo를 넣어보면서 이 oligo들이 complex에 붙는지를 확인하는 것임. 이 때 당연히 oligo가 complex에 붙으면 그 부분이 RNase H에 의해 분해될 것이므로 band가 나타나지 않을 것임.
실험 결과를 보면 complex에 exon 1 oligo를 넣어줬을 경우 band가 거의 그대로 관찰된 반면, intron oligo, exon 2 oligo를 넣었을 때는 band가 사라짐. 이를 통해 complex에 exon 2와 intron이 포함되어 있음을 알 수 있음. 이와 함께 lane 5, 6을 비교해보면 아무 oligo를 넣지 않았을 때(5)에 비해 U5 oligo를 넣었을 때(6) complex에 해당하는 위치에서의 band가 연해짐을 관측 가능함.
결과적으로 앞서의 실험에서 관찰된 complex에 U5, intron, exon 2가 있음을 확인할 수 있었음. (다시 말해 U5가 intron, exon 2와 interaction한다는 것을 확인함)
한편 추가적인 실험을 통해 U5 snRNA가 첫 번째 exon의 마지막 nucleotide, 두 번째 exon의 첫 번째 nucleotide와 상호작용을 해 아래와 같은 구조를 만들어낸다는 것을 알아냄.
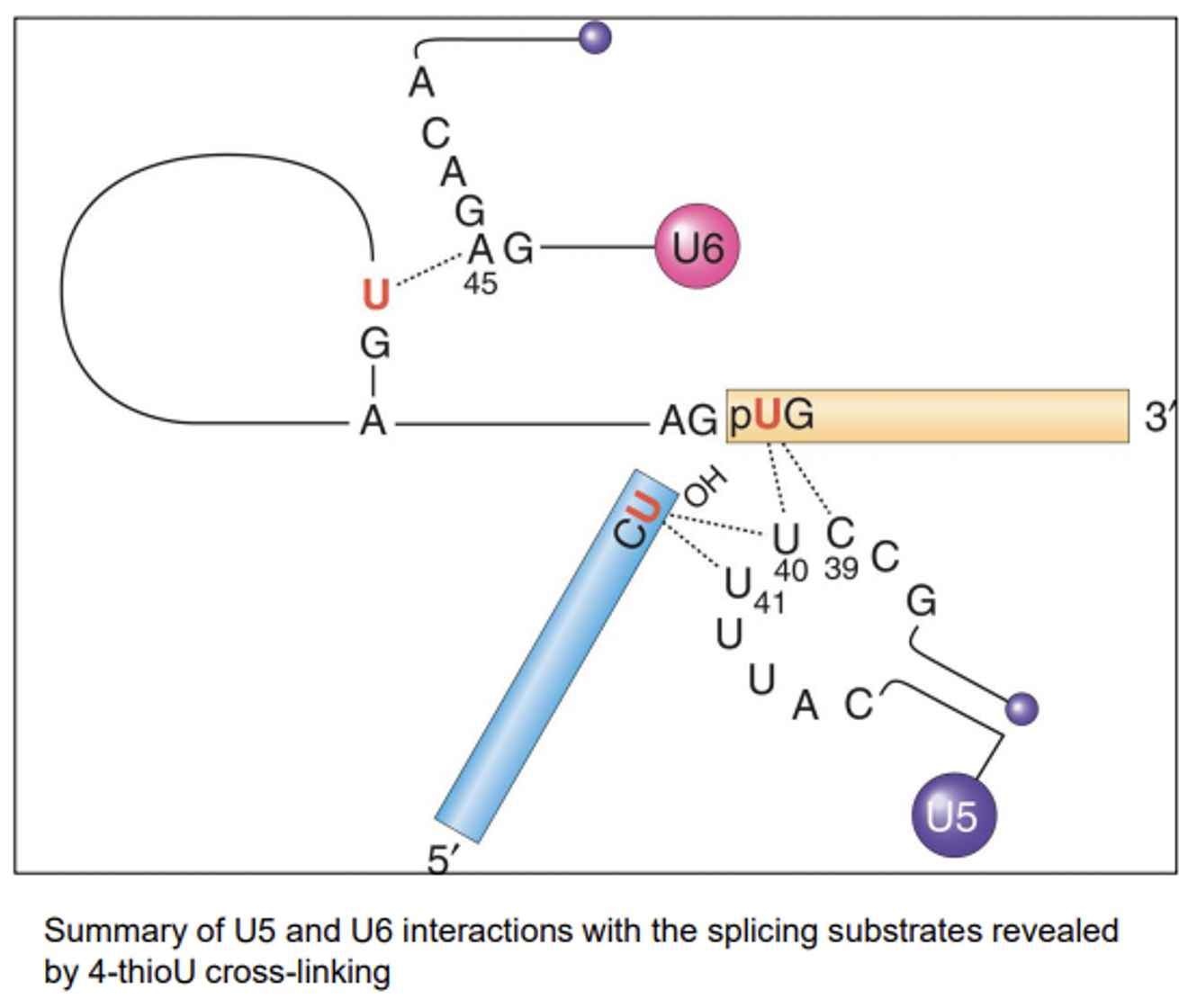
결국 U5 snRNP는 위 그림과 같이 splicing을 위해 두 exon을 잡아주는 역할을 수행함. (위 그림에 앞서 언급했던 U6도 나타나 있으므로 참고할 것)
U4 snRNP
U4는 snRNP 중 가장 characterize가 잘 안되어있는 녀석임.
U4는 U6와 base-pair를 형성 가능함. 그러나 구체적으로 U4가 splicing에서 어떤 역할을 하는지는 아직 불분명함.
한 가지 흥미로운 사실이 있는데, U6가 splicing 과정에 참여하는 시점에서 U4는 이미 제거된 상태라는 것임. 이와 다른 사실들을 종합해봤을 때, 아마도 처음 U4가 들어올 때부터 U4와 U6가 base-pairing을 하고 있는데, 이 때 U6는 U4와의 base-pairing에 의해 inactivation되어 있고, U4가 (정확히 뭔지는 모르지만) 제 할일을 마치고 빠져나가면 그제서야 U6가 activation되어서 (U1이 있었던 자리를 차지하고)역할을 수행하는 것이 아닐까 생각하고 있음.
다음 포스트에서는 snRNA가 splicing에 있어서 얼마나 핵심적인 역할을 수행할 수 있는지에 대해 알아보자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 14.4 : spliceosome cycle (0) | 2023.12.19 |
|---|---|
| [분자생물학] 14.3 : splicing에서 snRNA의 역할 (0) | 2023.12.19 |
| [분자생물학] 14.2 : splicing의 분자적 작동원리 - 1 (0) | 2023.12.18 |
| [분자생물학] 14.1 : 진핵생물에서의 splicing (0) | 2023.12.18 |
| [분자생물학] 13.8 : heterochromatin, chromatin methylation (0) | 2023.12.18 |