이번 포스트부터는 splicing의 작동원리에 대해 알아보자.
splicing이 일어나는 mechanism을 정리해보면 아래와 같음.

하나하나 살펴보자. 우선 앞서 살펴본 branchpoint인 A(그 중에서도 2번탄소)에 달려있는 OH기가 5' intron과 exon 사이에 있는 phosphodiester bond를 공격함. 그렇게 되면 위 그림에서도 나타나 있듯이 고리형으로 lariat shape이 형성되게 됨. (GU부분이 A에 붙어버림)
한편 직전의 과정에 의해 파란색 exon 끝부분에 OH가 생기게 되고, 이 OH가 3' 쪽에 위치한 노란색 exon의 phosphodiester bond를 공격하게 됨. 그 결과 exon끼리 연결되고, 중간의 intron 부분은 lariat shape 그대로 잘려나가게 됨.
그렇다면 이런 mechanism에 대해서는 어떻게 알게 되었을까.
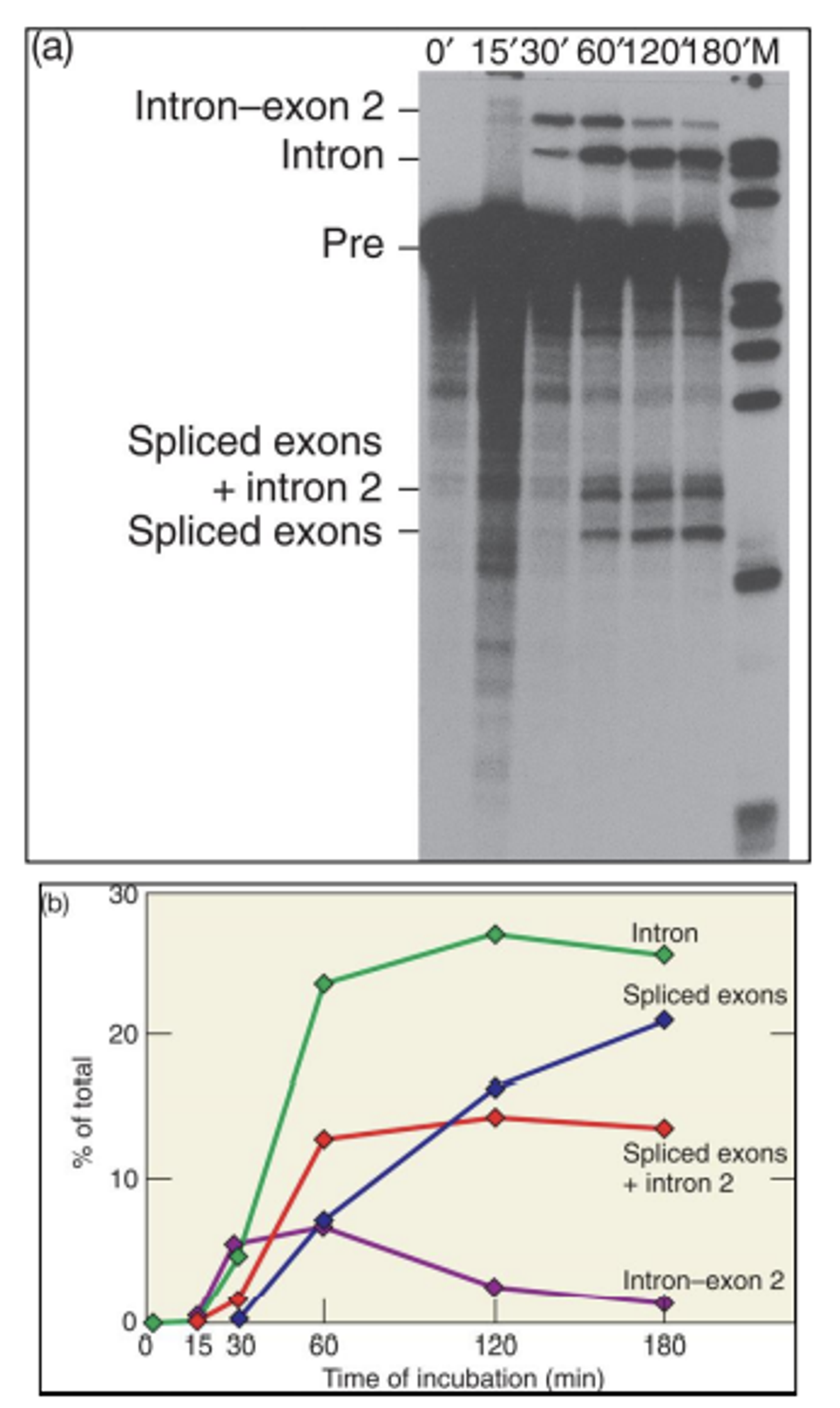
위와 같은 실험 결과를 통해 알게 됨. 우선 위 그림 위쪽에 나와있는 gel 전기영동 결과에서 pre는 아직 splicing되지 않은 RNA의 band임. 그런데 splicing을 시켜주고 나면 pre보다 더 위쪽에서 band들이 관찰됨. (즉, 뭔가 더 무거워진것)
그런데 흥미롭게도 본 실험에서 사용한 gel인 polyacrylamide(주로 RNA, 단백질 분리시에는 polyactylamide gel을 많이 사용하는데, 이 녀석은 agarose gel에 비해 구멍이 작기 때문에 더 촘촘하게 입자들을 통과시킬 수 있음)의 농도를 10%로 했을 때는 위 그림에서와 같이 초반에는 pre 위쪽에 신호가 나타나는 반면 농도를 4%로 했을 때는 pre보다 아래쪽에 band가 나타남. 이런 경향을 보이는 가장 대표적인 구조가 circular form 혹은 branched form임. 따라서 이를 통해 splicing 중간에 생기는 lariat 구조의 존재여부에 대해 알게 됨.
한편 실제로 나중에 각각의 band들을 assign해서 봤더니 위 그림 위쪽 좌측에 나타난 것처럼 각 band가 identify됨. 보면 처음에는 intron과 2번째 exon(뒤쪽에 있는 exon)간의 결합체가 많이 나타나다가 시간이 지날수록 이 녀석의 농도는 감소하고, 그 대신 intron 혹은 spliced exon의 농도가 증가함. 이를 통해 처음에는 exon 1이 나머지 녀석들과 분리되고 intron과 exon 2가 붙어있는 중간체가 생성되었다가, 이후 exon 1과 exon 2가 다시금 합쳐지고 intron은 분리되는 mechanism으로 splicing이 진행된다는 것을 알게 됨.
앞서 nuclear intron의 5', 3' 말단 근방의 서열이 보존되어있다고 말했었는데, 이와 유사하게 branchpoint 근방의 서열도 일종의 consensus sequence임. yeast의 경우 UACUAAC의 서열이 매우 보존되어 있음. (이 중 마지막 A가 branchpoint임) 한편 고등진핵생물의 경우 yeast보다는 consensus sequence가 더 variable하기는 하지만, 그럼에도 꽤나 잘 보존되어 있다는 사실은 변하지 않음.
branchpoint 근방의 consensus sequence가 splicing에 있어 어떤 역할을 하는지를 알아보기 위해 아래와 같은 mutant study를 수행함.

우선 맨 위는 WT이며 정상적으로 exon과 exon사이가 잘 붙음.
mutant #1의 경우 아예 consensus sequence를 없앤 mutant인데, 이 경우에는 splicing이 일어나지 않음을 확인할 수 있었음.
mutant #2의 경우 consensus sequence는 그대로 놔뒀지만 그 뒤에 새로운 서열들을 첨가해줌. 그랬더니 consensus sequence로부터 일정 거리만큼 떨어진 새로운 서열 안에 있던 AG 부분이 어떤 식으로던 인식이 되어서 그 부분까지만 intron으로 인식되어 잘려나간다는 것을 확인할 수 있었음.
mutant #3의 경우 consensus sequence는 그대로 있으나 이 consensus sequence의 위치를 노란색 exon 사이로 옮김. 그랬더니 노란색 exon 중 consensus sequence로부터 일정 거리만큼 뒤쪽에 있는 AG 서열 부근까지가 잘려나가는 식으로 splicing이 진행됨.
이를 통해 어떤 식으로든 consensus sequence가 downstream의 AG 서열을 인지하는 데 중요한 역할을 할 것이라는 것을 알 수 있음. (특히 mutant #2와 #3의 결과로부터 이를 추측할 수 있음)
이후 사람들이 splicing을 일으키는 구체적인 녀석은 무엇인지에 대해 관심을 가지기 시작했고, spliceosome이 그 역할을 한다는 것을 알게 됨. (이 때 spliceosome은 단백질과 RNA로 이루어져 있음)

처음 spliceosome의 존재를 확인한 실험 결과는 위 그림과 같음. 이 실험은 pre-mRNA에 nuclear extract를 전부 다 통과시켜본 후, ultracentrifuge를 통해 분리해서 혹시라도 pre-mRNA와 결합하는 일부 nuclear extract가 있는지를 확인해본 것임.
보면 wild-type pre-mRNA를 써준 경우 10정도의 fraction number에서 약 40S의 침강계수를 가지는 단백질 때문에 생긴 peak이 관찰됨. 즉, pre-mRNA와 붙는 단백질이 있다는 것을 이 실험을 통해 확인함. 한편 5' splice site에 mutation을 유발한 경우(파란색) 앞서 나타나던 40S 근방에 대한 extract signal이 유의미하게 약해진다는 것을 확인함. 이를 통해 5' 부근에 결합하는 어떤 녀석이 존재할 것임을 유추할 수 있음.
spliceosome을 구성하는 여러가지 요소들을 snRNP(small nuclear ribonuclear protein, 읽을 때는 snurp로 발음함)라고 부름. (protein + RNA) 이 녀석에는 RNA성분인 snRNA(small nuclear RNA)도 포함되어 있음. snRNA에는 U1, U2, U4, U5, U6가 있으며, 이들 component들이 모두 splicing에 매우 핵심적으로 작용함.
다음 포스트에서는 snRNP 하나하나의 기능과 역할에 대해서 자세히 살펴보도록 하자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 14.3 : splicing에서 snRNA의 역할 (0) | 2023.12.19 |
|---|---|
| [분자생물학] 14.2 : splicing의 분자적 작동원리 - 2 (0) | 2023.12.18 |
| [분자생물학] 14.1 : 진핵생물에서의 splicing (0) | 2023.12.18 |
| [분자생물학] 13.8 : heterochromatin, chromatin methylation (0) | 2023.12.18 |
| [분자생물학] 13.7 : histone code (0) | 2023.12.18 |