이번 포스트부터는 splice site selection에 대해 알아보자.
snRNP만 있어서는 exon-intron boundary에 충분히 강하게, 그리고 특이적으로 결합하지 못함. 따라서 snRNP의 binding을 도와주는 additional한 splicing factor들이 필요함.
우선 이와 같은 논의를 하기 위해서는 spliceosome의 작용 방식과 관련되어 있는 exon definition과 intron definition이 무엇인지에 대해 알아야 함.

하나하나 살펴보자. exon definition의 경우 쉽게 말하면 exon을 인지해서 intron을 잘라내는 mechanism을 의미함. 그렇기에 위 그림 (a)와 같이 특정 exon-intron boundary site를 변형시키게 되면 그 결과 노란색 부위도 intron으로 인지되어 제거됨.
intron definition의 경우 쉽게 말하면 intron을 인지해서 intron을 잘라내는 mechanism을 의미함. 그렇기에 위 그림 (b)와 같이 특정 exon-intron boundary site를 변형시키게 되면 그 결과 노란색 오른쪽에 있는 intron 부위도 exon으로 인지되어 남아있게 됨.
마지막으로 (c)의 경우에는 exon definition이기는 하나 특정 splice site를 변형시켜줄 시 그 site 대신 다른, cryptic splice site를 기점으로 해서 splicing을 해버리게 됨.
organism에 따라 exon definition인지, intron definition인지 여부가 다 다르며, 일반적으로 higher eukaryote, 혹은 척추동물의 경우 exon definition meahanism을 주로 사용하고, fission yeast, budding yeast 등은 intron definition mechasism을 주로 사용함. (참고로 이 때 exon definition이 제대로 되려면 exon의 길이가 짧아야 할 것임. 이 때문에 exon의 상대적인 길이가 yeast에 비해 많이 짧고 intron이 긴 higher eukaryote, 척추동물 등이 exon definition mechanism을 잘 사용할 수 있는 것)
다음으로 이전 포스트에서 잠시 등장했던 commitment에 대해 조금 더 알아보자.
commitment를 일으키는 단백질 중 SC35라는 녀석이 가장 먼저 관측됨. 이와 관련된 실험 결과를 살펴보자.

lane 1번의 경우 pre mRNA와 nuclear extract만 넣어줌. 그랬더니 mature mRNA의 band가 잘 나타남. 한편 lane 4의 경우 pre mRNA와 nuclear extract에 더해 5' splice site를 가지고 있는 competitor를 같이 넣어줌. (대략 원래 pre mRNA보다 40배 많이 넣어줌)
그러면 결국 이 competitor가 spliceosome을 다 끌어모아버려서 결과적으로 mature mRNA band가 나타나지 않게 됨. 한편 lane 6, 7, 8의 경우 competitor를 넣어주기 전에 먼저 SC35와 pre mRNA, nuclear extract를 넣어준 뒤 incubation을 시켜줬음. 그럴 시 이후 competitor를 넣어줬음에도 불구하고 mature mRNA band가 나타남.
즉, SC35가 pre mRNA와 spliceosome이 한 번 붙으면 다시 떨어지지 않게 해줘서(commitment complex를 형성하게 해줘서) 특정 mRNA 부근에 끝까지 splicing이 일어나게 된다는 것을 알 수 있음.
한편 다른 여러 실험들에 의해 실제로 SC35가 U1보다 먼저 들어가게 되고, 이후 U1도 들어가서 결과적으로 commitment complex가 형성된다는 것이 밝혀짐. 참고로 SC35는 SR protein의 한 종류인데, SR protein은 RNA-binding protein이며 serine, arginine이 많은 domain을 가지고 있는 녀석들임. 이 SR protein에는 SC35뿐 아니라 SF2/ASF도 포함되며, SF2/ASF도 commitment complex의 형성에 관여함.
다음으로 yeast에서 bridging protein에 의해 commitment가 유발되는 경우를 살펴보자. 이 때 bridging protein은 일반적으로 앞서 살펴본 exon definition 혹은 intron definition을 위해 exon 혹은 intron 부위를 잡아주는 역할을 함.
yeast에서는 이 역할을 수행하는 녀석이 branchpoint bridging protein(BBP)임. (이 녀석의 이름이 이렇게 지어진 이유는 BBP가 branchpoint sequence에 dependent한 단백질적인 특성을 가지고 있기 때문임. 다만 BBP가 branchpoint와 직접 붙는 것은 아님)
한편 이 밖에 intron의 5' end에 붙는 U1 snRNP, intron의 3' end에 붙는 Mud2p, U1과 BBP 사이를 연결해주는 yPrp40p도 존재함.
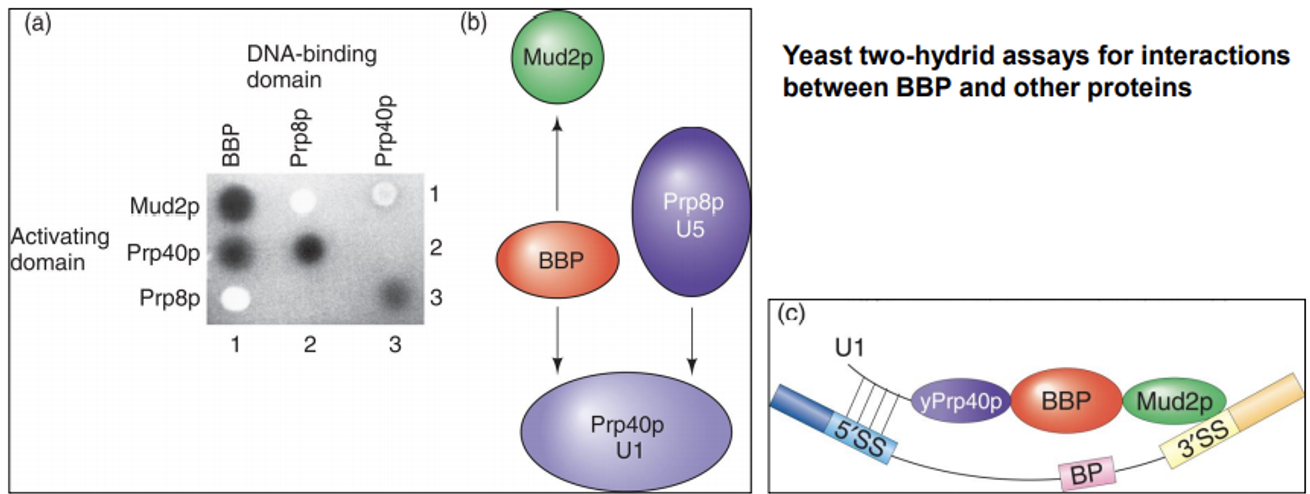
위 그림 왼쪽 (a)를 보면 Prp40p, Mud2p, Prp8p, BBP 간의 상호결합여부를 판단하기 위해 수행한 yeast two-hybrid assay의 결과가 나타나 있음. 이를 통해 (b)와 같이 단백질들이 어떤 식으로 상호작용하는지를 알 수 있고, 결과적으로 이들 단백질들이 연결되어 (c)와 같은 bridge를 형성한다는 것을 알 수 있음. 이 bridge에 의해 intron이 define되고, 그 결과 define된 intron 부분이 제거될 수 있음.
(참고로 mammalian BBP는 SF1으로, 이 녀석도 동일한 bridging function을 수행할 수 있음)
한편 splicing factor 중에 Slu7이라는 녀석도 있는데, 이 녀석은 3'-splice site를 selection하는 데에 있어 매우 중요하게 작용함. (이 밖에 U2AF라는 녀석도 3'-splice site를 recognition하는 데 중요함)
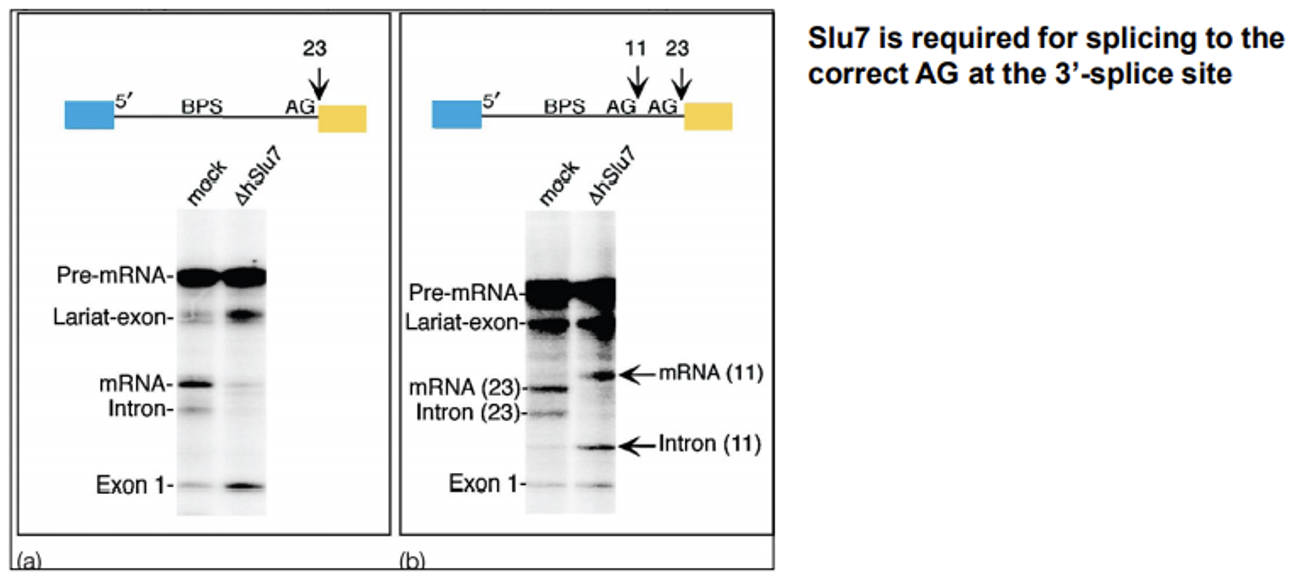
이와 관련된 실험결과에 대해 살펴보자. 우선 (a)부터 보면, mock(like WT)의 경우 mature mRNA band가 관찰되는 반면 Slu7을 antibody를 이용해 immunodepletion시킨 ΔhSlu7의 경우에는 mature mRNA band가 관찰되지 않음.
한편 (b)에서는 원래 3' splice site인 23번 부위보다 더 앞, 11번 위치에 3' splice site가 첨가되어 있음. 이 경우 mock에서는 23에 해당하는 정상적인 mature mRNA band가 나왔지만 ΔhSlu7에서는 원래라면 나오지 말아야 할 11에 해당하는 mature mRNA band가 관찰됨. 결론적으로 Slu7이라는 녀석이 3'-splicing site를 제대로 selection하는 데 매우 중요한 역할을 한다는 것을 알 수 있음.
다음 포스트에서는 RNA polymerase II가 splicing 과정과 어떤 관련이 있는지에 대해 알아보자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 14.7 : alternative splicing (대체 스플라이싱), silencing of splicing (0) | 2023.12.19 |
|---|---|
| [분자생물학] 14.6 : RNA polymerase II에 의한 splicing 조절 (0) | 2023.12.19 |
| [분자생물학] 14.4 : spliceosome cycle (0) | 2023.12.19 |
| [분자생물학] 14.3 : splicing에서 snRNA의 역할 (0) | 2023.12.19 |
| [분자생물학] 14.2 : splicing의 분자적 작동원리 - 2 (0) | 2023.12.18 |