이번 포스트부터는 DNA 복제의 개시과정에 대해 알아보도록 하자.
replication이 시작되기 위해서는 일단 melting, unzipping 등에 의해 bubble이 생겨야 함.

원핵생물, 진핵생물 각각에서 bubble이 생기는 과정을 묘사한 그림이 위에 나타나 있음.
어느 정도 비슷한 부분도 있지만, 일단 원핵생물의 경우 ATP가 소모되는 DNA melting이 먼저 일어난 후 뒤이어서 helicase가 loading되고 unzipping이 일어나는 반면 진핵생물의 경우 ATP가 소모되는 helicase loading이 먼저 일어난 후에 DNA melting이 뒤이어 일어난다는 것이 다름.
bacteria에서의 origin of DNA replication

bacteria의 경우 TTATCCACA(AT-rich)의 보존된 서열이 반복되어있는 oriC라는 부분이 존재함. 이 서열에 일단 DnaA와 ATP가 첨가되게 되고, DnaA는 9-mer에 결합하게 됨. 한편 DnaA는 ATPase activity를 가지는 AAA box를 포함하고 있어서 결국 ATP 가수분해를 시킬 수 있고, 이 때의 energy로 13-mer에 해당하는 부분을 melting시켜서 벌려주게 됨. (정확히는 에너지를 써서 DNA를 bending시켜주게 되면 그 결과로 melting이 일어나게 됨) 한편 DnaA와 함께 HU라는 녀석도 들어가는데, HU는 DNA의 bending을 유발하는 factor임. 참고로 이 때 ssDNA가 형성되므로 SSBP도 당연히 recruit될 것이고, melting 과정에서 supercoiling이 벌써 유발될 것이므로 DNA gyrase도 recruit될 것임.
이런 과정에 의해서 open complex가 형성되고 나면 이제 뒤이어 DnaB(helicase), DnaC가 들어옴. 이때 DnaC와 ATP의 도움을 받아 DnaB가 DNA에 loading됨. (참고로 E. coli의 경우에는 DnaB와 primase가 같이 loading되므로 이 단계에서 primer의 합성도 일어남)
yeast에서의 origin of DNA replication
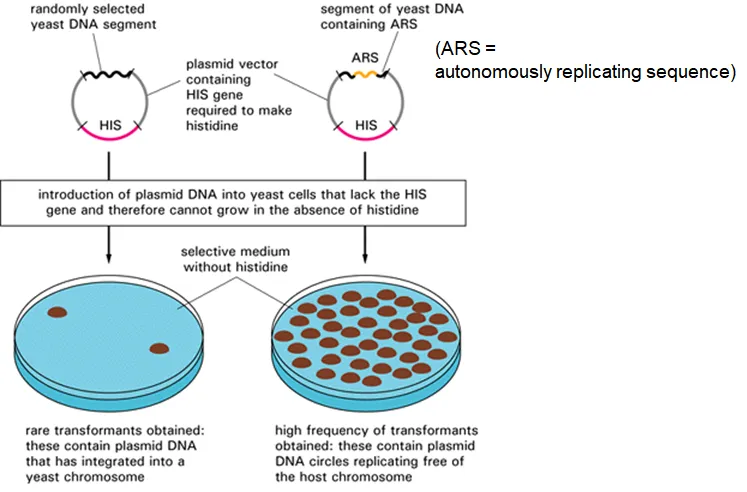
yeast에서는 ARS(autonomously replicating sequence)라는 부위가 DNA replication의 origin으로 작용함. 이를 확인한 실험이 위에 나타나 있음.
실험의 기본적인 concept은 단순함. 일단 yeast가 가지고 있는 genome을 조각조각낸 다음 HIS(histidine을 합성할 수 있도록 해주는 gene)가 포함된 vector에 각각의 조각을 random하게 삽입함. 참고로 이 plasmid는 E. coli에 접종될텐데, 이 때 사용하는 E. coli는 histidine 합성에 있어 deficiency가 있는 종임. 따라서 plasmid가 제대로 들어가서 HIS를 함유하고 있어야 비로소 de novo synthesis에 의해 그 E. coli가 histidine을 합성할 수 있을 것이고, 이들의 경우 histidine을 넣어주지 않은 배지에서도 살아남을 수 있을 것임.
만약 위 그림 왼쪽과 같이 삽입된 yeast gene 조각이 ori 역할을 하지 못한다면 plasmid가 복제되지 못할 것이므로 결국 HIS의 수가 너무 적어 cell이 거의 자라지 못할 것임. (분열 X) 한편 만약 위 그림 오른쪽과 같이 삽입된 yeast gene 조각이 ori 역할을 할 수 있다면 plasmid가 복제될 것이고 그 결과 HIS의 수가 많아져 cell이 활발히 자랄 수 있을 것임.
이런 방법을 이용해서 yeast genome 중 ori 역할을 할 수 있는 ARS를 찾아냄.

참고로 이런 식의 실험은 HIS 뿐 아니라 TRP, ARG 등에 대해서도 동일한 방식으로 수행할 수 있음.
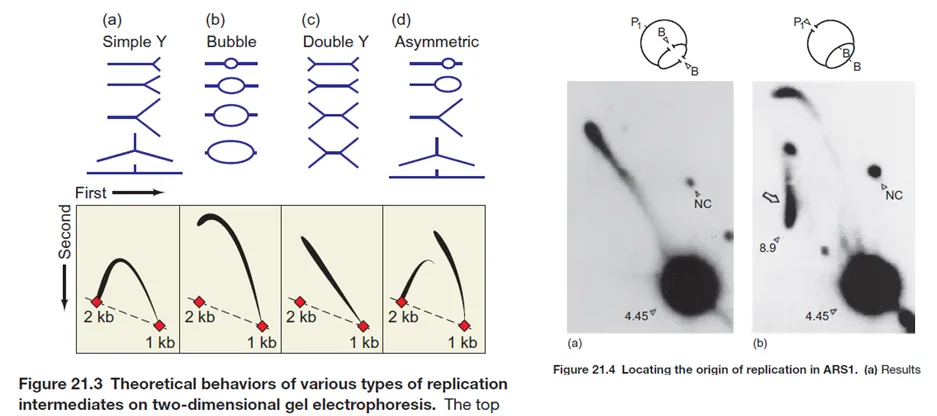
다음으로 위 실험 결과를 살펴보자. 이 경우에는 DNA의 size, size + shape을 바탕으로 DNA 2D electrophoresis를 수행하였음. 이런 실험을 수행하면 위 그림 왼쪽에 나타나는 것처럼 DNA의 모양에 따라서 서로 각기 다른 개형의 확산 양상이 나타나게 됨.
실제로 ARS1이 있는 곳 주변의 서열을 cloning을 통해 확보한 뒤 B, P를 통해 각각 절단을 해봄. 그랬더니 B로 절단했을 때는(a) double Y에 해당하는 2D 결과가 관찰되었고 P로 절단했을 때는(b) bubble에 해당하는 2D 결과가 관찰됨. 결과적으로 ARS1 주변에 bubble이 실제로 형성되어 있다는 것을 확인함.
이제 ARS의 서열은 알고 있는 상황에서 과학자들은 ARS 서열 중에서도 특별히 더 중요한 서열이 있는지에 대해 궁금증을 가지고 아래와 같은 실험을 수행함.
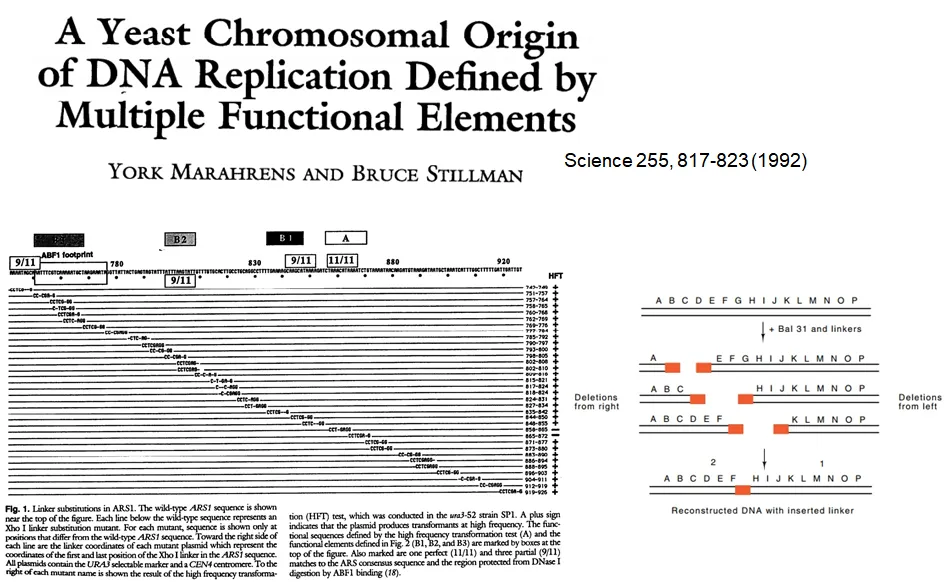
위 그림 왼쪽 위에 나타나 있는 것이 전체 ARS 서열임. 이 중 어떤 부분이 특별히 더 중요한 역할을 하는지 알아보기 위해서 각각의 부분을 site directed mutagenesis시킨 후 phenotype을 관찰해봄.
일단 site directed mutagenesis를 위해 위 그림 오른쪽에 나온 것과 같은 Bal을 사용함. Bal은 random하게 특정 DNA를 자를 수 있는 활성이 있음과 동시에 자른 부분의 일부를 exonuclease 활성을 가지고 갉아먹을 수도 있음. 따라서 제한된 양의 Bal을 넣게 되면 그 결과 ARS 중 각종 부분을 mutagenesis시켜줄 수 있음. (참고로 이 과정에서 linker도 넣어줬는데, linker를 넣어준 이유는 이후에 linker scanning analysis를 수행하기 위해서임)
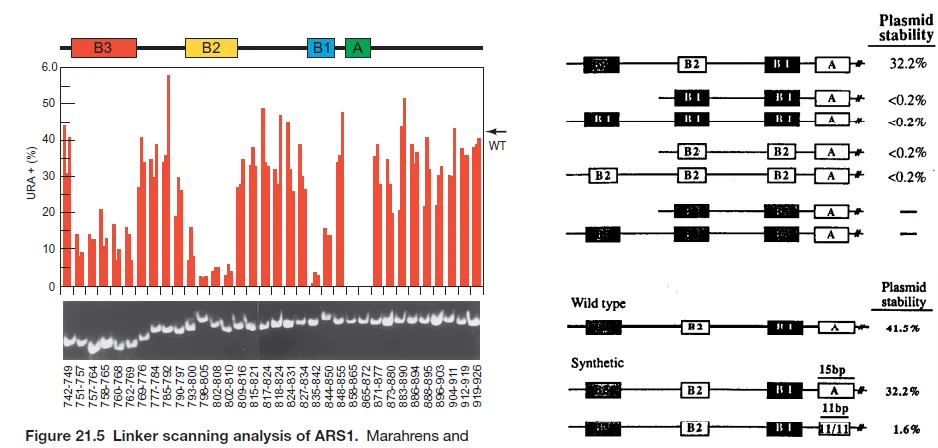
위 그림에는 실험 결과가 나타나 있음. 일단 이 경우에는 U의 생합성을 못하게끔 해준 U knockout E. coli와 함께 U를 합성가능한 gene이 들어있는 plasmid를 활용함. 그리고 각각의 plasmid에 앞서 만든 각종 변형 ARS를 넣은 채로 E. coli에 접종시킨 후 U의 합성량을 관찰해봤음. 그랬더니 다른 부분을 mutagenesis시킨 부분에서는 대부분 U 합성량이 높게 나왔지만 특이적으로 B3, B2, B1, A 부분을 바꿔줄 시 U 합성량이 확 떨어짐. 이를 통해서 yeast chromosomal replication에 있어 특히 이들 4개의 부분이 key motif로 기능한다는 것을 알 수 있음.
다음 포스트에서는 진핵생물에서의 DNA 개시 과정에 대해 알아보도록 하자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 20.2 : DNA 복제 종결 (0) | 2024.10.27 |
|---|---|
| [분자생물학] 20.1 : DNA 복제 개시 - 2 (0) | 2024.10.27 |
| [분자생물학] 19.2 : enzymology of DNA replication - 2 (1) | 2024.08.17 |
| [분자생물학] 19.2 : enzymology of DNA replication - 1 (1) | 2024.08.10 |
| [분자생물학] 19.1 : DNA replication(DNA 복제)의 특징 (1) | 2024.08.10 |