DNA replication의 general한 특성은 아래와 같음.
1. semiconservative하게 replication됨.
2. half-discontinuous함. (즉 절반의 경우는 오카자키 절편이라 불리는 짧은 가닥으로 조각난 채 합성됨)
3. primer가 필요함. (RNA polymerase와 달리 DNA polymerase는 primer가 제공하는 3' 말단이 필요함)
4. 일반적으로 bidirectional함.
이에 대해 하나하나 알아보자.
1. semiconservative DNA replication (반보존적 DNA 복제)

위 그림에 semiconservative, conservative, dispersive DNA replication model이 나타나 있음. 이 중 아래와 같은 Meselson과 Stahl의 실험 결과를 통해 semiconservative model이 맞다는 것이 확인됨.

실험 idea는 단순함. heavy media에서 cell을 배양하다가 다시금 light media에 옮겨서 cell을 배양함. 위 그림 오른쪽에는 radiography 결과가 나타나 있는데, 이때 first generation에 한 개의 band만 생긴다는 사실을 통해 conservative model이 아님을 확인할 수 있고, 이후 second generation 이후에 두 개의 band가 생긴다는 사실을 통해 dispersive model이 아님을 확인할 수 있음.
2. half-discontinuous

과거에는 replication이 어떻게 일어나는지에 대해 잘 몰랐고, 그러다 보니 위 그림과 같이 replication fork를 기점으로 양 방향의 서열이 모두 continuous하게 합성되는 경우, semidiscontinuous하게 합성되는경우, 두 방향이 모두 discontinuous하게 합성되는 경우 중 어느 것이 맞는지에 대해 잘 몰랐음. (다만 5'→3' 방향으로 replication이 일어난다는 것은 알고 있었음)

그러던 중 위 그림상에 나타난 것과 같이 Okazaki 부부가 okazaki fragment(오카자키 절편)의 존재와 관련된 연구를 수행함.
일단 과거에도 in vitro 환경에서 5'→3'으로 DNA 합성이 일어난다는 사실은 알고 있었으므로 이론적으로는 semidiscontinuous한 model이 부합해보였음. 그러나 in vivo에서도 과연 이런 일이 일어나는가에 대한 확신이 없었음.
그러던 중 매우 짧은 radioactive pulse를 이용해서 label한 후 시간대별로 추출한 DNA 추출물을 봤더니 위 그림 오른쪽의 data에서도 나타나는 것처럼 매우 짧은 길이의 DNA가 관찰됨. 즉, denaturation 이후에 unconnected short chain이 isolation된 것임.

한편 WT T4 phage와 temperature sensitive ligase-defective T4 phage를 서로 비교해봤더니 위 그림 아래에 나타난 graph와 같이 ligase-defective할 시 bacteriophage DNA의 E. coli 내에서의 합성이 더 적게 일어남. (실제로 early gene 말고는 모두 T4 phage로부터 유래된 일부 system이 gene replication에 관여함) 실제로 위 그림 오른쪽과 같이 ligase-defective할 시에 큰 size의 DNA가 더 줄어든다는 사실도 확인함. 이를 통해서 ligase가 어떤 식으로든 작은 fragment를 다시금 연결해줘서 큰 DNA로 만들어주는 역할을 수행한다는 사실을 알 수 있음.
그런데 당시에는 이런 실험 결과에 대해서 혹시 중간에 DNA 합성 과정에서 RNA가 들어간 뒤 Ung(Uracil N-glycosylase)와 같은 RNA 염기(그 중에서도 U의 염기)를 당으로부터 제거하는 효소들에 의해서 RNA 부분이 잘리고 이 때문에 이런 fragment가 생기는 것이 아니냐고 반박이 들어옴. 이 반박에 대해 검증하기 위해 아래와 같은 실험이 추가적으로 시행됨.
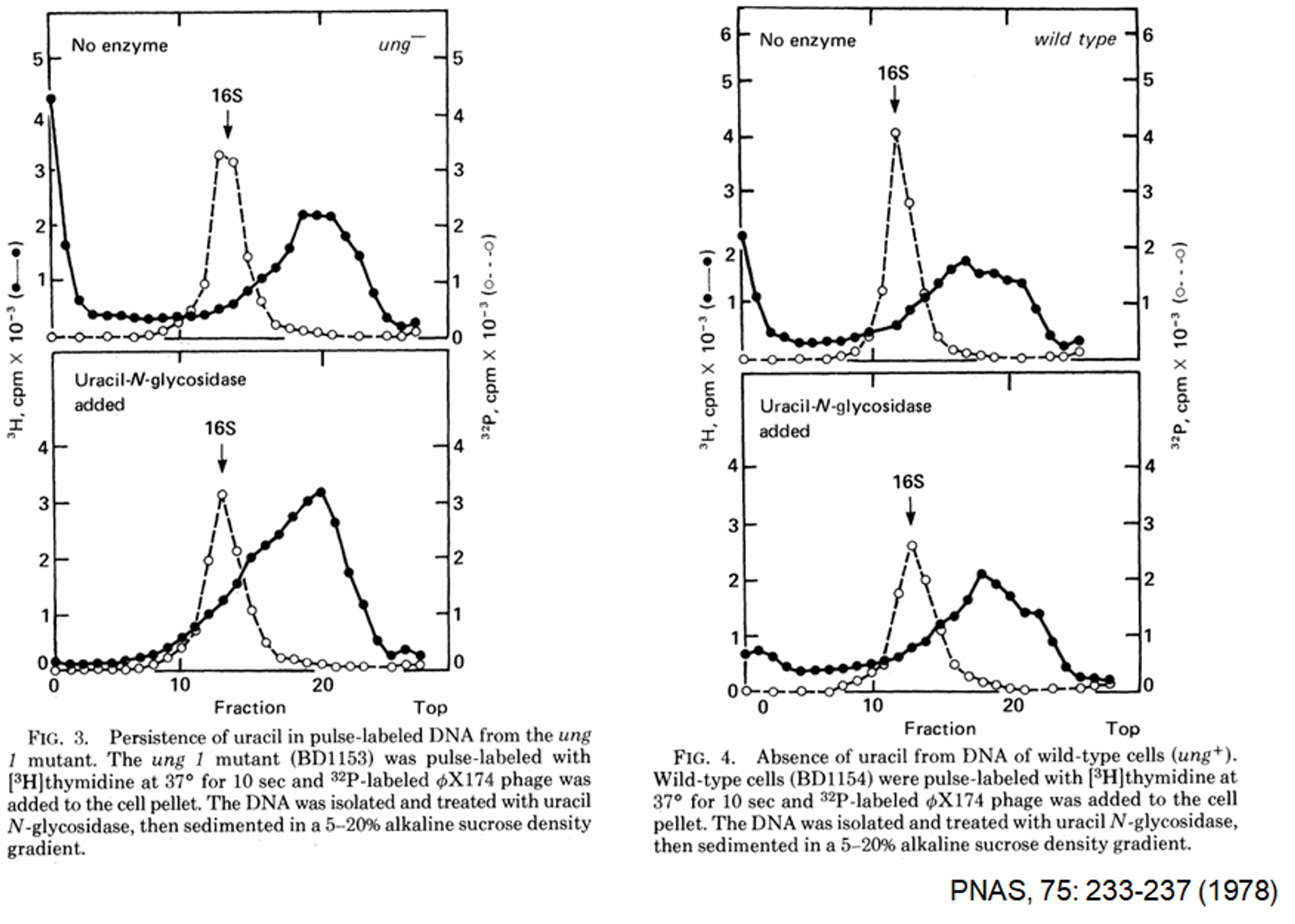
이 실험의 경우 UTP를 넣어주는 실험 setting 하에서 Ung가 있을 때(오른쪽), 없을 때(왼쪽) 각각에서 short fragment가 만들어지는 정도를 비교함. 그 결과 별 차이가 없다는 사실이 입증됨.
결과적으로 Okazaki fragment는 Ung incorporation 과정이 아니라 DNA replication 과정에서 생기는 fragment라는 것을 확인함.
3. requirement of primers
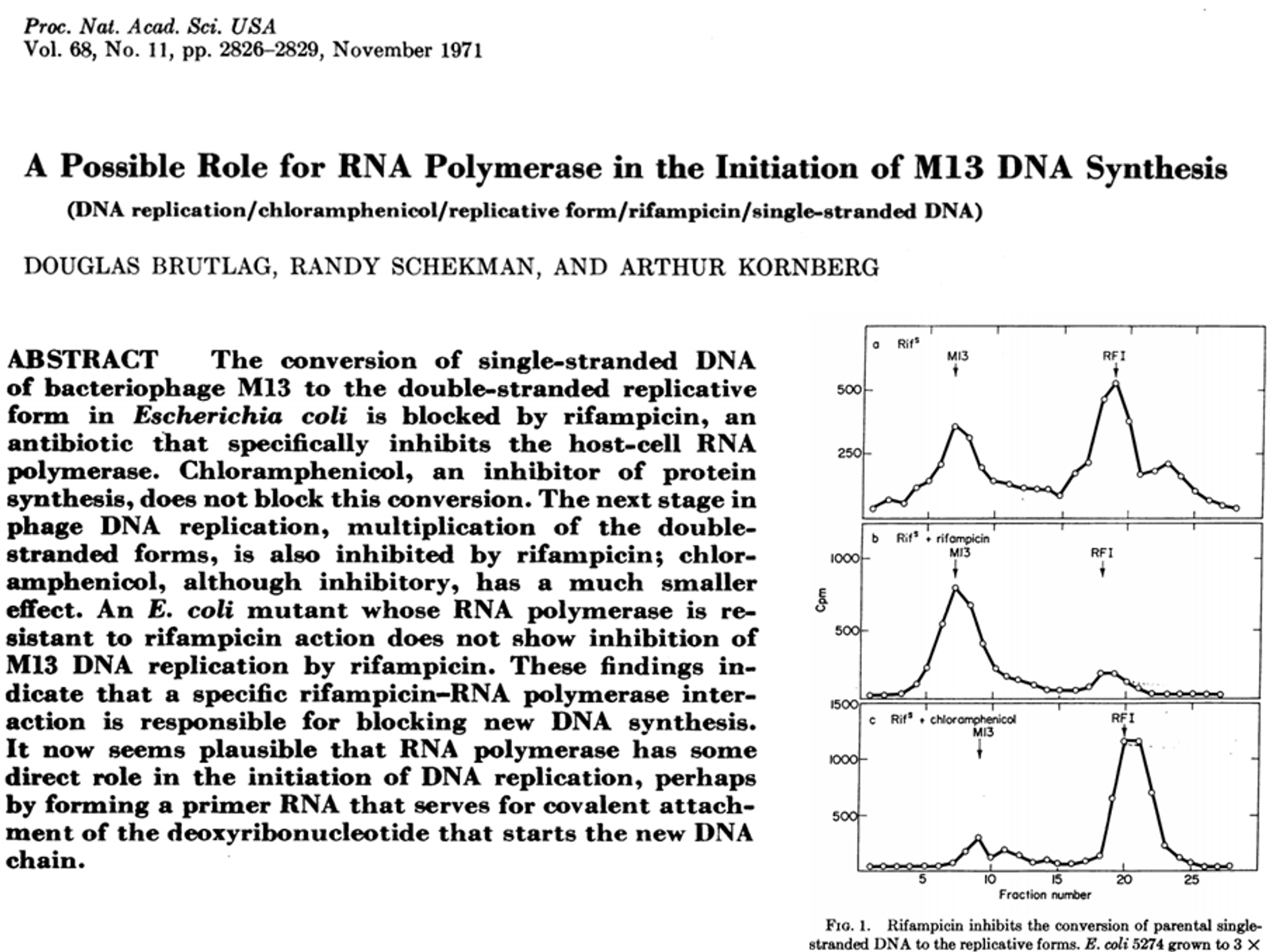
한편 위 논문에서는 primer와 관련된 실마리를 얻음.
이 실험에서는 M13 DNA synthesis를 중점적으로 살펴봤고, 이 때 rifampicin과 같이 RNA polymerase의 작용을 inhibition 하는 녀석, 혹은 chloramphenicol과 같이 peptidyl transfer를 방해하는 녀석을 넣어줬을 때 normal한 경우에 비해 M123 DNA synthesis 정도가 어떻게 변하는지를 관찰함.
그 결과 rifampicin을 넣어줬을 때는 DNA replication product가 감소하지만(RF1 peak이 감소) chloramphenicol을 넣어줬을 때는 DNA replication product가 그대로임.
이를 통해서 DNA polymerase에 의한 replication 과정에서 RNA polymerase 활성이 somehow 필요하다는 것을 알게 됨. (우리는 지금 이 것이 primase에 의한 primer 활성 때문이라는 것을 알고 있음)
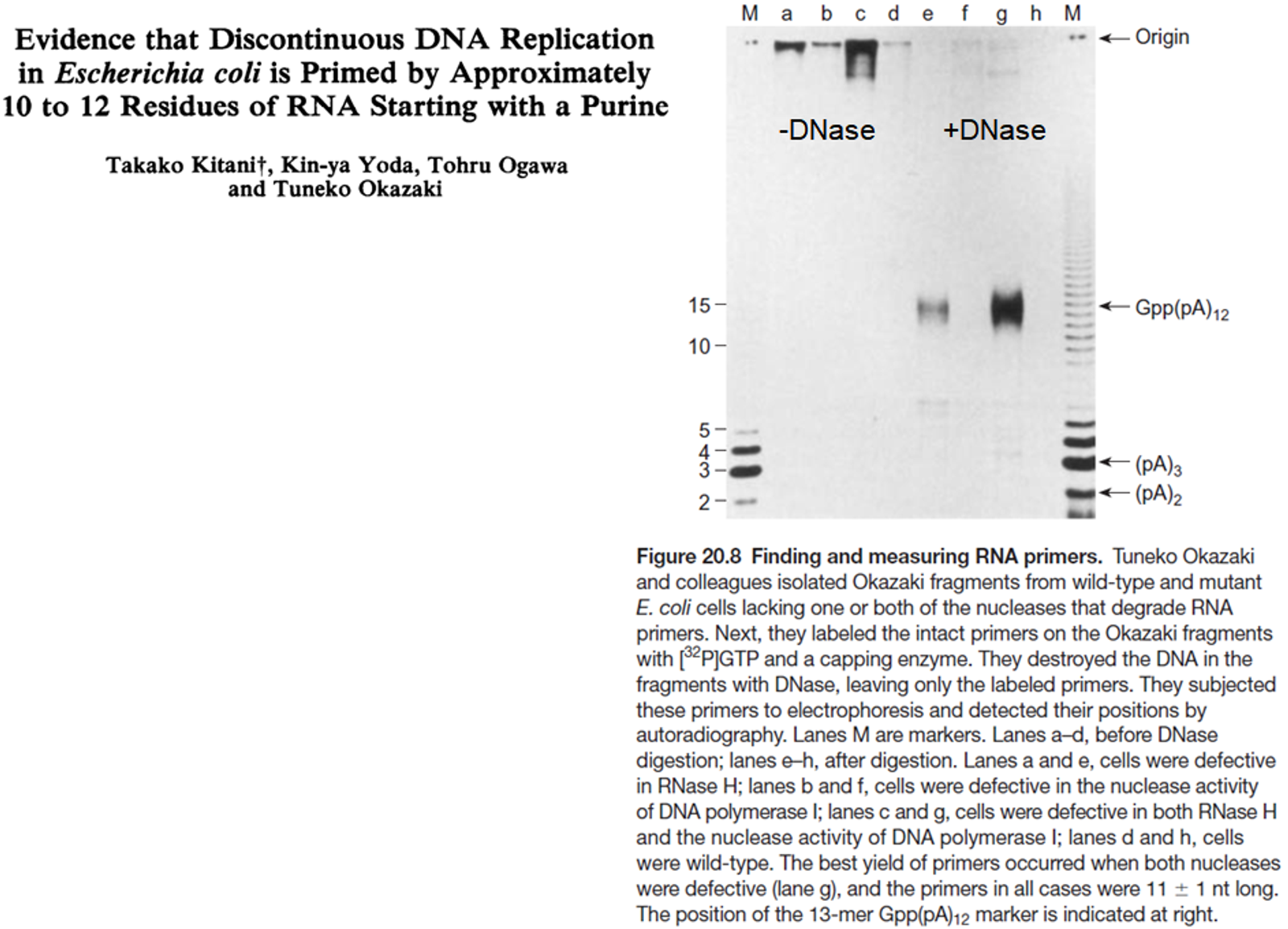
한편 위 그림에 나타나 있는 논문은 실제로 10~12bp정도의 길이를 가지며 purine기로 시작하는 RNA fragment가 DNA replication 시 RNA primer로써 존재하고 있다는 사실을 밝힌 것임.
사실 RNase H나 DNA pol I 등에 의해 RNA는 쉽게 분해될 수 있으므로 RNA primer는 발견하기 쉽지 않음. 이 때문에 본 실험에서는 RNase H(RNA-DNA hydrid에서 RNA 제거)와 DNA Pol I(exonuclease 활성으로 primer 제거)의 활성을 block시킴. 이런 상황에서 추출한 replication product와 함께 위 그림 오른쪽 위에 나타난 것과 같이 DNase를 넣지 않아보기도 하고 DNase를 넣어보기도 했을 때, DNase를 넣게 되면 DNA가 다 분해된 뒤 결과적으로 RNA primer 부분만 남아서 band로 관찰되게 됨.
4. bidirectional
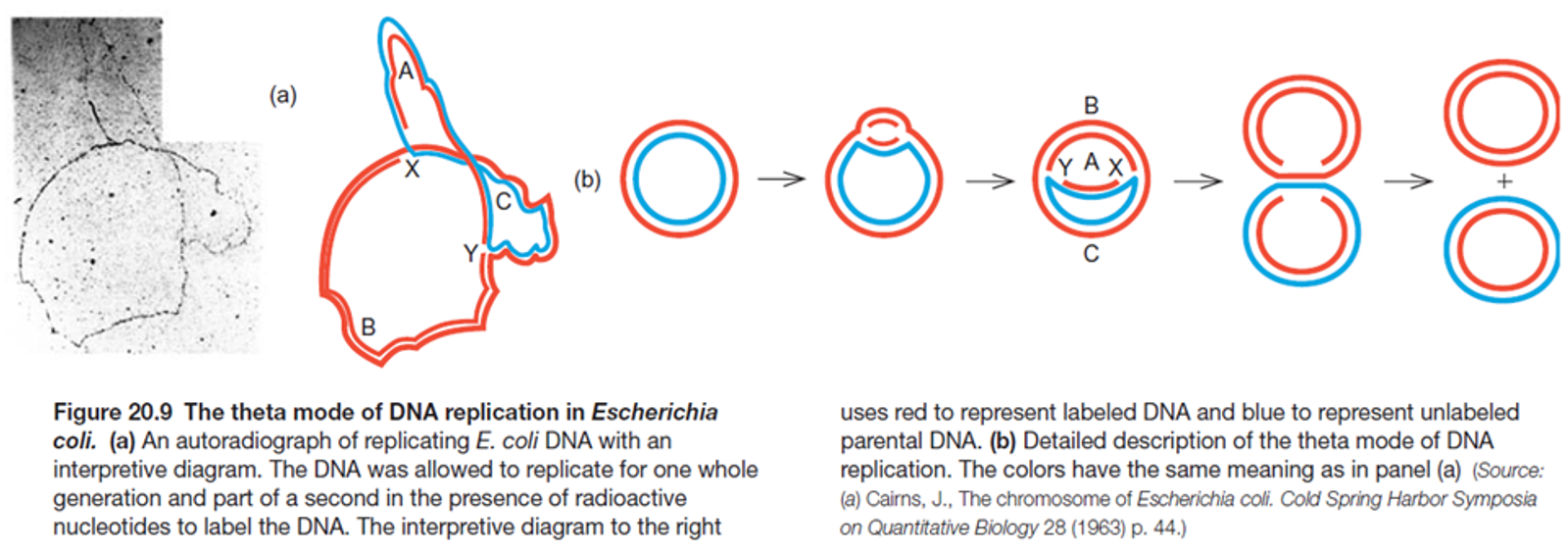
원형 DNA의 replication 과정을 관찰한 결과가 위 그림 왼쪽과 같음. 이 때 θ 모양이 관찰됨. 이 모양이 어떻게 만들어질 수 있을지를 생각해보면, 결국 위 그림상에 전반적으로 나타나 있는 것처럼 bidirectional하게 replication이 일어나야 한다는 것을 알 수 있음.
이에 대한 조금 더 확실한 확인을 위해서 아래와 같은 실험을 수행할 수 있음.
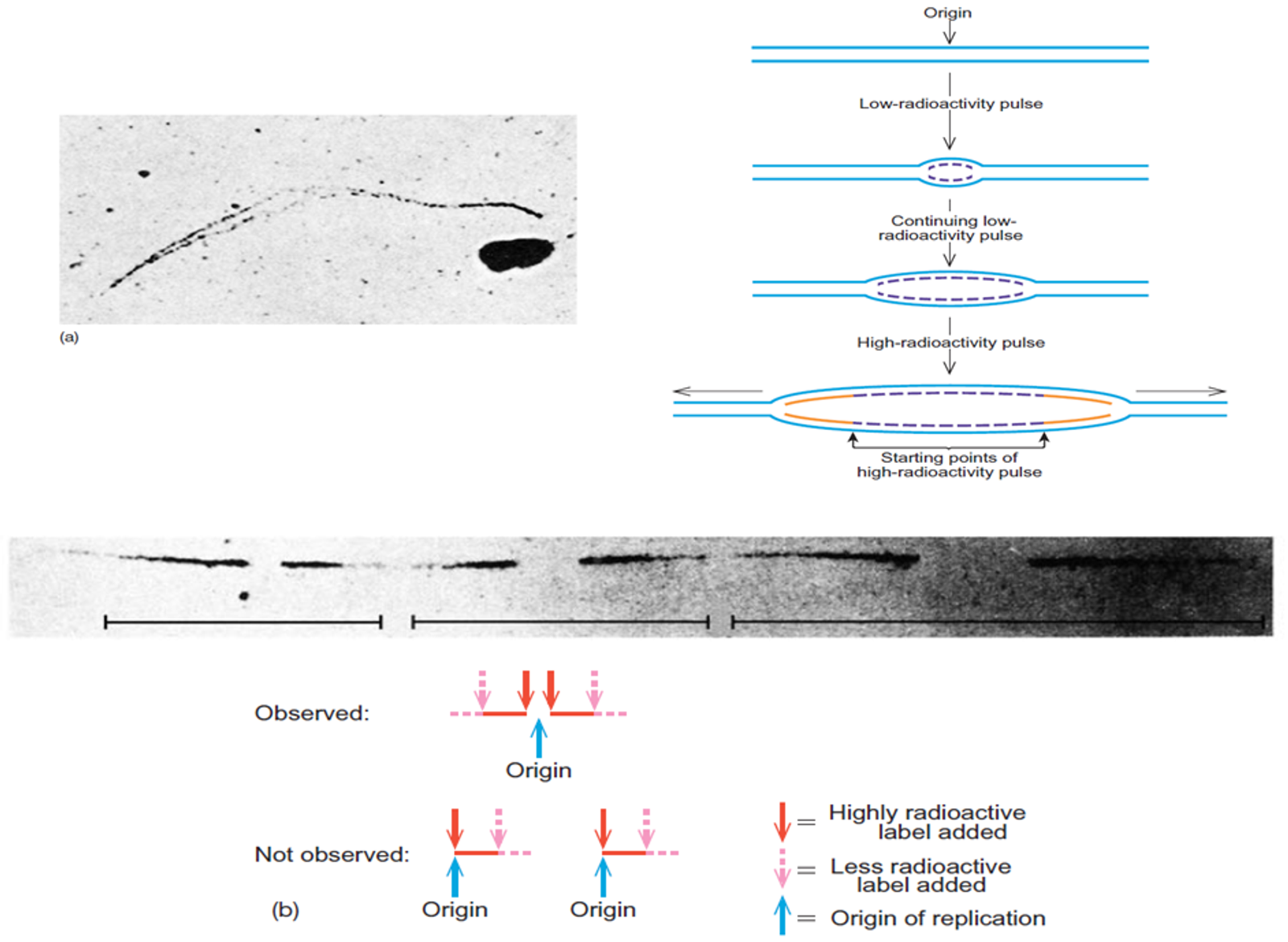
이 경우 처음에는 low-radioactivity pulse를 주다가 나중에는 high-radioactivity pulse를 준 다음에 결과물을 관찰해봄. (이러면 위 그림 오른쪽 위 맨 아래에 나타나 있는것처럼 시간차에 따른 차등적인 pattern이 생기게 될 것임) 이론적으로는 (b)에 나타난 것처럼 origin을 중심으로 대칭적인 pattern과 평행이동된 pattern 두 개가 관찰될 수 있는데, 실제로는 origin을 중심으로 대칭적인 pattern이 관찰됨. (위 그림 중간에 symmetric한 실제 관찰 결과가 나타나 있음)
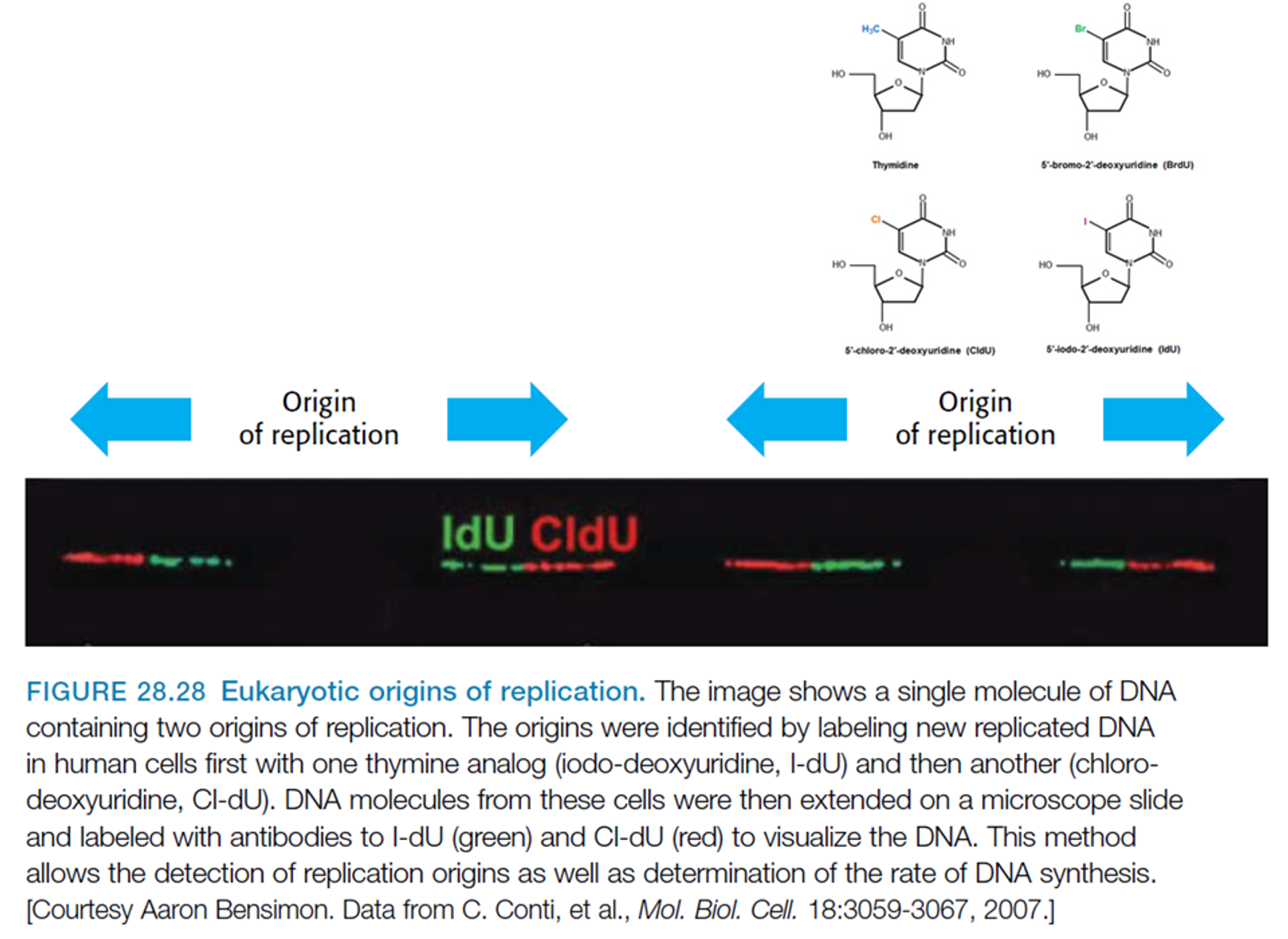
비슷한 실험을 thymidine variant(위 그림 오른쪽 위에 나타나 있음)를 이용해 수행할 수도 있음. T 자리에 T variant를 incorporation 시킨 후 이 variant를 인지할 수 있는 primary antibody, 그리고 신호를 증폭하기 위해 넣어주는 secondary antibody를 이용해서 형광을 관찰할 수 있게 됨.
실제로 위 그림 가운데에 나타난 결과는 초반에 IdU를 넣어준 다음 wash를 수행하고, 그 뒤에 CIdU를 넣고 replication을 이어서 진행시켜준 뒤 이들 각각을 인지 가능한 서로 다른 antibody를 이용해 둘을 서로 다른 형광으로 표지하고, 결과적으로 형광 pattern을 관찰한 것임. 보면 앞서와 유사하게 origin을 중심으로 symmetric한 pattern이 관찰된다는 것을 알 수 있음.
한편 본 실험과 직접적인 관련은 없지만 T의 variant 중 antibody를 사용하지 않는 표지법으로 많이 사용되는 EdU라는 녀석도 있음.
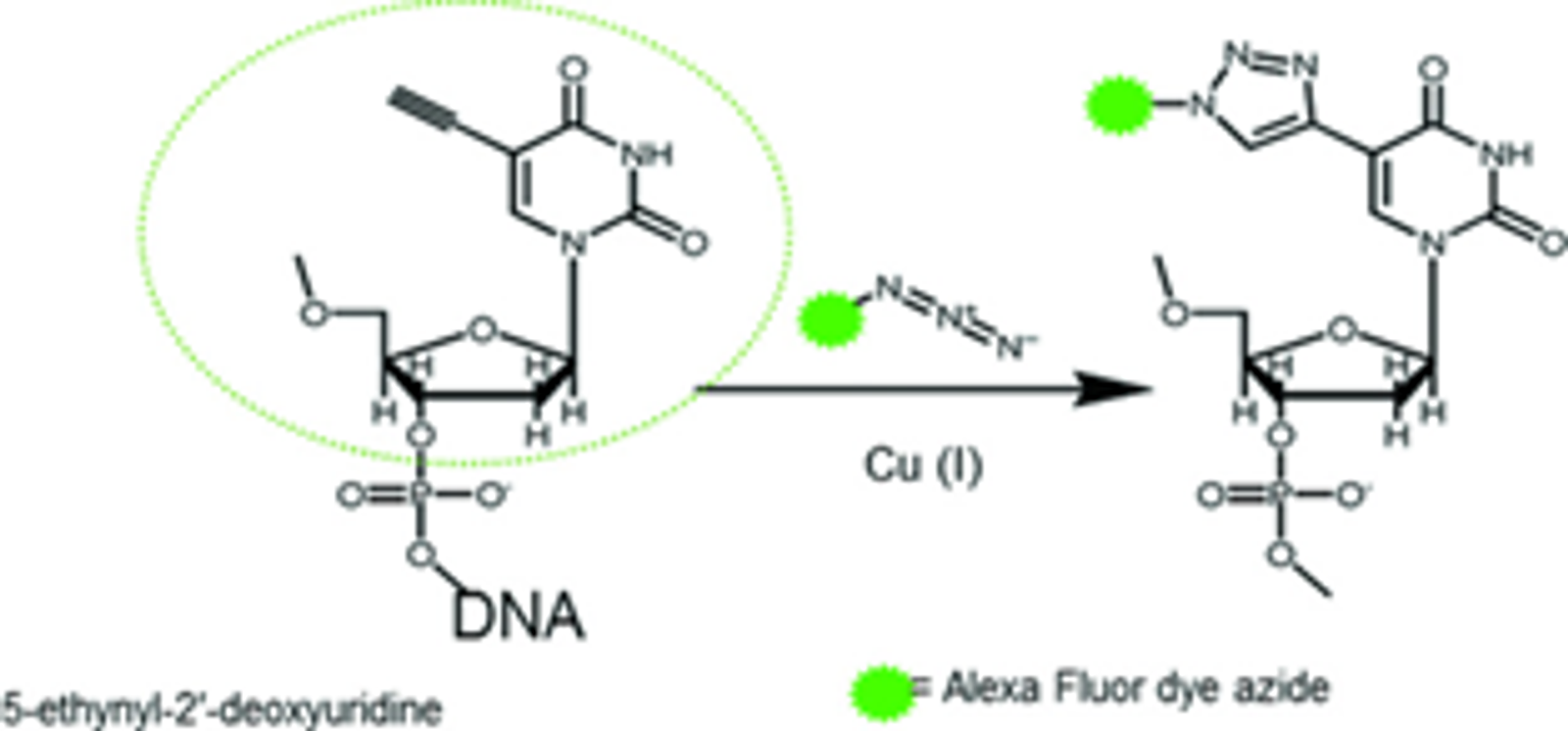
이 녀석이 표지에 사용되는 방식이 위 그림에 나타나 있음. 보면 이 녀석은 삼중결합 말단을 가지고 있음. 그런데 이 triple bond가 N3와 만나게 되면 click chemistry에 의해 바로 연결되어버림. 이 때 N3 말단부에 형광을 낼 수 있는 dye를 달아주게 되면 결국 EdU에 antibody 없이 바로 labeling을 수행할 수 있게 됨. 이 방법도 DNA replication을 measuring하는데 많이 사용될 수 있음.
그렇다면 실제로 replication fork는 어떤식으로 구성되어 있을까. 일단 unidirectional한 경우에는 한 개의 fork만 생기고 origin of replication으로부터 fork의 위치가 고정되어 있음. 한편 bidirectional한 경우에는 2개의 fork가 생기며 두 fork가 origin을 기점으로 서로 반대 방향으로 이동함.
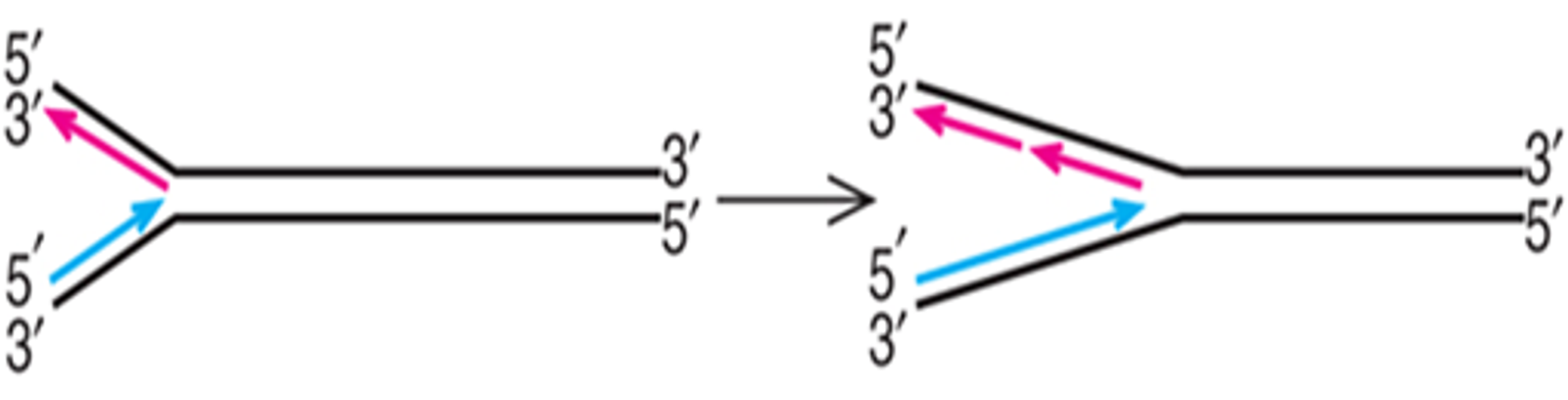
실제로는 위 그림과 같이 replication fork가 묘사됨. 위 그림에서는 이 fork가 그냥 벌어져 있는 것처럼 나타나 있지만, 사실은 fork를 만들기 위해 unzipping하는 과정에서 helicase가 작용함. (물론 이 과정에는 ATP가 필요함) 원핵생물, 진핵생물 모두 ATP를 사용해 DNA를 벌려주는 helicase가 있다는 사실은 동일하며, 그 밖에 melting이 쉬운(AT-rich한) 부분에서부터 replication fork의 형성이 시작된다는 것 또한 동일함. 다만 원핵과 진핵의 경우에는 melting이 먼저 일어나는지, machinery의 loading이 먼저 일어나는지의 선후 event 순서가 다름.
우리가 흔히 일반생물학 책에서 많이 보는 replication fork는 모두 bidirectional한 case를 상정하고 그린 그림임. 그렇다면 unidirectional한 경우에는 replication fork가 어떻게 구성될까. 일단 unidirectional한 replication이 일어나는 대표적인 예로 phage의 일종인 \phiX174가 있음. 이 녀석은 아래 그림에 나타난 것과 같이 rolling circle replication을 함.
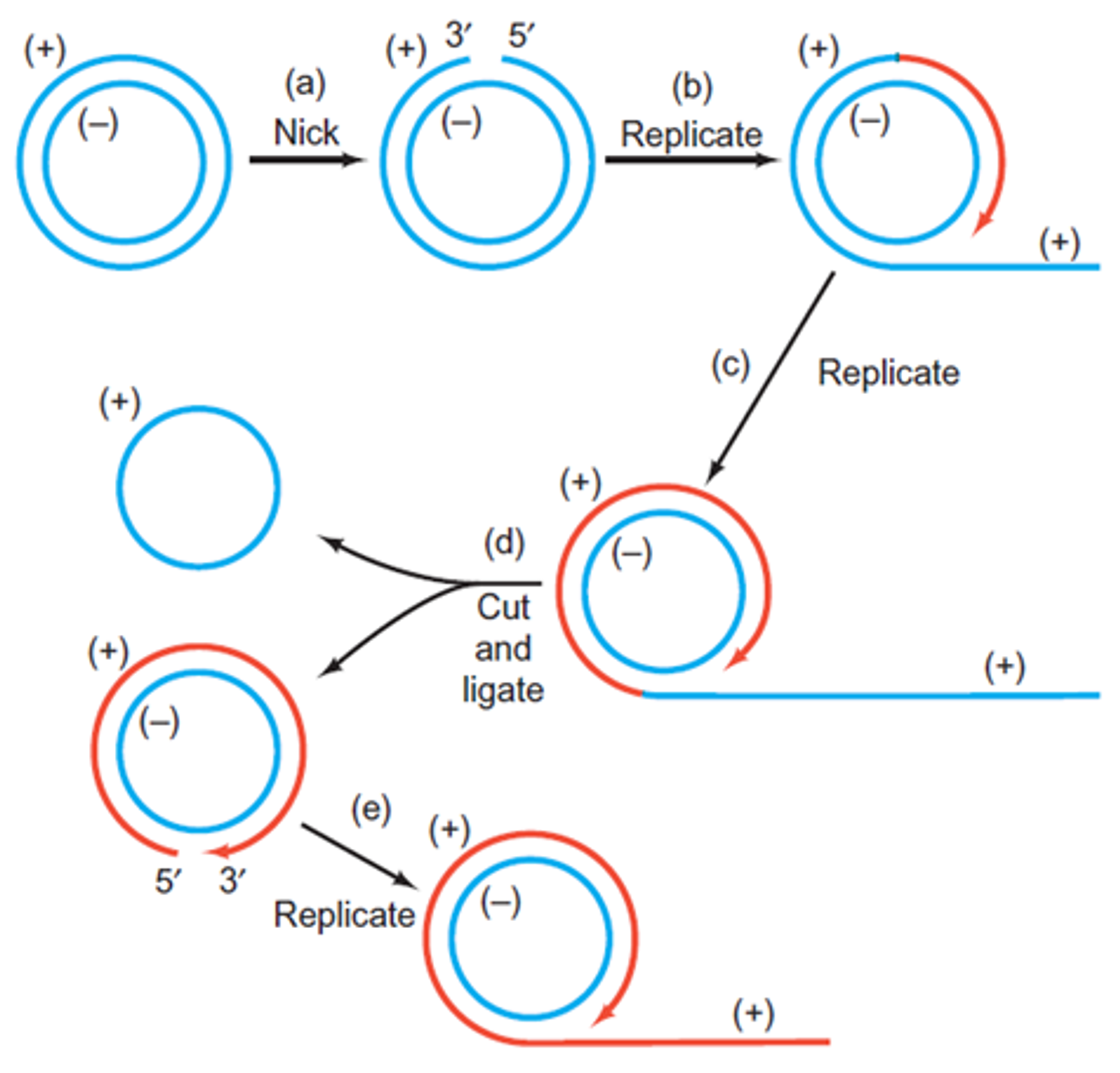
(이 때 +, -는 두 개의 원형가닥을 구분하기 위한 명명임. 이 밖에 heavy, light으로도 구분함. 특히나 heavy, light으로의 구분은 mtDNA에서 많이 이루어짐. 이 때 heavy한지 light한지의 여부는 서열의 purine 함유량에 따라 결정됨)
일단 nick을 만들어주는 효소에 의해 처음에 nick이 생김. 그러면 이 nick 중에 3' 말단 부위가 polymerase에 의해 연장되면서 원래 건너편 5' 가닥이 붙어있었던 부분을 쭉 밀어내며 replication이 일어남. (즉, strand displacement가 일어남) 참고로 이 때 원래 intact하게 DNA와 붙어있었던 건너편 DNA 가닥의 경우에도 (b)과정에서 나타난 것처럼 unzipping이 일어나야 하는데, 이 때는 별도의 helicase가 작용하지는 않고 polymerase 그 자체가 unzipping 활성을 가져서 replication을 수행하면서 동시에 건너편 가닥을 원래 가닥과 분리시켜줌)
그런 식으로 위 그림상에 빨간색으로 나타난 것과 같이 새로운 DNA가 계속적으로 합성되게 됨. 참고로 이 때 (e)와 같이 한 바퀴가 다 합성되고 난 이후에도 계속 roll이 일어나며 replication이 지속되는 것이 가능함. (polymerase가 unzipping 활성을 가져 새로 형성된 circular DNA의 5' 부분도 unzipping시켜버릴 수 있기 때문)
그렇다면 (b) 과정에서 벌어져버린 5' 파란색 서열에서는 새로운 replication이 일어나지 않는가. 사실 여기서도 일어남.
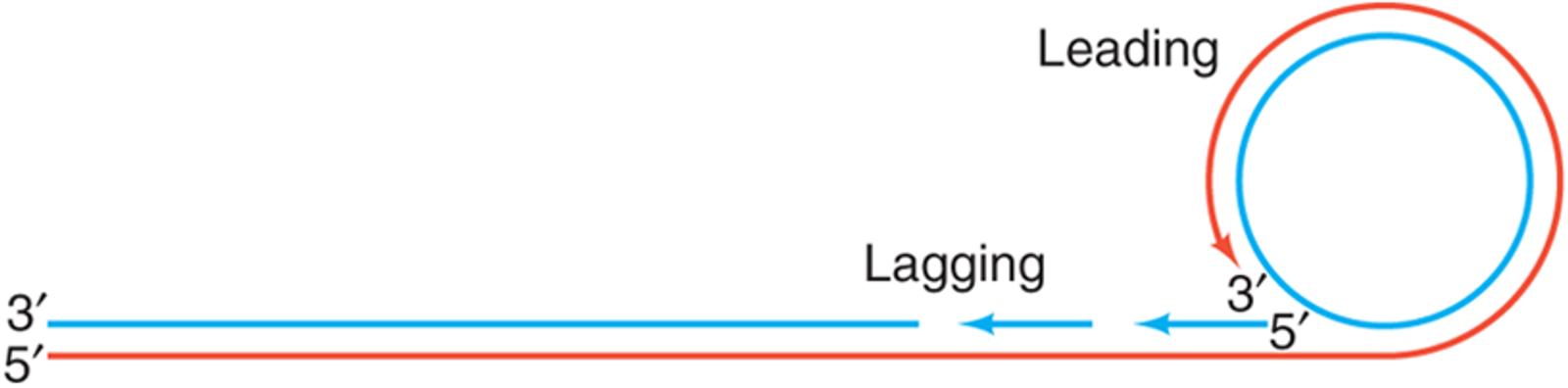
그 대신 이 경우 위 그림과 같이 5'→3'으로의 짧은 가닥끼리의 합성 후 ligase가 각각의 짧은 가닥들을 이어주어야 함. 그렇기에 앞서 그림에서 나온 빨간 가닥의 합성을 leading이라 한다면 위 그림에 나타난 파란 가닥의 합성은 lagging임.
참고로 이 때 concatemer라는 개념이 등장하는데, concatemer는 여러 genome이 반복적으로 연결되어있는 합성물을 의미함. 즉, 1배수 이상의 genome을 가지고 있는 유전정보가 바로 concatemer임. 이 concatemer는 추후에 하나의 genome에 해당하는 양씩 clipped off됨.
다음 포스트에서부터는 DNA replication에 관여하는 각종 효소들에 대해 알아보자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 19.2 : enzymology of DNA replication - 2 (1) | 2024.08.17 |
|---|---|
| [분자생물학] 19.2 : enzymology of DNA replication - 1 (1) | 2024.08.10 |
| [분자생물학] 18.3 : 번역 종결(translation termination) - 3 (0) | 2024.08.10 |
| [분자생물학] 18.3 : 번역 종결(translation termination) - 2 (0) | 2024.08.10 |
| [분자생물학] 18.3 : 번역 종결(translation termination) - 1 (0) | 2024.08.10 |