지금부터 DNA replication 시 필요한 각종 enzyme들에 대해 알아보자.

하나하나 살펴보자.
우선 위 그림의 왼쪽 윗부분부터 살펴보자. 이 때 주황색으로 표시된 DNA가 새롭게 합성된 DNA이고 파란색 DNA가 leading-strand template임. 한편 이 때 남색으로 표시된 C자모양 단백질이 DNA polymerase임. 그리고 그 바로 뒤쪽에 붙어있는 하늘색 단백질은 sliding clamp로, 이 녀석은 DNA polymerase가 DNA와 더 잘 붙을 수 있도록(affinity를 증가시켜줌) 해줌.
한편 녹색으로 표시된 단백질은 DNA helicase이며 이 녀석이 downstream에 있는 dsDNA를 풀어줌. 한편 특히 E. coli의 경우에는 DNA helicase와 함께 DNA primase(primer를 만들어줌)가 같이 붙어서 complex를 이루고 있는데, 이 complex를 primosome이라고 부름. 그리고 그보다 더 downstream을 보면 topoisomerase가 있는데, 뒤에서 다시 살펴보겠지만 topoisomerase, DNA gyrase와 같은 녀석들은 과꼬임이 일어난 DNA 부분을 다시금 풀어주는 역할을 함. (보통 topoisomerase, gyrase는 원핵생물과 진핵생물에서 상당히 구조가 다르므로 이를 이용해서 원핵생물의 topoisomerase, gyrase만을 targeting할 수 있는 항생제들도 많이 만들어지고 있음)
한편 위 그림 왼쪽 아래에 나타난 lagging strand를 살펴보자. 이 경우에는 조금만 생각해보면 leading strand와는 달리 별도의 단백질 없이 single strand 상태로 노출된 부위가 생길 수밖에 없음. (fork의 이동방향과 DNA의 합성방향이 반대이기 때문) 그런데 이런 부위는 nuclease에 의해서 쉽게 분해될 수 있음. 따라서 이를 막기 위해서 이 부분에 SSBP(single-strand DNA-binding protein)가 붙음.
참고로 b에서도 나타난 것과 같이 lagging strand도 leading strand와 DNA 합성 방향을 맞추기 위해서 중간에 한번 꼬인 채로 구조를 형성하고 있음.
DNA polymerase
이제 본격적으로 DNA polymerase에 대해 먼저 알아보자.
E. coli는 크게 pol I, pol II, pol III 세 종류의 DNA polymerase를 가짐. 이 중 E. coli가 가지는 DNA polymerase I이 polymerase 중 처음으로 identify된 녀석임. (그 유명한 Arthur Kornberg가 이를 발견함)
그렇다면 DNA polymerase는 어떤 실험에 의해 발견된 것일까.

기본적으로 위 그림 중간에 나타나 있는 것과 같은α labeling된 NTP를 이용해서 발견됨. 이α labeling NTP와 DNA를 넣어주고, 단백질이 포함된 특정 fraction을 같이 첨가시켜주었을 때, 단백질 중 DNA를 중합시키는 활성이 있는 녀석이 있다면 기존의 DNA에 labeled NTP가 붙어서 결과적으로 radioactivity가 증가할 것임.

우선 위 그림에는 3단계의 stage를 거쳐서 labeled NTP를 제작하는 과정이 나타나 있음. 보면 labeled dNMP에 ATP를 처리한 후 kinase activity를 통해서 labeled dNTP를 제작하게 됨. 이 때 labeled dNTP만 제작하기 위해서 labeled dNMP는 제거해주어야 하는데, 이를 위해서 phosphatase를 처리해줌.

위 그림은 본격적인 실험 결과임. 우선 위 그림 왼쪽에 나타난 table을 살펴보자
이 경우 다양한 purification step을 거치면서 fraction들을 엄청나게 많이 얻어내고, 각각의 fraction에 대해서 protein 양에 따른 radioactivity를 측정한 후 상대적으로 radioactivity가 큰 fraction을 계속 모으는 방식으로 농축을 수행함. 결과적으로 마지막 step에서는 - charge protein을 분리하는 DEAE를 통해 한번 더 분리해줌.
(참고로 이 때 사용된 streptomycin은 단백질을 침전시켜주기 위해 사용하고, ammonium sulfate는 protein surface charge를 중화하고 결과적으로 단백질을 fractionation하는 효과를 내기 위해 사용함)
결과적으로 이런 농축의 마지막에 얻어낸 enriched fraction을 이용해서, 이 fraction 내에 있는 단백질군중에 polymerase 활성을 띄는 녀석이 있는지를 확인해보기 위해 위 그림 오른쪽 위와 같은 실험을 수행함.
이 때 측정한 것은 DNA가 incorporation되는 정도이며, 그 결과 polymerization을 위해서 nucleotide, Mg2+와 같은 성분들이 다 필요하다는 것을 알 수 있음. (특히 4종류의 nucleotide가 다 필요함)
사실 E. coli의 경우에 DNA polymerase I이 genome에 있어서 main replication enzyme은 아님. main enzyme은 바로 DNA polymerase III임. 다만 DNA polymerase I도 다양한 기능을 가지고 있음.
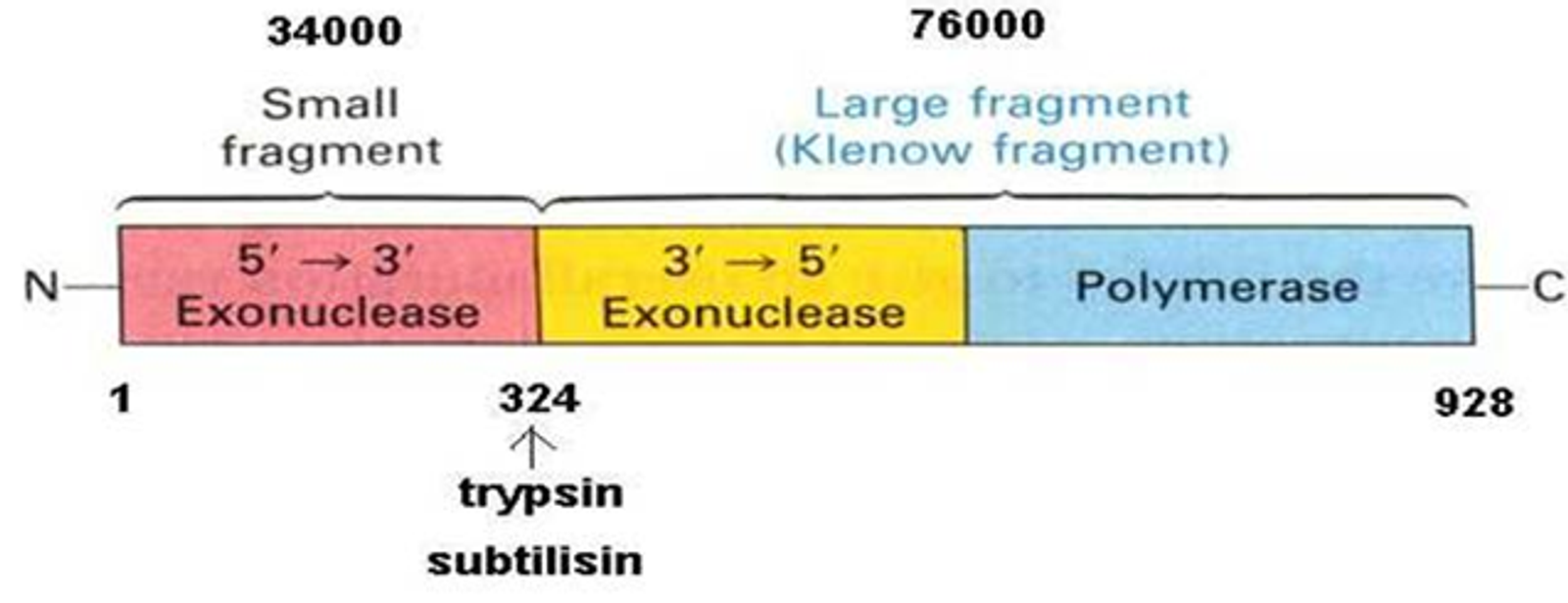
DNA polymerase I의 holoenzyme structure는 위와 같음. 보면 5'→3' exonuclease, 3'→5' exonuclease, 5'→3' polymerase 활성이 있는 3개의 domain으로 이루어져있다는 것을 알 수 있음. 그런데 이 때 324 부분이 trypsin에 의해 잘려나갈 수 있어서, 실제로는 5'→3' exonuclease 활성을 가지는 small fragment, 3'→5' exonuclease 활성, polymerase 활성을 가지는 large fragment(Klenow fragment) 등으로 분리된 채로 존재하는 경우도 많음.
이 때 5'→3' exonuclease 활성은 주로 end processing을 위해 많이 사용되고 3'→5' exonuclease 활성은 주로 backtracking에 이어서 수반되는 proofreading을 위해 많이 사용됨. (결국 3'→5' exonuclease 활성은 DNA replication의 error rate를 낮춰주고 결과적으로 fidelity를 높여줌)
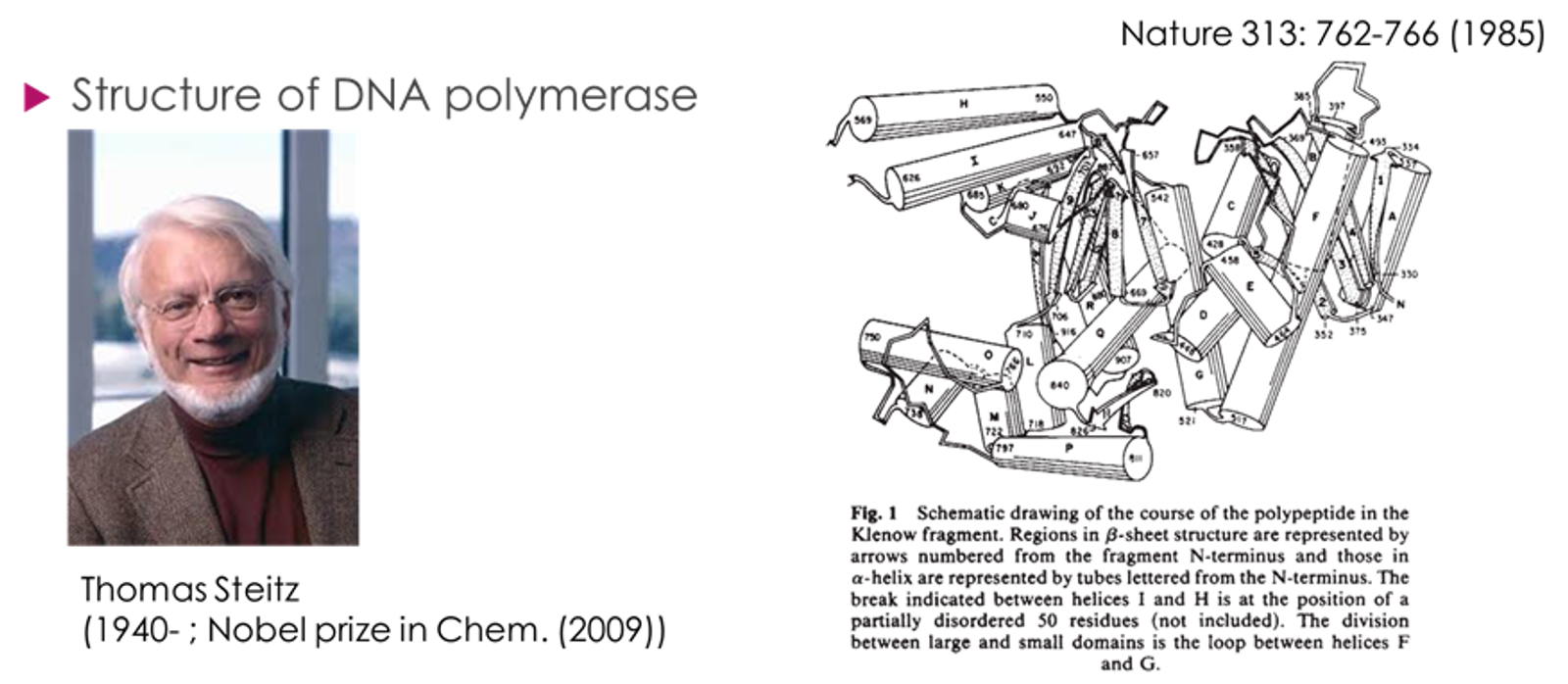
실제로 Thomas Steitz는 x-ray crystallography를 통해서 위 그림 오른쪽과 같이 Klenow fragment의 구조를 밝힘.
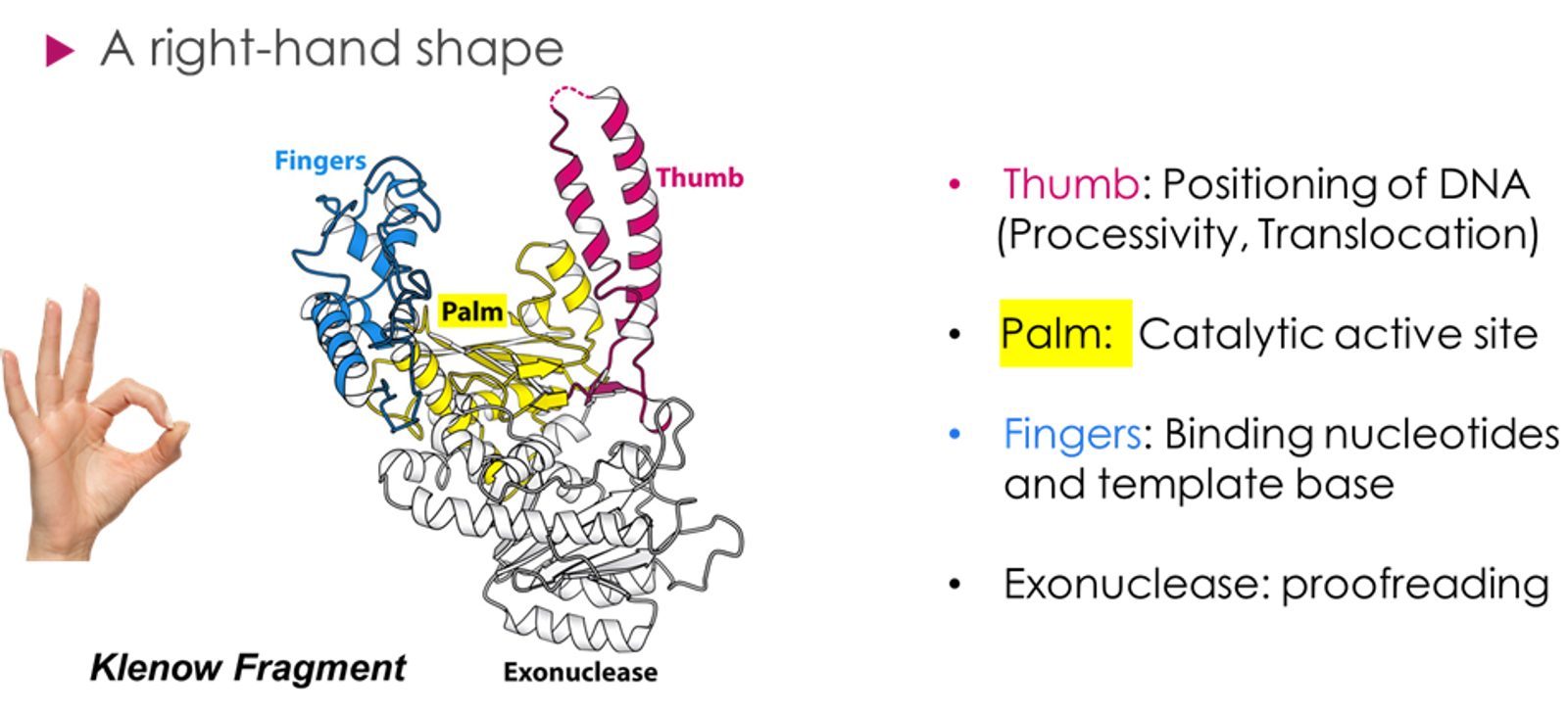
그 결과 Klenow fragment가 위와 같은 구조를 가지고 있음을 확인함.
보면 위 구조 아랫부분(회색)이 3'→5' exonuclease 활성을 가지는 부분이고 위쪽이 polymerase 활성을 가지는 부분임. polymerase 활성을 가지는 부분은 다시금 3개로 나눠지는데, Thumb, Palm, Finger가 그것임. (합쳐서 오른손으로 많이 비유됨) Thumb의 경우 DNA의 positioning에 관련되어있고 Palm은 catalytic active site이며 finger는 nucleotide의 binding이 일어나는 부위임. 결과적으로 nucleotide가 붙게 되면 전체 구조가 오므려지고 반응이 끝나면 다시 펴지게 됨. 이 과정에서 특히 finger 부위가 nucleotide와 template base를 손가락 집게와 같이 잡아주는 역할을 수행하게 됨.
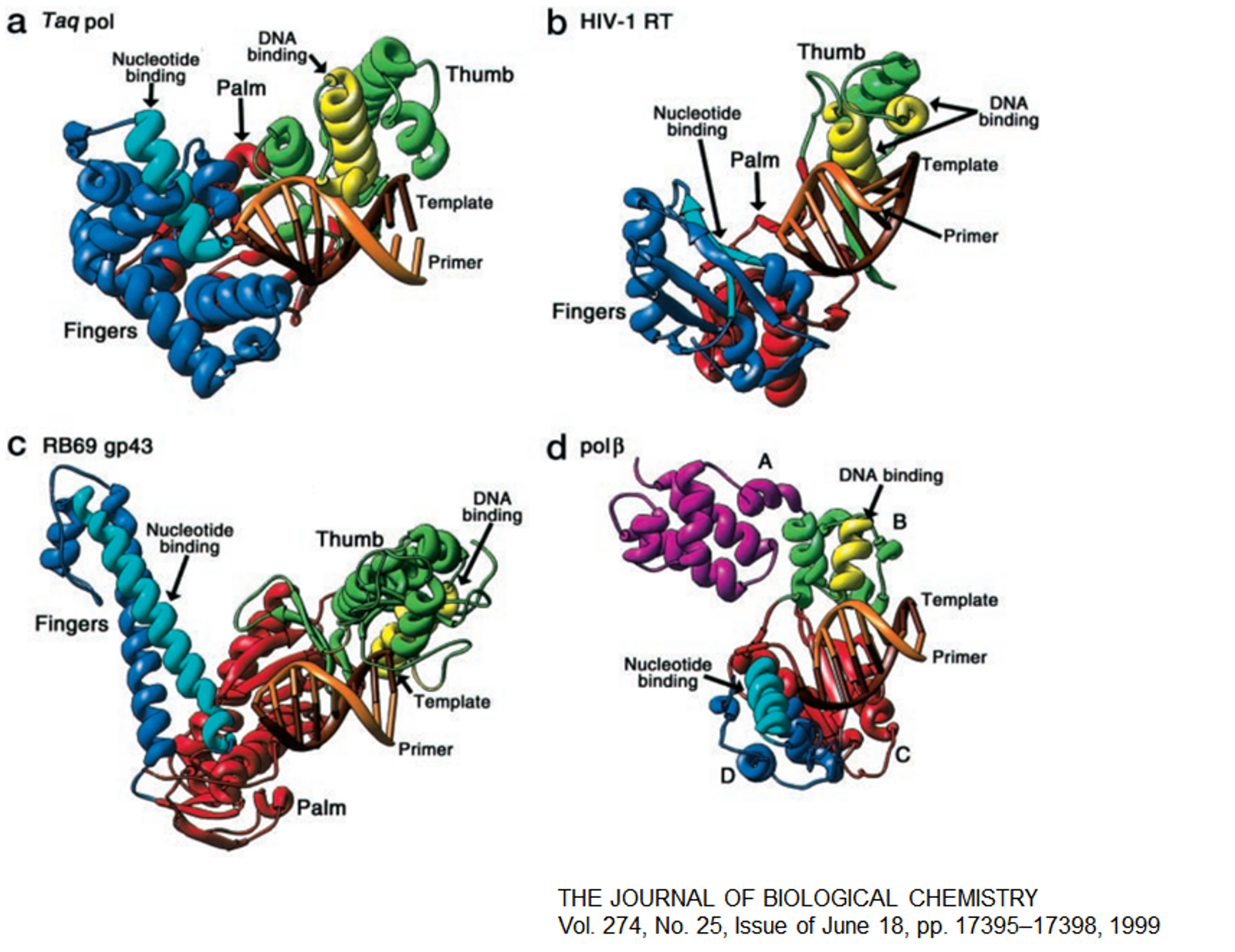
한편 위 그림을 보면 매우 많은 종류의 pol 혹은 pol 활성을 가지는 다른 단백질들이 나와있는데, 이들 간에 어느 정도의 homology가 관찰됨. (다만 Taq polymerase의 경우에는 나머지는 비슷하나 3'→5' exonuclease가 없음. 이 때문에 error rate이 엄청나게 높음)
다음으로 구체적으로 5'→3' exonuclease 활성이 어떤 식으로 이용되는지에 대해 살펴보자.
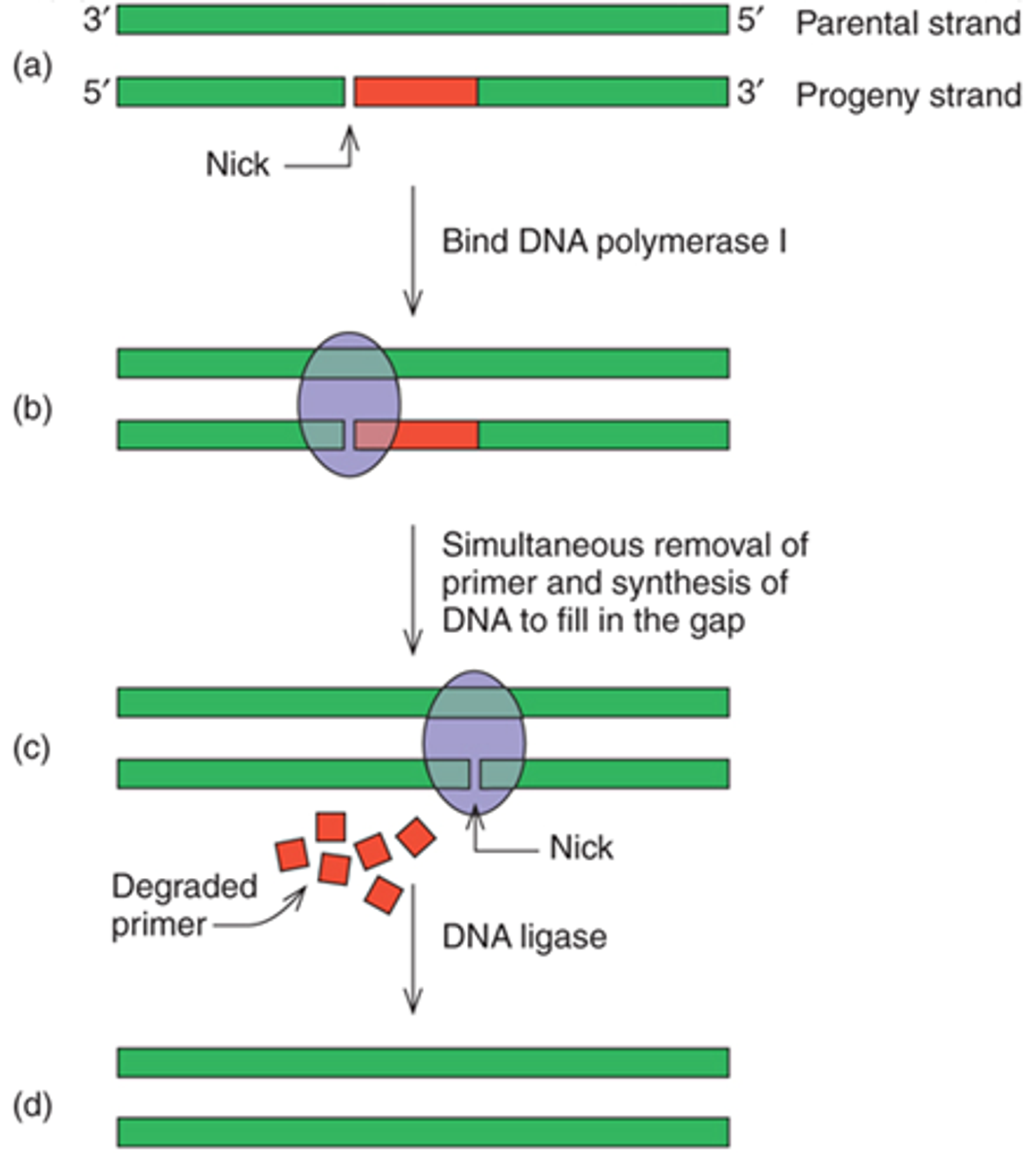
위 그림상에 나타나 있는 것처럼 DNA 상에 nick이 존재하게 되면 이 nick 부위를 DNA polymerase 활성을 가지는 부위 앞쪽의 5'→3' exonuclease 부위가 인지하게 되고, 그 결과 primer의 remove가 일어나게 됨. 이후 뒤따라오는 polymerase에 의해서 remove된 부분이 다시금 DNA로 중합되게 됨. 한편 그렇게 nick이 계속 이동해가다가 결국에는 이 nick이 DNA ligase에 의해서 연결됨.
5'→3' exonuclease 활성을 이용하면 labeling을 수행할 수도 있음. nick이 있는 strand가 있을 시 이 nick을 5'→3' exonuclease가 갉아먹는 과정에서 radioactive nucleotide를 넣어주게 되면 결과적으로 원래 nick의 위치보다 downstream 위치에 labeled nucleotide가 들어가게 되고 strand를 labeling할 수 있게 됨. (nick translation)
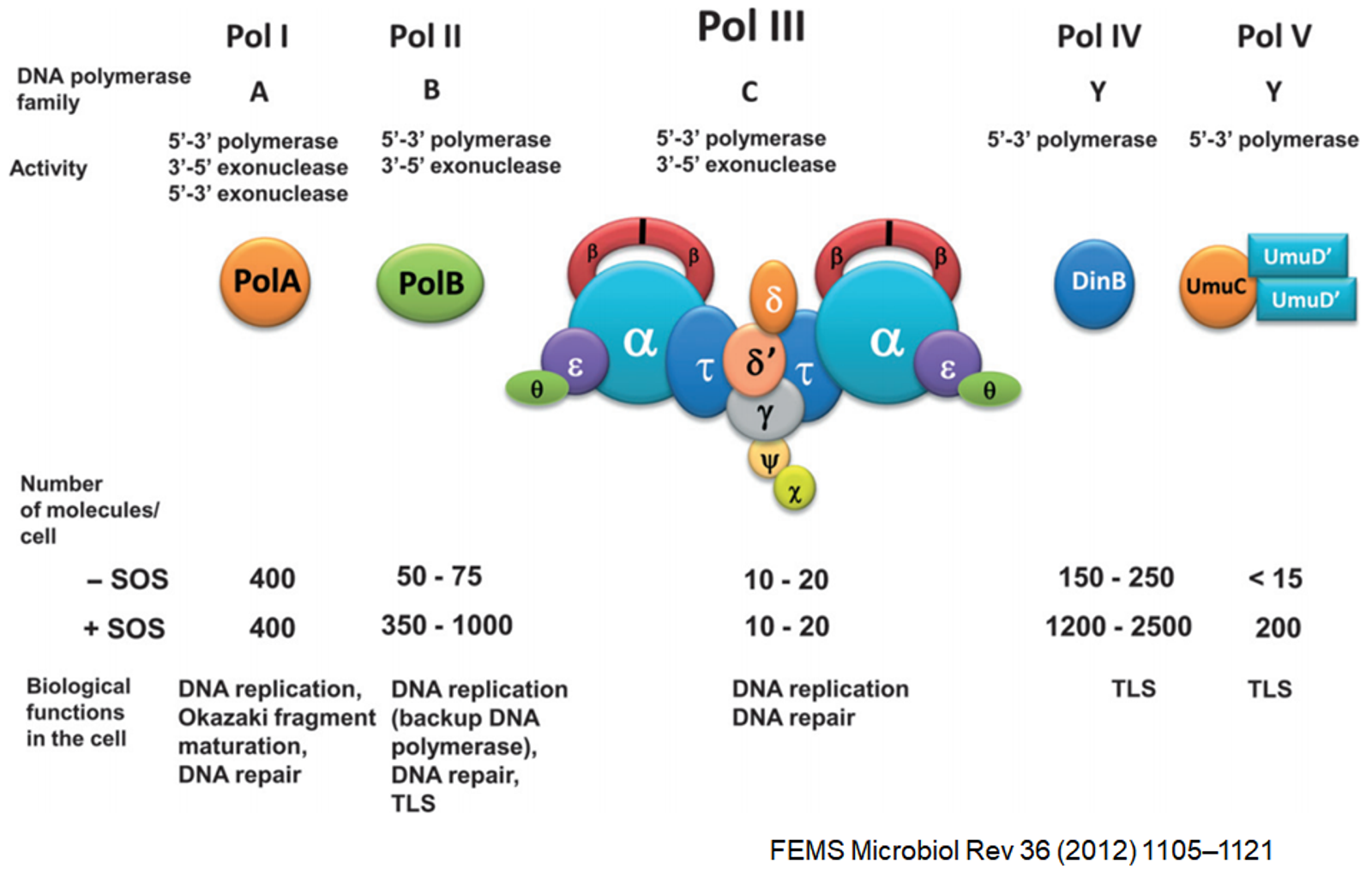
사실 E. coli에서 DNA polymerase의 종류는 I, II, III말고 IV, V까지 있음. 그러나 이 중 IV, V는 최근에 발견되었으므로 대부분의 교재에는 I, II, III만 나와있음. (IV, V는 주로 repair activity에 중요)
pol I은 앞서 본 것과 같은 활성을 가지며, DNA replication, Okazaki fragment의 maturation(primer 제거), DNA repair 등에 관여함. 한편 pol II는 5'→3' and 3'→5' exonuclease 활성을 가지며 주로 DNA repair에 관여함. pol III는 5'→3' polymerase, 3'→5' exonuclease 활성을 가지며 이 녀석이 주로 DNA replication에 많이 관여함. pol III는 위 그림에서도 나타나 있는 것처럼 매우 거대하며, 이 중 α에 해당하는 소단위체가 실제로 polymerase activity를 가짐.
그러면 pol III를 구성하는 각각의 subunit들은 어떤 역할을 수행할까.
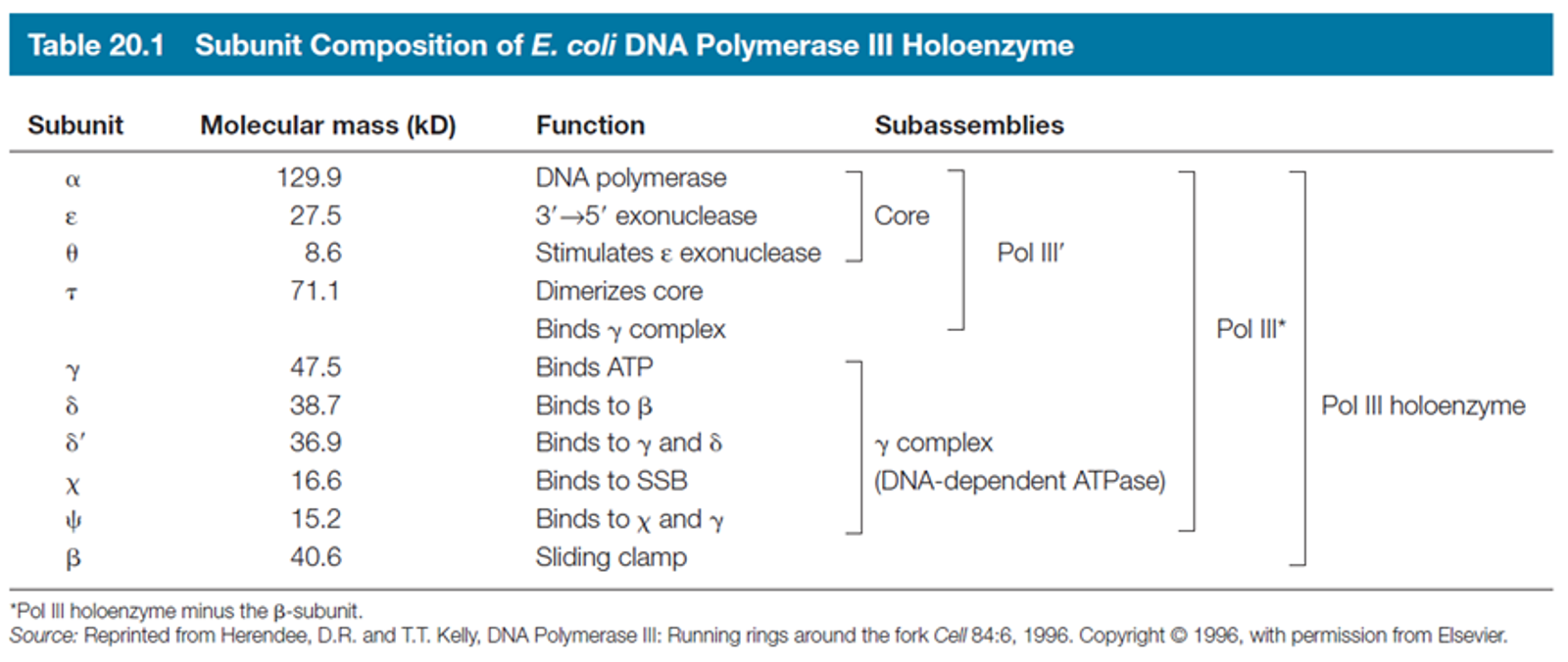
이와 관련된 table이 위에 제시되어 있음.
이 때 α, ε, θ를 합쳐서 core라고 부르며, 이 core와 τ를 합쳐서 pol III'이라고 부름. pol III'은 exonuclease 활성, polymerase 활성을 다 가지고 있음.
한편 τ에 의해서 pol III'에 γ complex라는 녀석이 recruit되고 결국 이 recruit에 더해 γ complex에 의해 recuit되는 β에 의해서 pol III holoenzyme이 형성됨. 이 때 γ complex는 DNA-dependent ATPase 활성을 가짐.

위 그림상에 녹색으로 나타나 있는 것이 core, 주황색 혹은 빨간색으로 나타나 있는 것이 τ이며, τ에 의해서 γ가 recruit되어있는 모습이 보임. 그렇다면 γ는 도대체 DNA-dependent ATPase 활성으로 무슨 일을 수행할까.
γ complex는 결과적으로 clamp loader로 기능함.
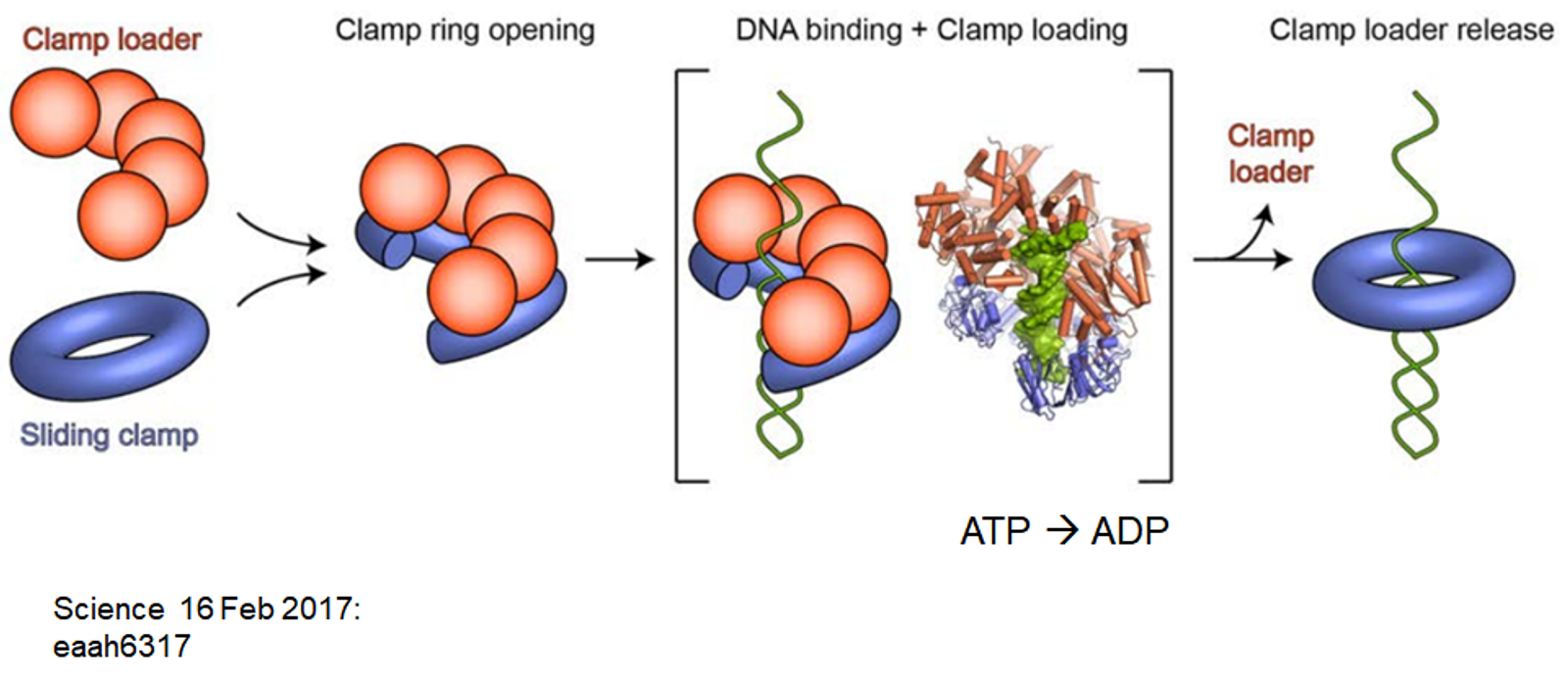
위 그림에 sliding clamp(e.coli의 경우 β가 sliding clamp 역할을 하는 반면 진핵생물의 경우 PCNA가 sliding clamp 역할을 함)와 \gamma complex가 나타나 있는데, 보면 γ complex에 의해서 sliding clamp의 일부가 절단된 후 DNA가 hole을 투과하게 되고, 이후에 다시금 sliding clamp가 닫힌 형태로 돌아가게 됨. (이 과정에서 γ complex가 가진 ATPase 활성에 의한 ATP→ADP로의 분해가 필요함)
다음으로 DNA polymerase III의 replication 속도에 대해 알아보자. 결과적으로 E. coli의 경우 replication 속도는 대략 1,000nt/sec 정도임. (이는 E. coli의 비정상적으로 빠른 분열속도에 replication 속도를 맞추기 위한 진화의 결과임) 그렇다면 이 속도값에 대해서는 어떻게 알게 되었을까.
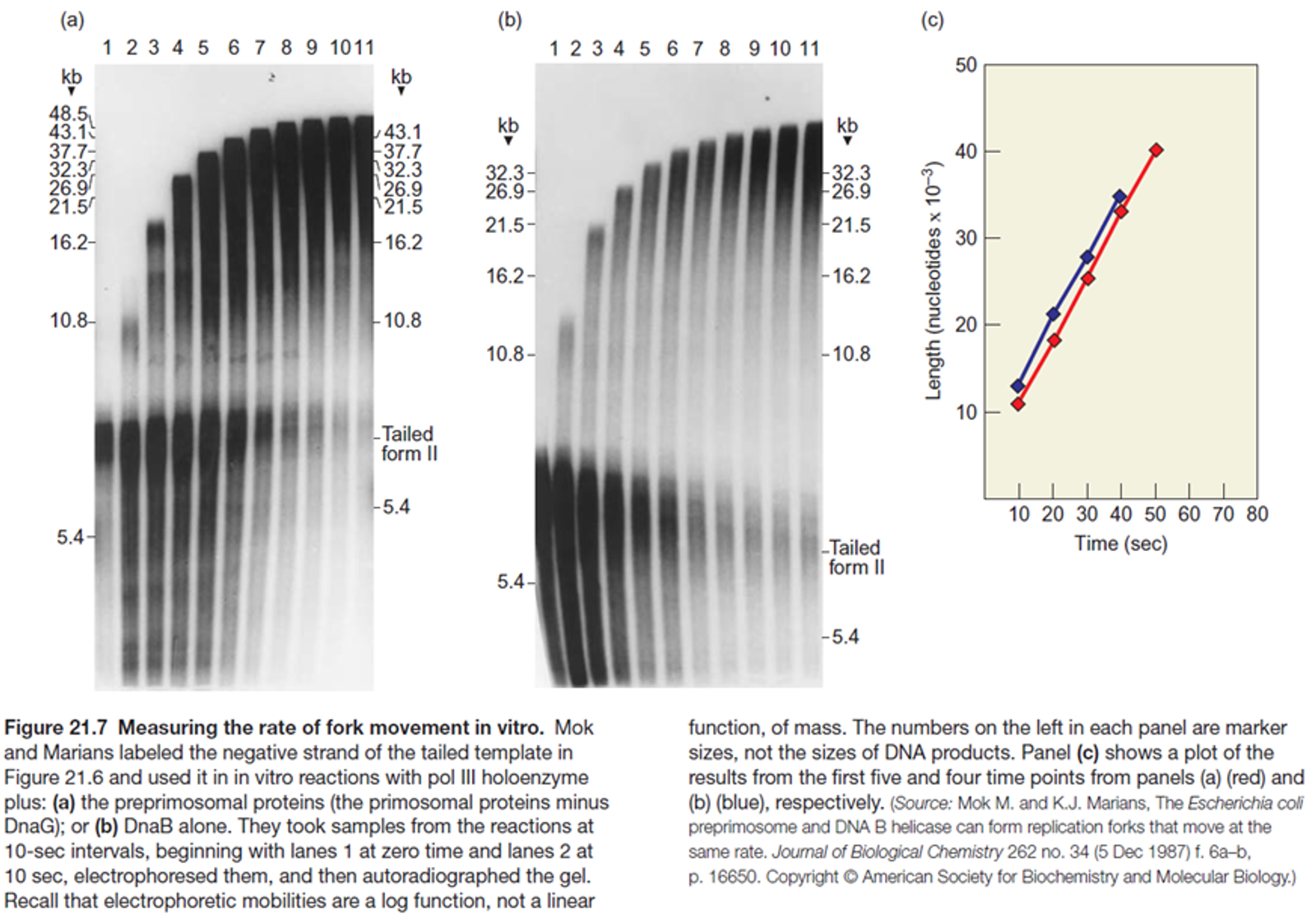
위와 같은 실험 결과를 통해 알게 됨. 이 실험은 기본적으로 특정 짧은(길이를 아는) 가닥을 긴 가닥에 붙인 후 짧은 가닥의 5'에 kinase를 이용해 labeling을 한 채로 DNA polymerase III를 넣어준 후 특정 시간이 지났을 때마다 신장된 길이의 정도를 확인한 것임. 구체적으로 위 그림상의 gel에서 y축이 DNA length가 늘어나는 정도를 의미하며 이를 위해 nucleotide radioactive pulse를 가해줘서 radioactive strand를 labeling함. 이런식의 실험 과정을 통해서 replication 속도를 알게 됨.
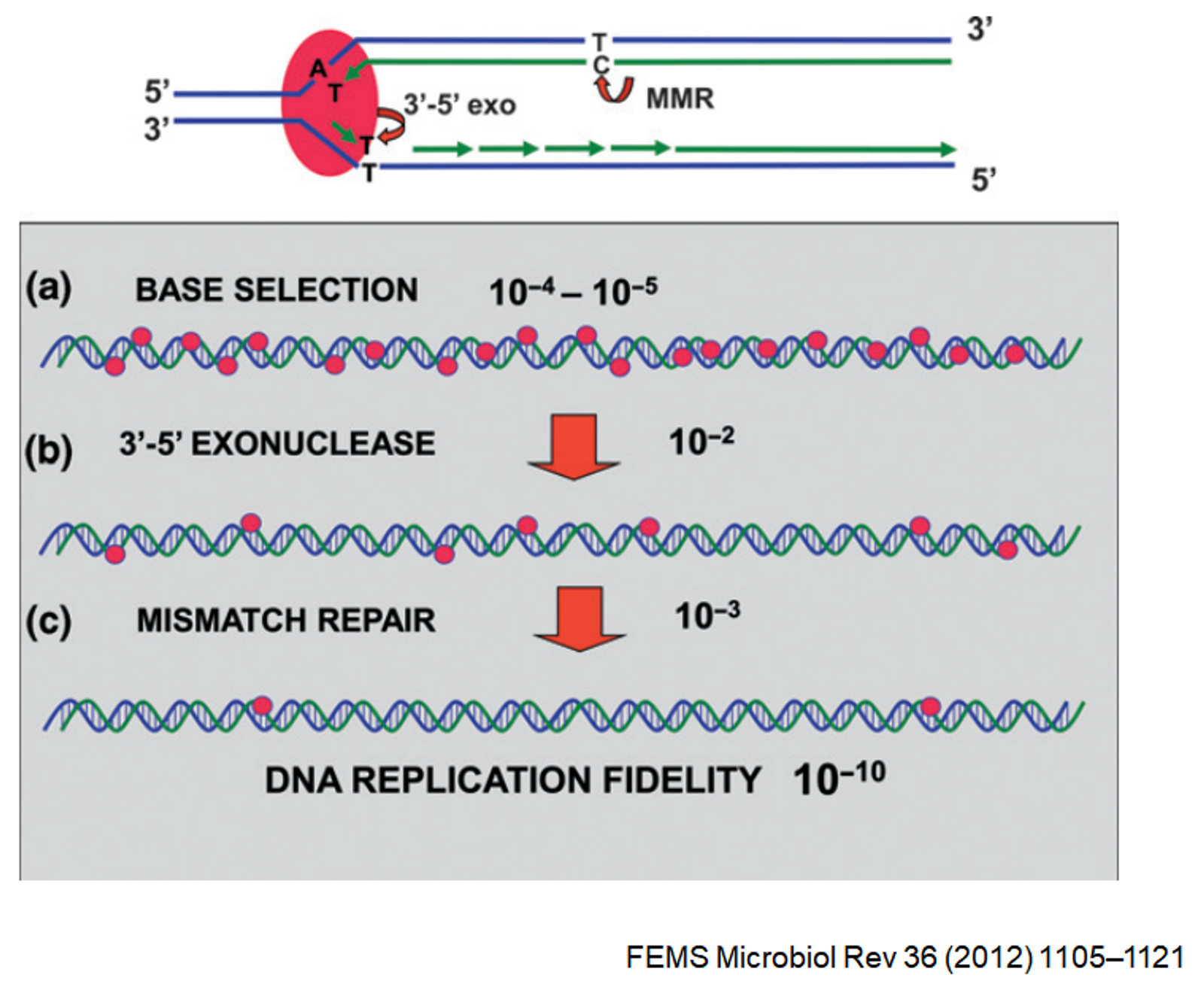
다음으로 앞서 언급했었던 replication 과정에서의 fidelity 문제에 대해 논해보자. 일단 base selection 과정에서 자발적으로 발생하는 error의 rate은 10^(-4)- 10^(-5) 정도임. 그런데 3'-5' exonuclease 활성에 의해서 error 중 10^(-2) 정도는 다 수선되고, 더불어 mismatch repair(MMR)를 통해서 10^{-3) 정도가 수선됨. 결과적으로 이런 수선이 다 이루어지고 나면 DNA replication fidelity가 10^(-10) 정도가 됨. (high fidelity)
다만 Taq polymerase 같은 경우에는 exonuclease 활성이 없으므로 (base selection 과정에서의 error rate이 그대로 남아있게 됨) 훨씬 더 poor한 fidelity를 가짐.
다음으로 진핵생물의 DNA polymerase에 대해 간단히 알아보자.
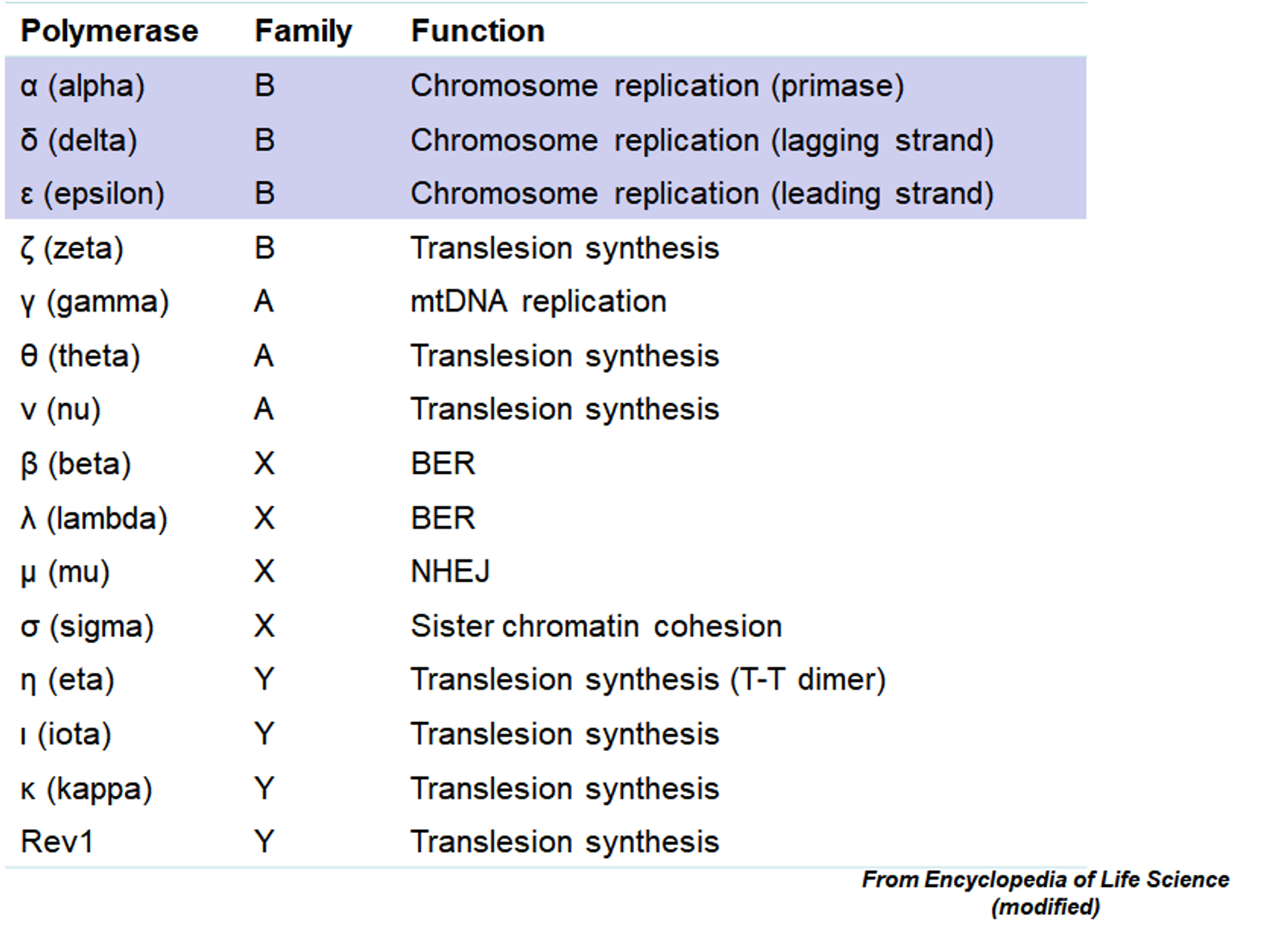
위 표에 진핵생물의 DNA polymerase가 종류별로 나열되어 있음. 이 중 보통 α, δ, ε이 많이 알려져 있음. 이 중 α는 primase로 기능하고 δ는 lagging strand의 replication에 관여하며 ε은 leading strand의 replication에 관여함. (이 3개 이외의 polymerase들은 대부분 repair와 관련되어 있음)
그 밖에 η는 T-T dimer(UV에 의해 생성)에 대응되는 base를 합성시켜주는, translesion synthesis에 관여함. (그렇기에 η가 고장나면 T-T dimer 후단의 DNA가 replication되지 않음. 즉, 딸세포가 만들어지지 않음. 이에 대한 결과로 발생하는 대표적인 질병 중 하나가 XP임) 결국 T-T dimer는 서로 인접하고 있는 두 개의 연속된 T 사이에 공유결합이 일어나는 것으로 이런 결합이 일어날 시 DNA의 rigid한 정도가 바뀌게 됨. 그런데 이렇게 이상한 T-T dimer가 형성되었다 할지라도 T-T dimer에 상보적인 base가 들어갈 수 있어야 replication이 일어날 수 있으므로 이러한 측면에서 η의 역할이 중요함.
참고로 BER은 base를 때낸 후 repair가 일어나는 현상을 의미하며, 이 때 β, λ가 관여함. 그 밖에 NHEJ는 DNA 중간에 분절이 일어났을 때 분절이 일어난 DNA끼리를 다시금 붙여주는 현상을 의미하며, 이 때 µ가 관여함.
다음 포스트에서는 DNA polymerase 이외의 나머지 단백질들에 대해 살펴보도록 하자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 20.1 : DNA 복제 개시 - 1 (0) | 2024.10.27 |
|---|---|
| [분자생물학] 19.2 : enzymology of DNA replication - 2 (1) | 2024.08.17 |
| [분자생물학] 19.1 : DNA replication(DNA 복제)의 특징 (1) | 2024.08.10 |
| [분자생물학] 18.3 : 번역 종결(translation termination) - 3 (0) | 2024.08.10 |
| [분자생물학] 18.3 : 번역 종결(translation termination) - 2 (0) | 2024.08.10 |