이번 포스트에서는 DNA polymerase 이외에 DNA replication에 관여하는 다른 효소들에 대해 알아보도록 하자.
sliding clamp
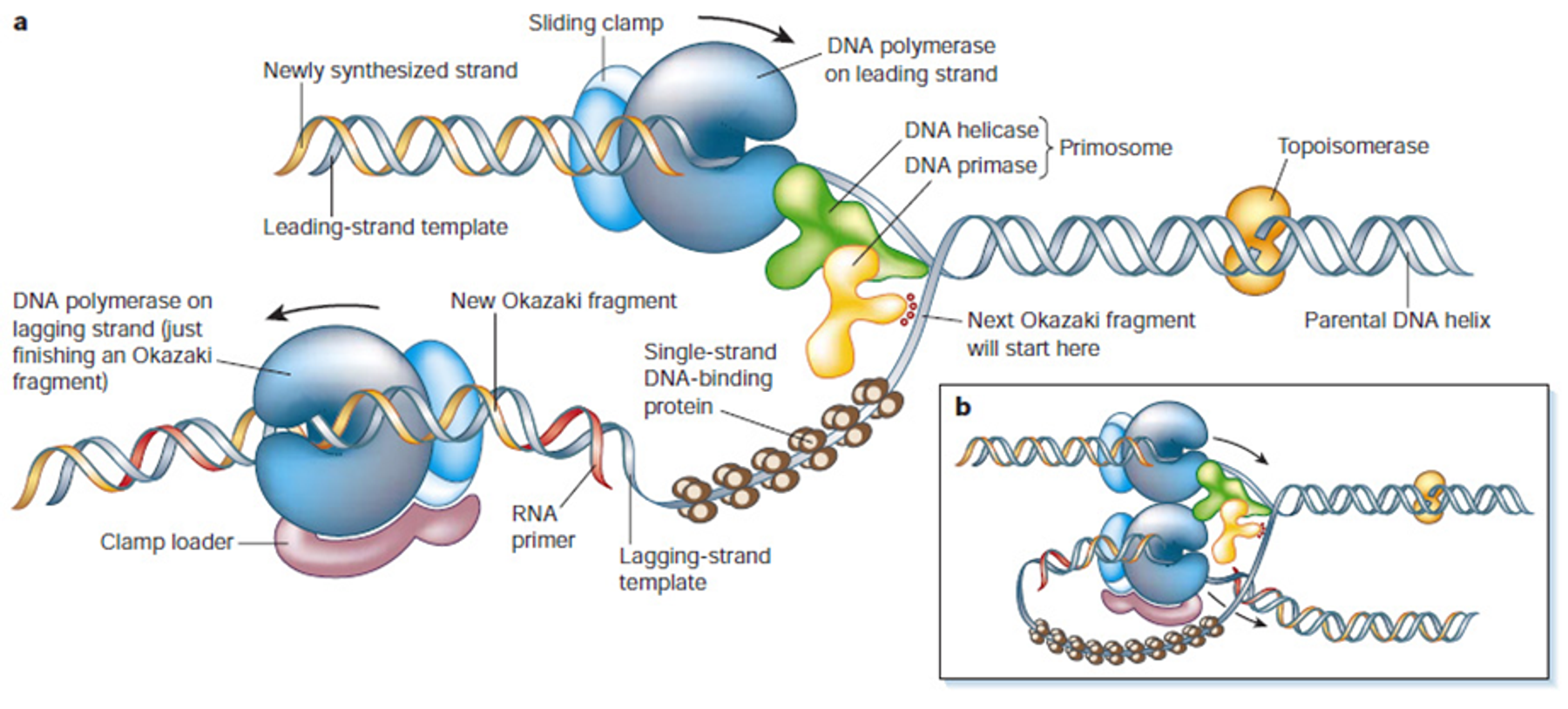
앞서 봤던 위 그림에서 하늘색으로 표시되어 있는 것이 바로 sliding clamp임. 이 녀석은 DNA polymerase와 DNA 사이의 affinity가 높아지게끔 만들어줌.
일반적으로 DNA polymerase의 합성 속도는 DNA polymerase 내의 palm 효율 혹은 DNA polymerase와 DNA, nucleotide 간의 binding하는 정도에 따라서 결정됨. 특히 후자의 경우 DNA polymerase와 DNA 사이의 binding, unbinding이 평형관계이므로 이 때 binding된 상태, unbinding된 상태 중 어느 곳에 평형이 치우쳐져 있는지에 따라서 DNA polymerase의 안정성이 달라지게 되어 결과적으로 DNA 합성 속도가 달라지게 됨. (참고로 너무 binding되는 경향이 강해버려도 문제가 되는데, 너무 강해버리면 아예 DNA polymerase가 이동하지 못할 것임. 따라서 적당히 binding이 잘 되는 것이 중요함)
만약 sliding clamp에 의해서 적당히 DNA polymerase와 DNA 간의 결합력이 높아지게 되면 DNA replication이 processive하게 일어남. 그렇기에 sliding clamp를 다른 말로 processivity factor라고 부름.

위 그림에는 진핵생물에서의 sliding clamp인 PCNA의 구조가 나타나 있음. 보면 전반적으로 원형으로 생긴 dimer라는 것을 알 수 있고, 중간에 뚫린 구멍에 dsDNA가 딱 맞게 들어가는 것을 알 수 있음.

실제로 위 실험 결과에 나타난 것처럼 PCNA의 양을 점점 증가시켜보면 DNA의 합성 길이가 점차 더 길어진다는 것을 알 수 있음. (processivity의 증가 결과)
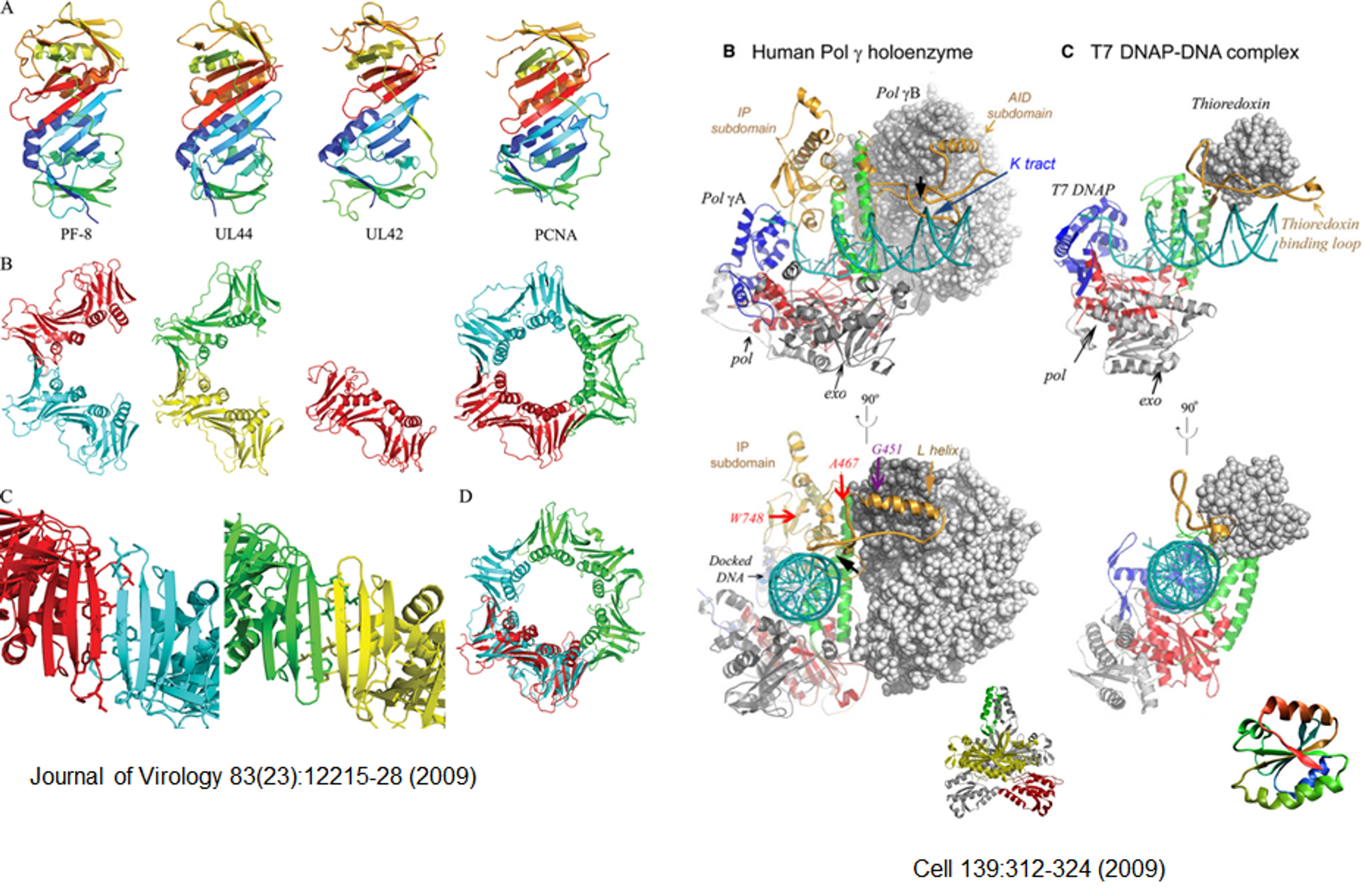
한편 processivity factor에는 위 그림과 같이 매우 다양한 종류가 있음. 보면 sliding factor가 무조건 동그란 모양인 것은 아니라는 것을 알 수 있음. (위 그림 오른쪽 구조 중 회색 큰 덩어리가 mitochondria에서의 sliding factor인데, 보면 구형이 아니고 그냥 주변부에 결합해 DNA polymerase의 binding affinity를 간접적으로 도와주는 형태로 존재함을 알 수 있음) 그리고 크기도 매우 다양함. (c의 경우 매우 작은, 그리고 hole이 없는 sliding factor(회색)임을 알 수 있음)
그리고 leading strand에 비해 lagging strand에서는 계속 DNA polymerase가 recruit되어야 하고, 이에 따라서 계속 sliding factor가 붙어야 하므로 결과적으로 lagging strand에서는 특히나 clamp loader(E. coli에서는 γ complex)가 필요함.

그래서 위 그림상에서 특히 lagging strand에서 clamp loader가 표시되어 있는 것임.
DNA helicase
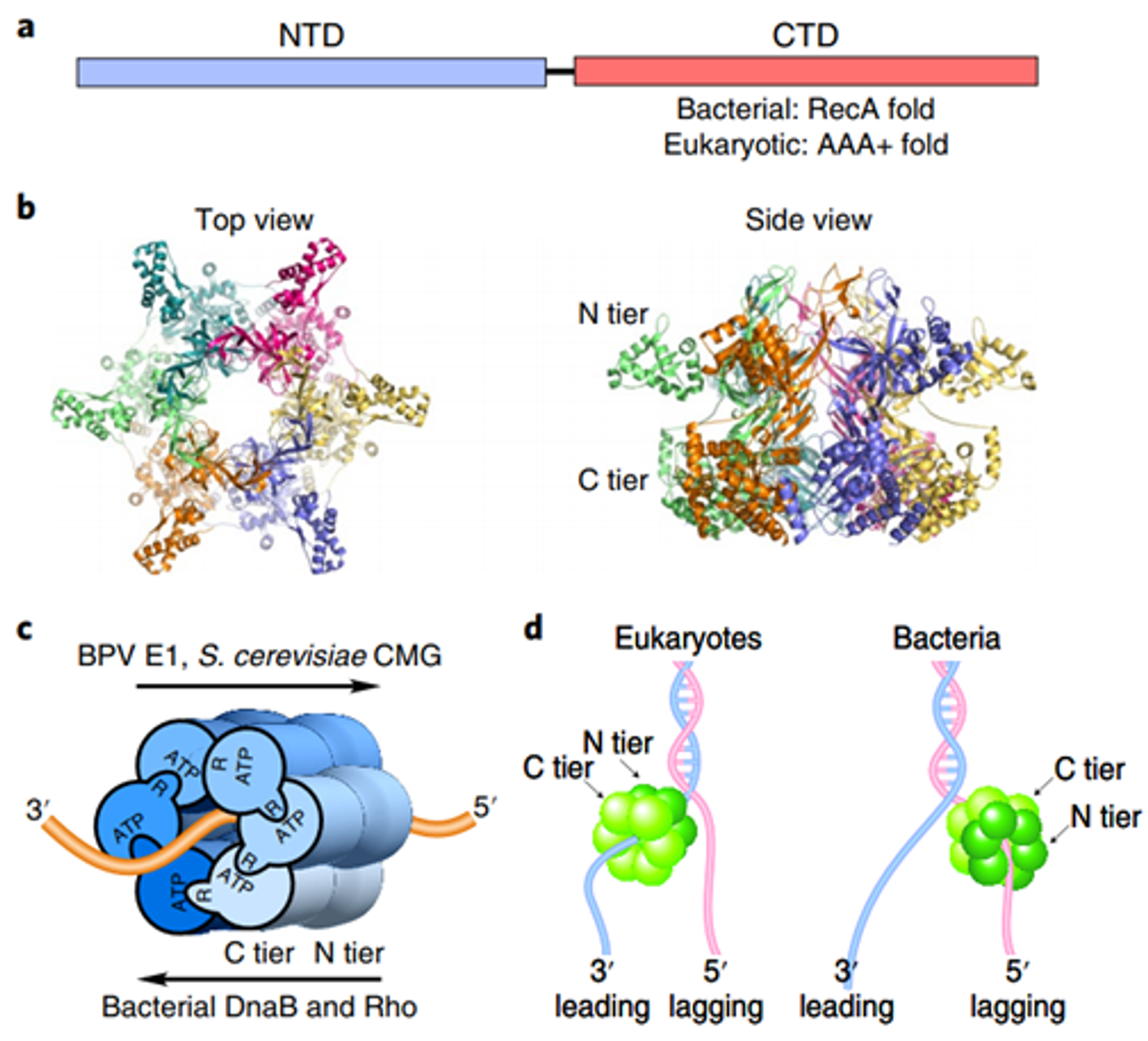
DNA helicase는 일반적으로 위 그림과 같이 hexamer를 이룬 채 존재하고 있음. 이는 CTD에 있는 RecA fold(bacteria), AAA+ fold(eukaryote) 때문인데, 이들이 hexameric fold로 작용해서 각각의 monomer들을 hexamer로 뭉칠 수 있게 해줌.
(참고로 hexamer를 구성하는 각각의 monomer들은 하나하나 ATPase 활성을 가짐)
일단 DNA helicase는 system에 따라서 어떤 서열을 따라서 tracking하며 unzipping을 수행할지가 다른데, eukaryote에서는 위 그림에서도 나와있는 것처럼 leading strand에 붙어서 unzipping을 수행하고 bacteria에서는 lagging strand에 붙어서 unzipping을 수행함. (이는 fold의 차이에 의해 directionality가 달라져서 발생하는 현상임)
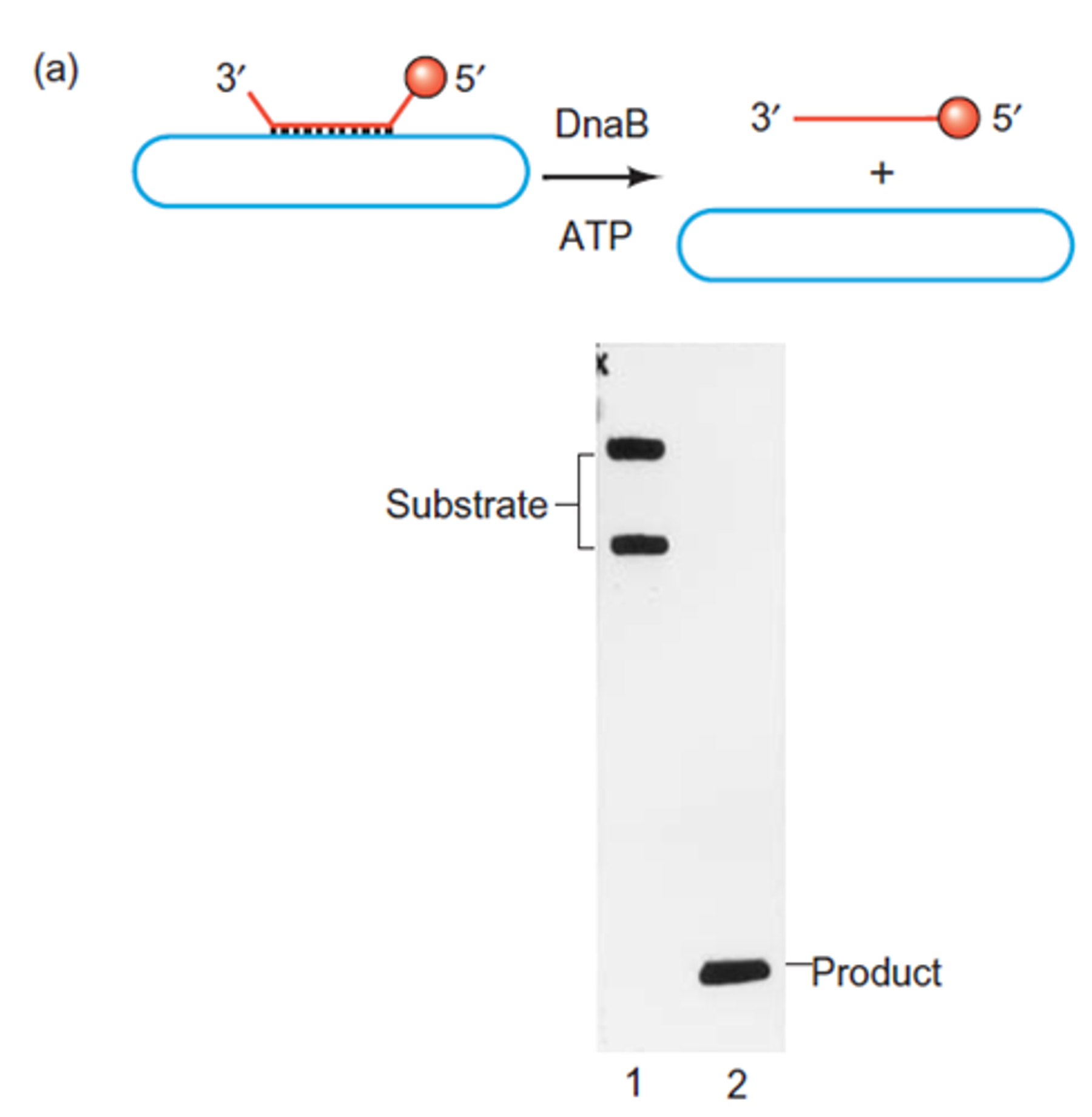
위 그림에 나타나 있는 실험 결과는 DNA helicase가 unzipping 활성을 가진다는 것을 보여줌. 보면 하늘색 ring 구조에 붉은색(labeled) DNA를 hydrid 시켜놓음. 이 상태에서 non-denaturation gel을 사용한 전기영동을 수행한 결과가 1과 같음. 이 때 2개의 band가 나오는 이유는 하늘색 ring 구조가 ring 그 자체로 있을 때, linear하게 풀어졌을 때 gel mobility가 달라지기 때문임. 한편 이 sample에 DnaB, ATP를 처리해준 경우에는 product에 해당하는 band만 나옴. (즉, 빨간색 서열이 따로 분리된 것) 이를 통해서 DNA helicase, 그 중에서도 DnaB가 helicase activity를 가진다는 것을 알 수 있음.
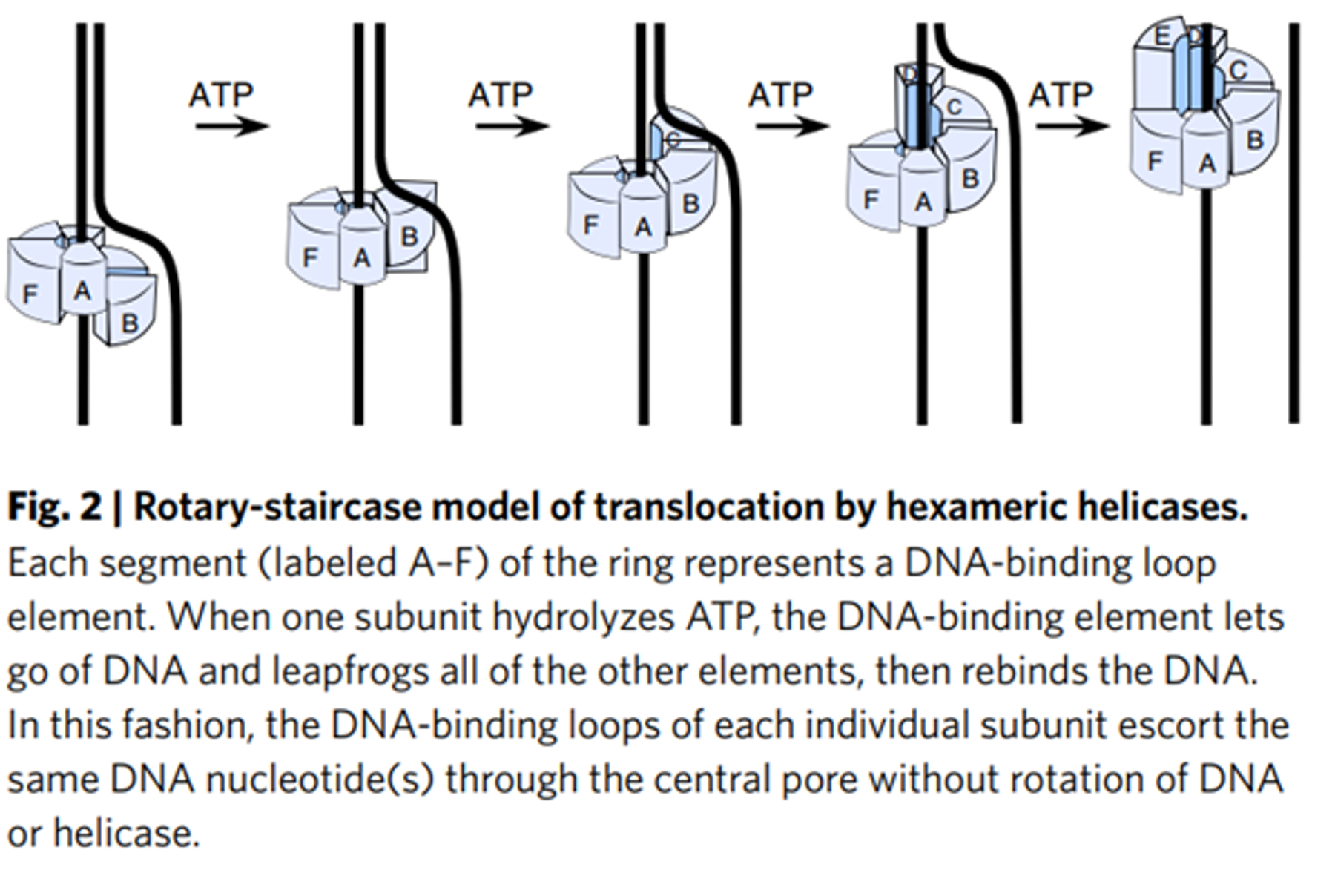
DNA helicase가 DNA를 unzipping시키는 것과 관련해서 많은 작동 model이 제안되어 있는데, 그 중 가장 대표적인 것이 바로 위 그림에 나타나 있는 rotary-staircase model임. 이 model은 helicase가 위 그림과 같이 차곡차곡 ATP를 쓰며 올라간다는 model임. 다만 1개의 ATP를 썼을 때 몇 개의 nucleotide 정도를 unzipping할 수 있는지에 대해서는 아직 의견이 분분함.
그런데 결국 DNA helicase도 닫힌 고리형의 육각구조이므로 DNA strand를 중간에 둔 채로 끼어들어가려면 앞서와 같이 loading 과정이 필요함.
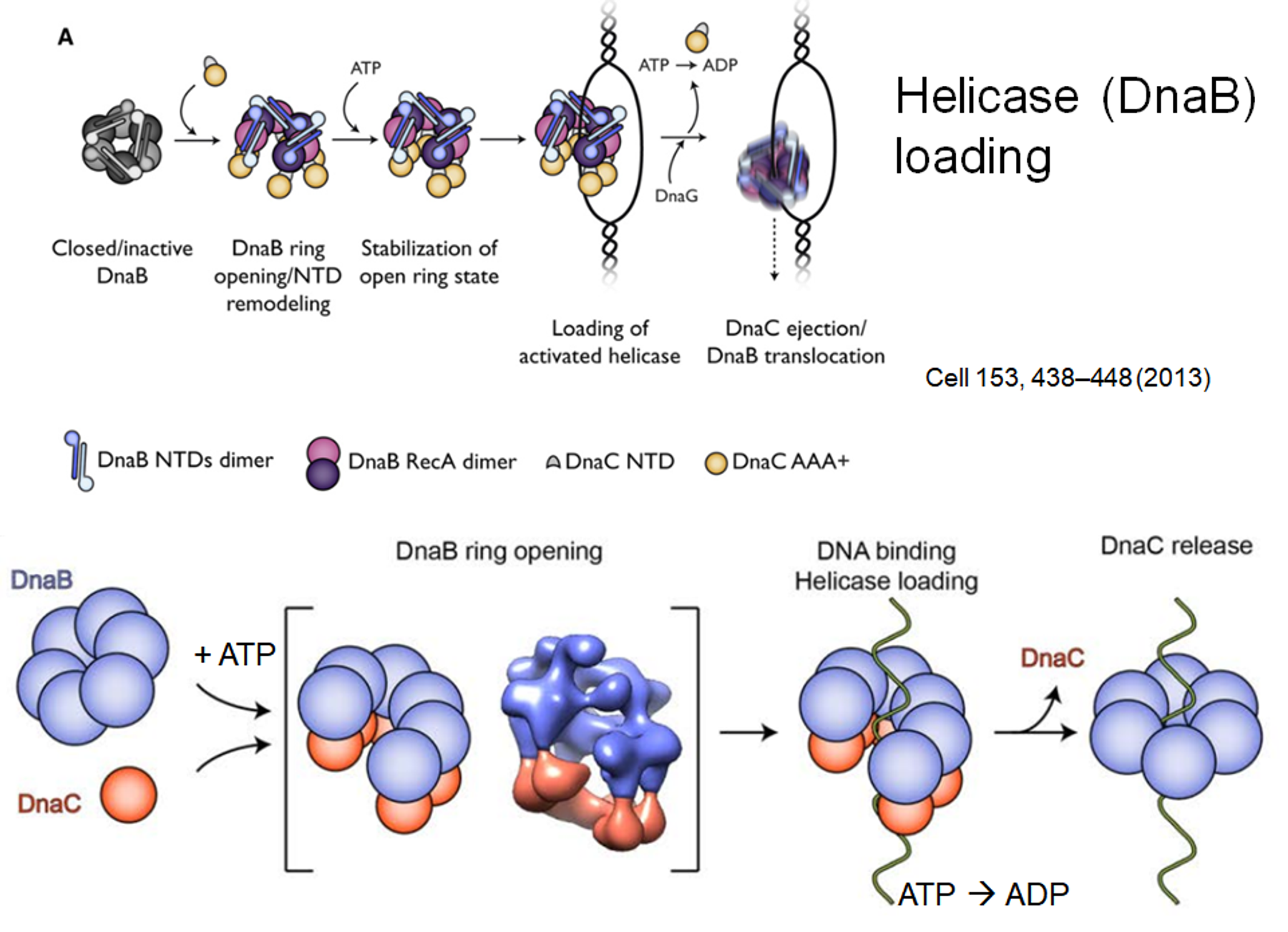
위 그림에서 DnaB가 DNA helicase 활성을 가진 녀석이며, 이 녀석에 DnaC라는, ATPase 활성을 가진 녀석이 붙고 이어서 DnaB의 닫힌 고리를 열어버림. (ring opening) 이어서 open된 고리에 DNA가 binding되고, DnaC가 다시 빠져나가면서 DNA helicase가 중간에 DNA를 물고 있는 채로 닫힌 고리 구조로 돌아가게 됨. 이 과정에서 ATP의 hydrolysis가 일어남. (위 그림 A에서 노란색으로 표현되어 있는 것이 DnaC임. 그리고 DnaC가 나가면서 동시에 들어오는 DnaG라는 녀석도 있는데, 이 녀석 또한 helicase activity를 가짐)
single-strand DNA-binding protein (SSBP)
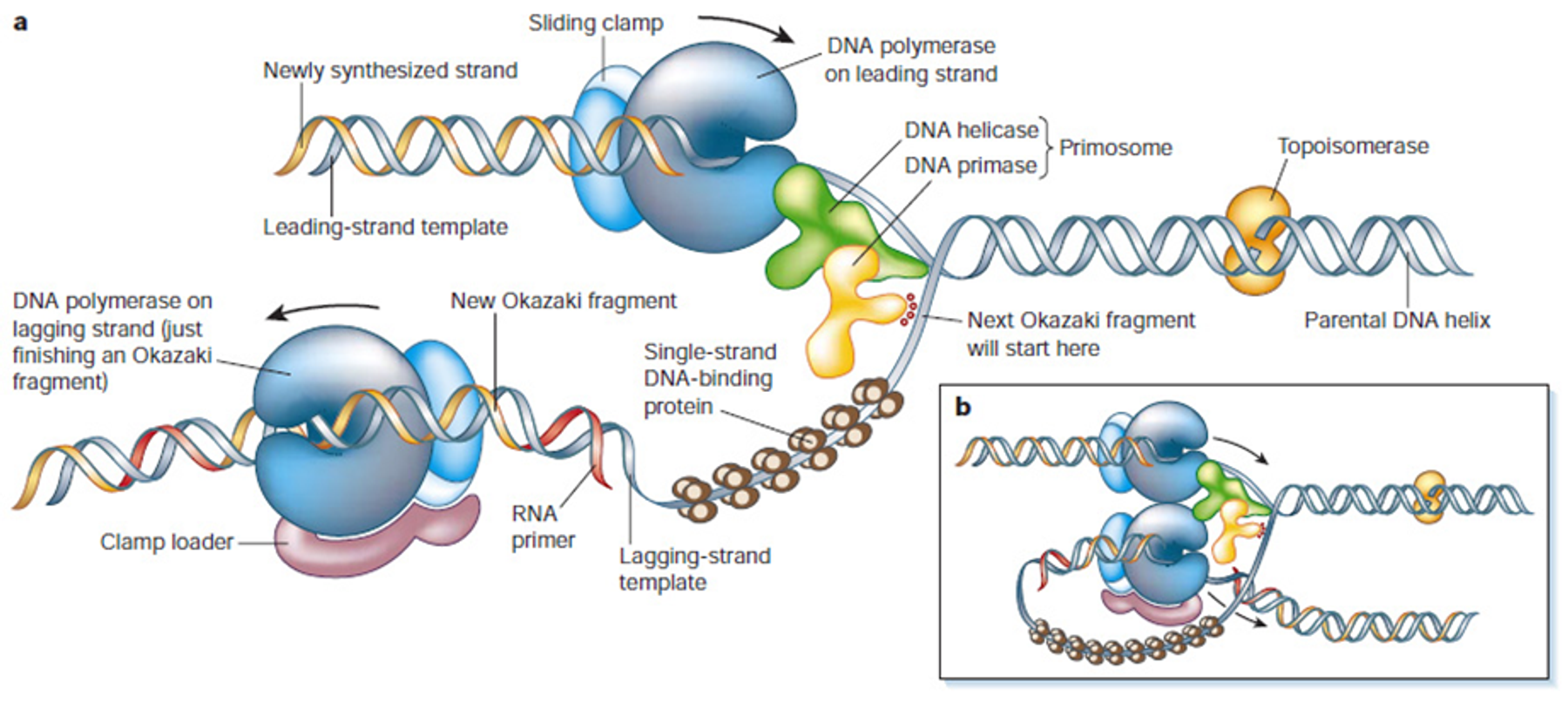
위 그림에서 갈색으로 표현되어 있는 것이 single-strand DNA-binding protein(SSBP)임. 이 녀석은 single strand로 노출되어있는 DNA가 스스로 접혀 secondary structure를 형성하는 것을 방지하고, nuclease의 활성으로부터 coating시켜주는 역할을 수행함.
SSBP는 매우 tight하게 binding하고, 또 cooperative하게 binding함. 즉, 하나가 붙으면 다 붙어버림. (1개 붙으면 나머지의 binding도 다 triggering되어서 줄줄이 다 결합해버림)
그냥 SSBP의 역할만 들어보면 그다지 DNA replication에 필수적이지 않은 것처럼 보임. 그러나 실제로는 SSBP 자체의 역할이 DNA replication에도 매우 중요함.
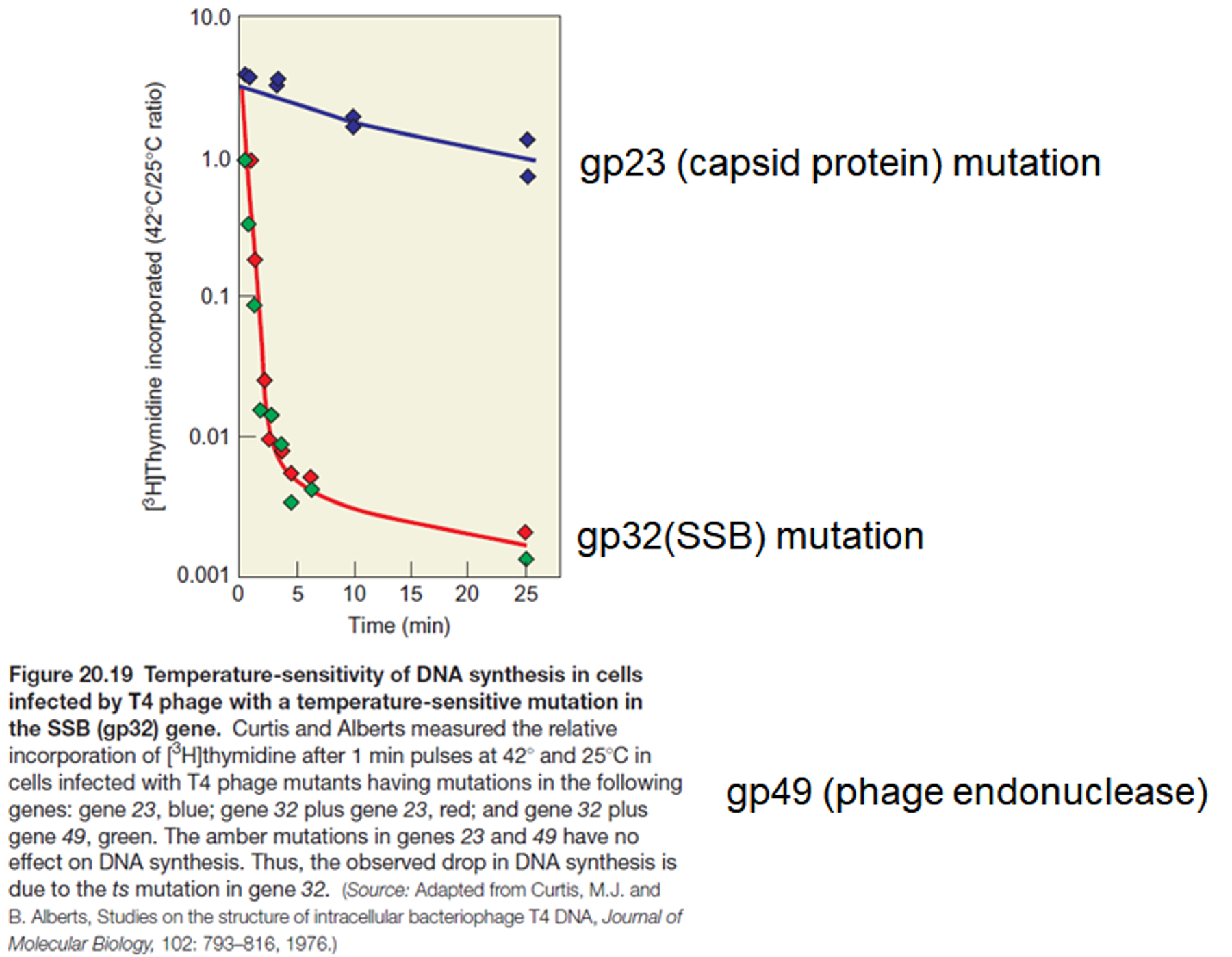
(이 때 capsid protein mutation은 일종의 control로 이해하면 됨)
보면 thymidine incorporation 정도를 확인해보았을 때 SSBP에 mutation이 일어난 경우 이 정도가 훨씬 더 감소한다는 것을 확인 가능함. 이를 통해 SSBP가 적어도 prokaryotic DNA replication에 있어서 중요하다는 것을 알 수 있음.
topoisomerase
topoisomerase는 앞서 말했듯 dsDNA의 supercoil을 풀어주는 역할을 함.
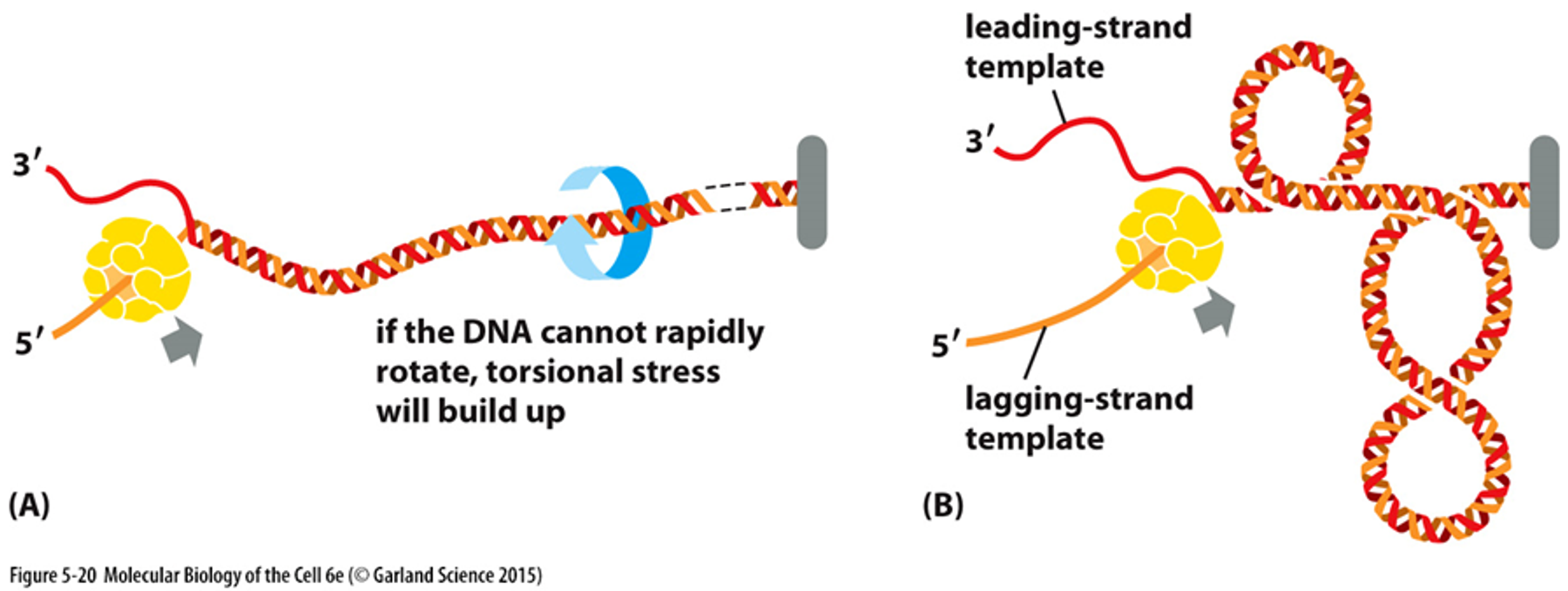
만약 topoisomerase가 없다면 위와 같이 helicase가 DNA strand를 풀어가는 과정에서 그 뒤쪽(진행 앞 방향)의 dsDNA들은 과도하게 꼬이게 될 것임. (bacteria의 경우에는 특히나 DNA가 circular하므로 반대쪽 끝이 고정된 것이나 다름없어서 과꼬임이 발생할 것이고 human의 경우에도 linear한 녀석들의 일부가 scaffold로, 나머지가 scaffold에 달린 loop로 존재하는 경우가 많고 꽤나 길이도 길기 때문에 이 경우에도 반대쪽 끝이 고정되어 있다고 봐도 무방하므로 과꼬임이 발생할 것임)
결국 이를 풀어주지 못한다면 (B)와 같이 supercoiling이 일어나 더이상 helicase가 replication fork를 진행시키지 못할 수도 있음.
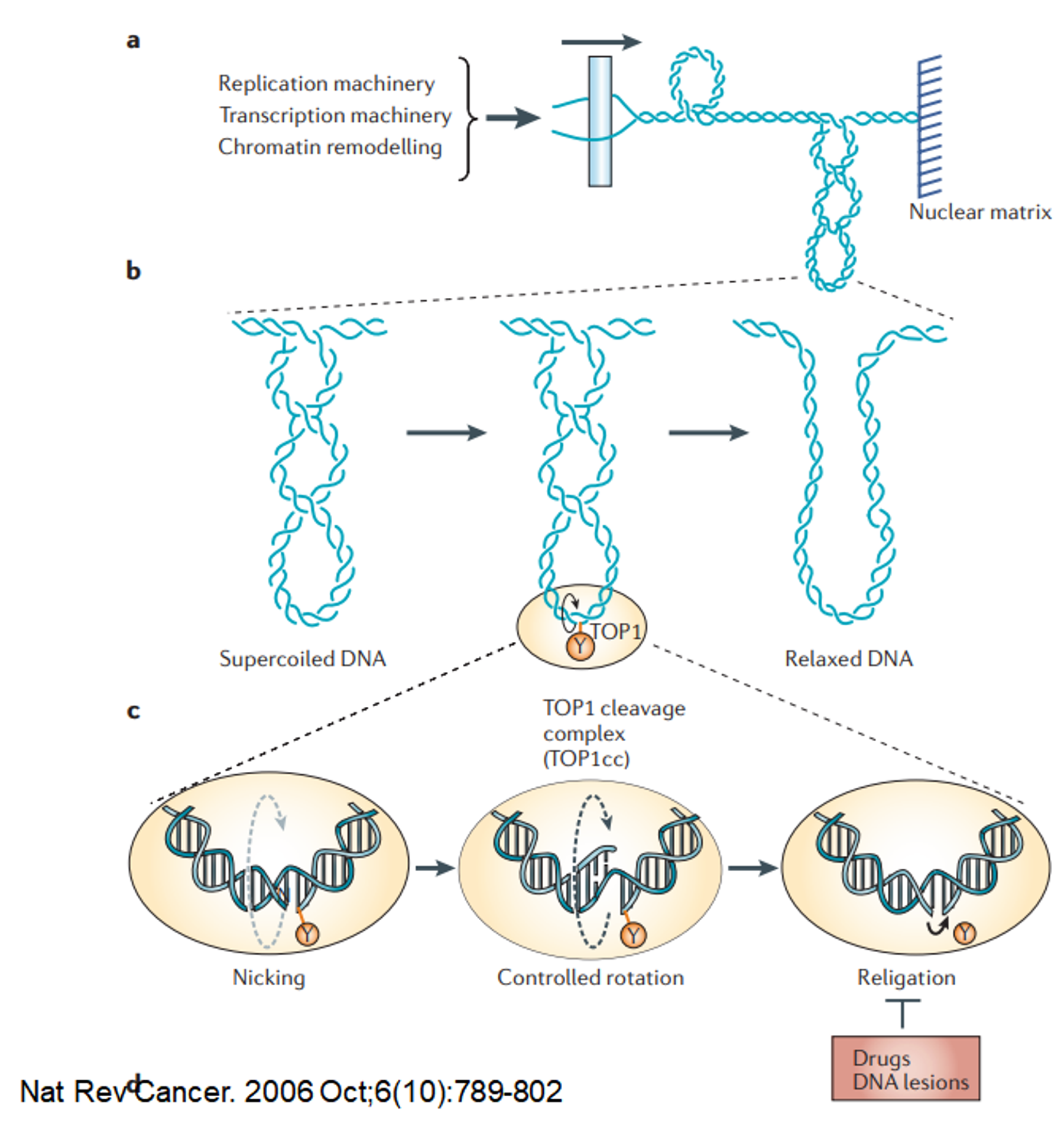
이 때 사용되는 효소가 topoisomerase임. topoisomerase에는 크게 2가지 종류가 있는데, 바로 type 1, type 2임.
우선 위 그림에서는 type 1 topoisomerase의 작동 기작이 나타나 있음. 보면 type 1 topoisomerase는 내부에 가지고 있는 tyrosine의 OH를 이용해서 특정 DNA 부분의 phosphodiester bond를 하나 끊어주게 되고(즉, 두 가닥의 DNA 중 한 가닥만 끊어줌) 이후 끊어진 가닥이 자유롭게 rotating하는 시간을 가진 다음(즉, 자발적으로 relexation하는 과정을 가진 다음) 다시금 phosphodiester bond가 형성됨. 이런 방식으로 torsional stress를 해소하므로 결국 type 1 topoisomerase의 작동에는 에너지가 필요하지 않음.
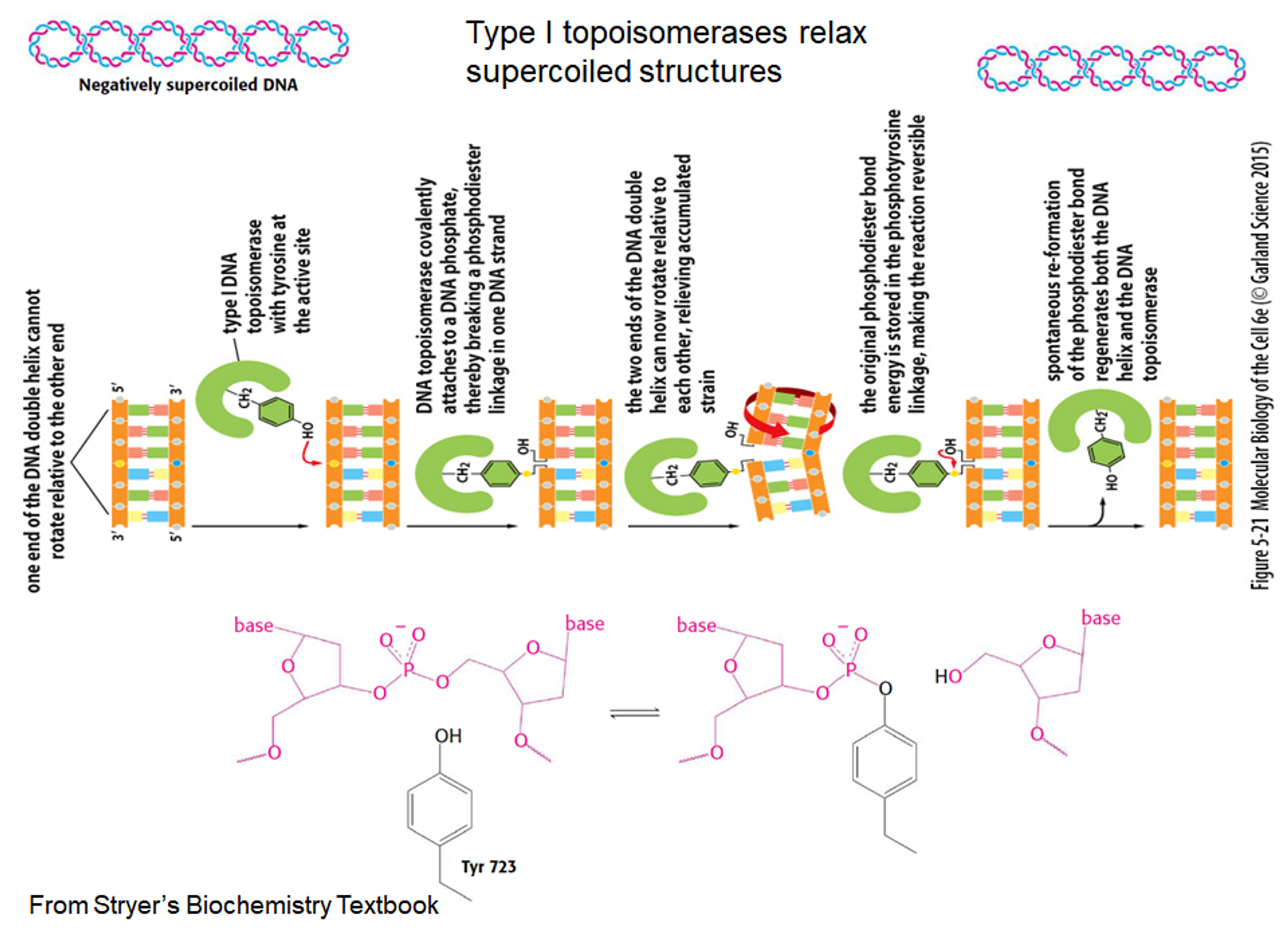
위 그림에는 구체적으로 type 1 topoisomerase의 Tyr 723 내부의 OH가 어떻게 phosphodiester bond를 공격해 끊고, 이후 다시금 rotation된 nucleotide의 OH에 의해서 어떻게 다시 phosphodiester bond가 재형성되는지의 과정이 나타나 있으므로 참고할 것.
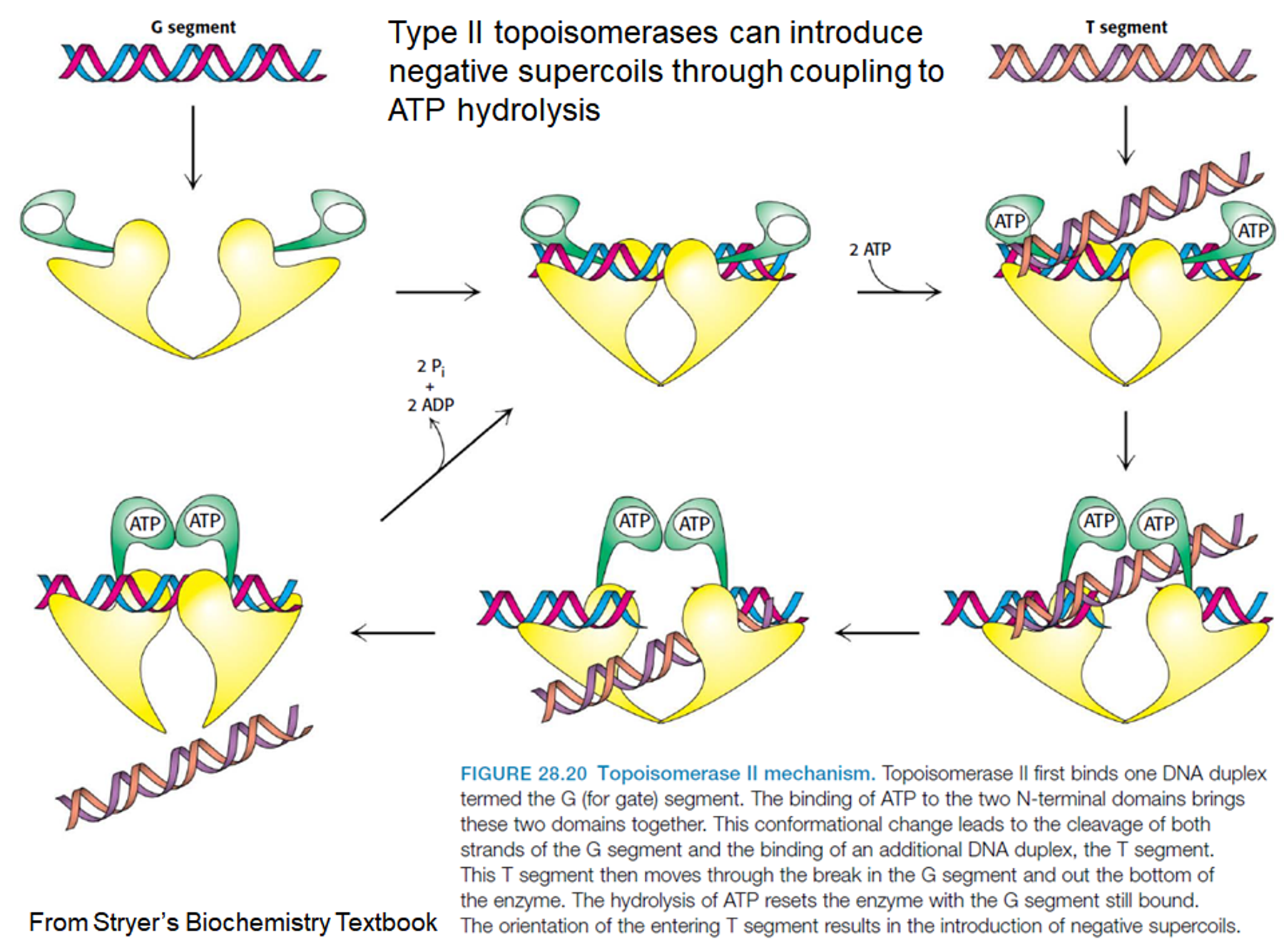
다음으로 type 2 topoisomerase에 대해 알아보자. type 2의 경우 위 그림과 같이 2가닥의 DNA를 다 끊어버림. 그러는 한편 일단 두 가닥을 다 끊은 이후 끊긴 틈 사이로 나머지 연결된 두 가닥을 통과시켜버림. 즉, type 2는 type 1과 달리 dsDNA 가닥 수준에서 supercoil을 완화시키는 것이 아니라 dsDNA 가닥끼리 꼬여 형성되는 supercoil 자체를 relexation시키는 역할을 수행함. 참고로 이 때 끊어진 DNA 가닥 사이로 나머지 dsDNA를 통과시키는 과정에서 ATP의 loading이 필요하며, 한편 끊어진 DNA끼리의 재결합 과정에서는 ATP의 가수분해또한 필요함.
type 1에서는 외부 energy가 필요하지 않고 type 2에서는 외부 energy가 필요하다는 사실을 꼭 기억해두자.
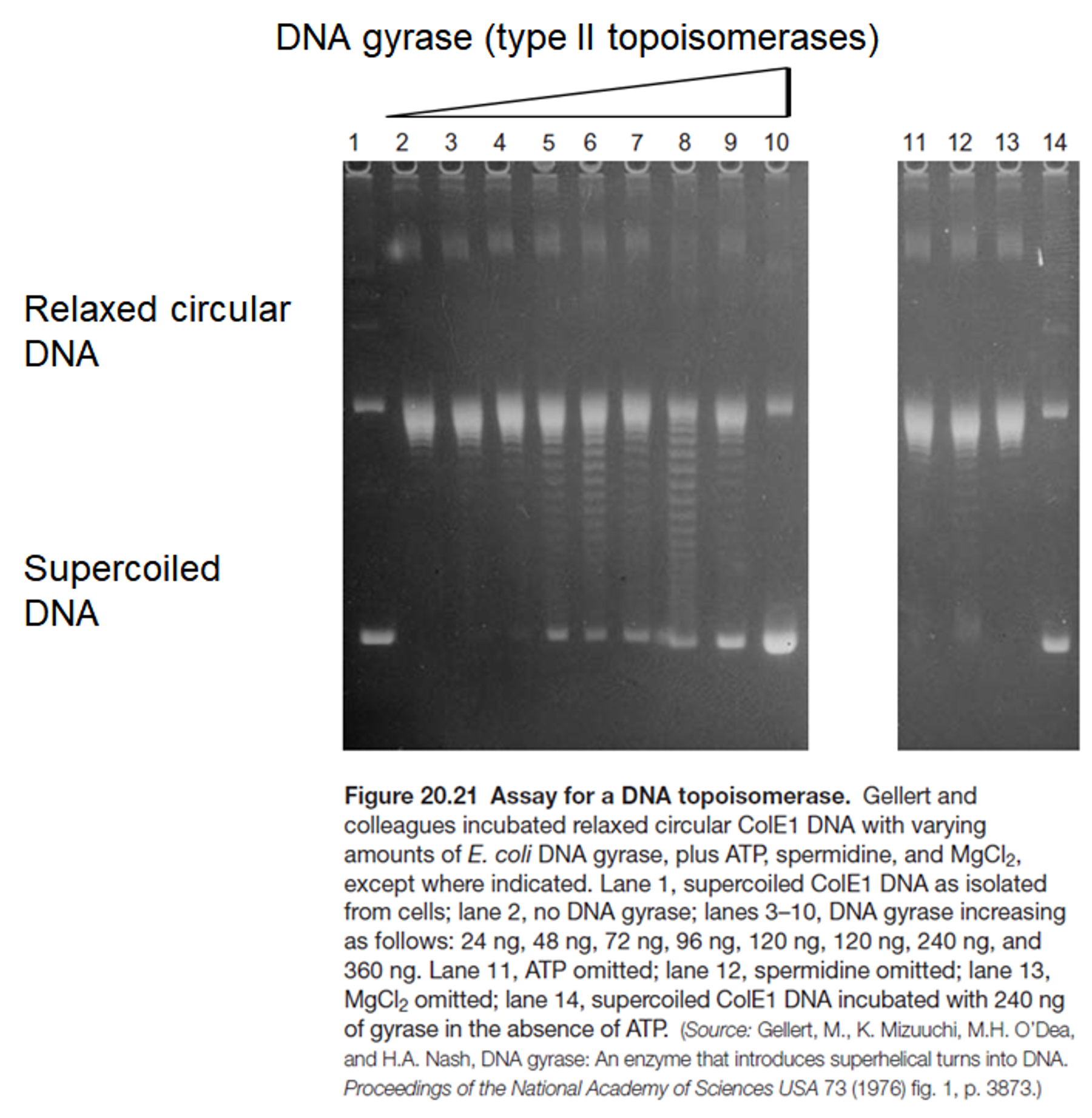
위 그림에는 실제로 DNA gyrase(type II topoisomerase)의 활성을 나타내주는 실험 결과가 나타나 있음. 이 때 relaxed circular DNA는 그냥 원형, supercoiled DNA는 꼬인 원형이며, supercoiled DNA가 더 compact하므로 잘 가라앉음.
(참고로 세균 내에 plasmid가 그냥 DNA보다 더 compact하므로 supercoil 효과에 의해 더 침전이 잘 됨. 이를 이용하면 plasmid와 bacterial DNA를 분리해내는 것이 가능하기도 함)
한편 lane 2~10까지를 비교해보면 DNA gyrase를 많이 넣어주면 넣어줄수록 relaxed circular DNA였던 녀석들이 끊기고 교차되어 다시 연결되는 과정을 거치면서 결국 supercoiled DNA로 변하게 되어서 결과적으로 lane 10을 보면 band가 supercoiled DNA로 거의 다 넘어간 것을 알 수 있음. 그리고 lane 11~14를 보면 결국 조효소로 작용하는 Mg2+, 혹은 기질로 작용하는 ATP가 없으면 gyrase activity가 발생하지 않는다는 것 또한 알 수 있음.
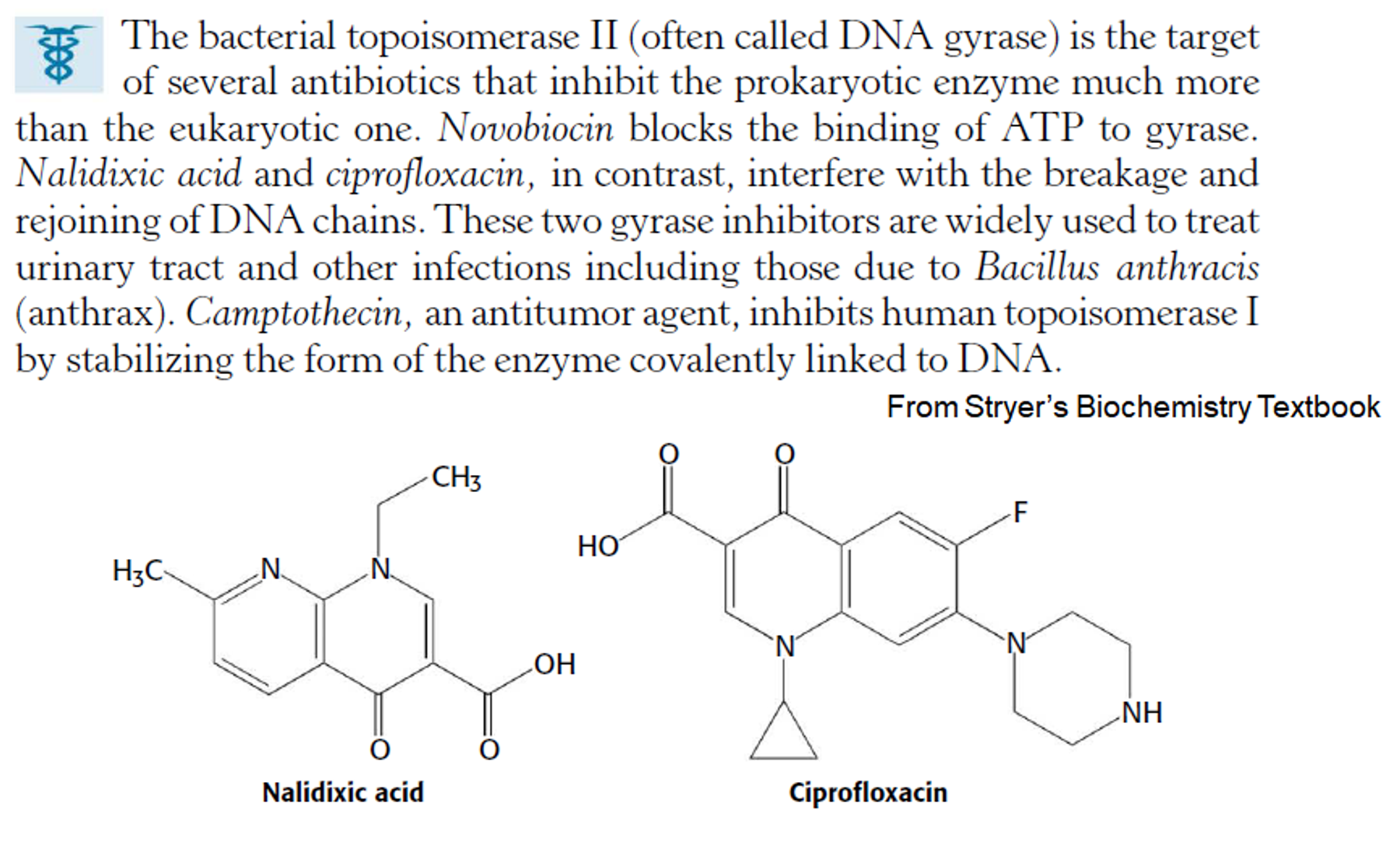
한편 topoisomerase를 inhibition시키는 inhibitor를 항생제나 항암제로 사용할 수 있음.
예를 들어 bacterial topoisomerase II(DNA gyrase)를 inhibit하는 Novobiocin이라는 녀석은 bacteria로 하여금 stress 누적으로 인해 분열을 제대로 안 되게 만드는 효과가 있어서 항생제로 사용 가능함. 한편 Ciprofloxacin은 human topoisomerase I의 경우 topoisomerase의 Y에 의한 phosphodiester bond 공격에 이어 DNA 가닥 하나가 끊긴 상태를 매우 안정화시켜버려서 결과적으로 끊겨있는 상태로 DNA를 유지시켜버림. 이 녀석은 특히 세포분열을 활발히 하는 암 세포에 치명적으로 작동하므로 anti-tumor agent로 쓰일 수 있음. (그 밖에 아예 한 쪽이 끊긴 형태의 DNA를 실험실 level에서 만들고자 할 때도 많이 사용함)
다음 포스트부터는 이제 DNA replication의 개시와 종결 과정에 대해 자세히 알아보자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 20.1 : DNA 복제 개시 - 2 (0) | 2024.10.27 |
|---|---|
| [분자생물학] 20.1 : DNA 복제 개시 - 1 (0) | 2024.10.27 |
| [분자생물학] 19.2 : enzymology of DNA replication - 1 (0) | 2024.08.10 |
| [분자생물학] 19.1 : DNA replication(DNA 복제)의 특징 (1) | 2024.08.10 |
| [분자생물학] 18.3 : 번역 종결(translation termination) - 3 (0) | 2024.08.10 |