지난 포스트에서는 진핵생물의 RNA polymerase II를 구성하는 다양한 Rpb들에 대해 알아봤었음.
넘어가기 전에 in vitro상에서 나타나는 Rpb1 band의 독특한 특징에 대해 살펴보고 넘어가자.

위 그림에 나타난 실험결과는 mouse plasmacytoma의 RNA polymerase II 전기영동 결과임. 이 때 c에 해당하는 band가 Rpb2임. 그런데 Rpb2보다 더 위쪽에 (원래는 Rpb1 band 하나만 있어야 할 곳에) o, a, b의 3개의 band가 나타나있음을 확인할 수 있음. 즉, Rpb1에 대해 heterogeneity가 관찰되는 것임.
즉, 3종류의 Rpb1이 존재하는 것인데, 이 각 종류의 Rpb1을 함유하고 있는 polymerase II를 각각 IIo, IIa, IIb로 명명함.

이 때 이들 각각은 위 그림과 같은 관계를 가짐. 우선 IIb부터 살펴보자. 이 녀석은 셋 중 가장 size가 작음. 이 녀석은 다른 녀석들과 비교해 보았을 때 CTD(carboxyl-terminal domain)라는 부분이 결실되어 있음. CTD는 원래 Rpb1 안에 포함된 domain으로 7개의 peptide, 즉 heptide(TSPTSPS)가 계속 반복되어 있는 형태임. 이 녀석은 peptide core 바깥으로 꼬리처럼 튀어나와있음. 따라서 이 CTD 부위가 상대적으로 protease에 의해 잘리기 쉽게 되고, 결과적으로 IIa, IIo에 protease가 작용해 IIb가 만들어지는 것임. 이 IIb는 사실 in vitro에서만 만들어지고, in vivo에서는 이런 protease의 작용이 block되어 있어 IIb가 만들어지지 않음.
한편 위 그림상에 나타난 IIa, IIo는 실제로 세포 내에서도 생성될 수 있는 두 개의 형태인데, 이들의 분자량을 비교해보면 IIa가 일반적으로 gene 발현에 의해 만들어지는 단백질이 가질 수 있는 분자량임. 이 때 IIa에 kinase가 작용해 phosphorylation을 시켜주면 조금 더 무거워진 IIo가 만들어지는 것임. (참고로 이 때 kinase는 S, 그 중에서도 주로 TSPTSPS 중 앞쪽 2개의 S를 인산화함. 물론 뒤쪽 S도 간혹 인산화하기는 함) 물론 IIo에 phosphatase가 작용해 탈인산화가 일어나면 다시 IIa가 형성됨. 이 때 IIa(RNA polymerase IIA)는 promoter와 binding하는 역할을 수행하고, IIo(RNA polymerase IIO)는 transcript elongation에 관여함. 즉, 인산화 여부에 따라 IIa, IIo로의 전환이 일어나고, 이들 각각이 서로 다른 역할을 수행함.
RNA polymerase II의 입체구조를 결정하는 데 가장 큰 기여를 한 연구자는 Roger Kornberg라 말해도 무방할 것임. 지금부터 살펴볼 대부분의 RNA polymerase II 구조 결정 과정은 거의 대부분 그에 의해 수행된 것임. (그는 이에 대한 업적을 인정받아 2006년 노벨 화학상을 수상함)
일반적으로 특정 분자의 구조를 파악할 때는 x-ray crystallography가 매우 유용하게 사용됨. (x-ray crystallography를 이용하면 atom들의 배열을 하나하나 coordinate할 수 있음) 그런데 x-ray crystallography를 수행하기 위해서는 우선 특정 물질을 crystal(결정) 형태로 만들어야 함. 이 때 결정을 얻는 것이 힘들어서 x-ray crystallography 결과를 얻지 못하는 경우가 매우 많음.
RNA polymerase II도 초기에는 결정을 얻기 쉽지 않았음. 이에 대한 이유에는 여러 가지가 있을 수 있지만, 일단 기본적으로 모든 단백질분자들이 다 identical해야 서로서로 잘 포개져 결정이 잘 만들어짐. 그런데 RNA polymerase II의 경우 (당시 이유는 몰랐지만) Rpb 4, 7이 어떨 때는 붙어있고 어떨 때는 떨어져 있어서 RNA polymerase II의 모양이 때에 따라 계속 달라짐. 이 때문에 RNA polymerase II끼리 규칙적으로 쌓이기가 어려워서 결정이 잘 만들어지지 않았던 것임.
따라서 일단 임시방편으로 RNA polymerase II의 일부 구조만이라도 결정하기 위하여 Roger Kornberg 및 그의 연구팀은 Rpb4, 7이 발현되지 않도록 만들어준 Delta 4/7 mutant의 RNA polymerase II를 만들고, 이를 이용해 결정화를 시도했음. 그랬더니 비교적 결정이 쉽게 얻어짐. 이를 이용해 x-ray crystallography를 수행한 결과 아래와 같은 입체구조를 얻어냄.
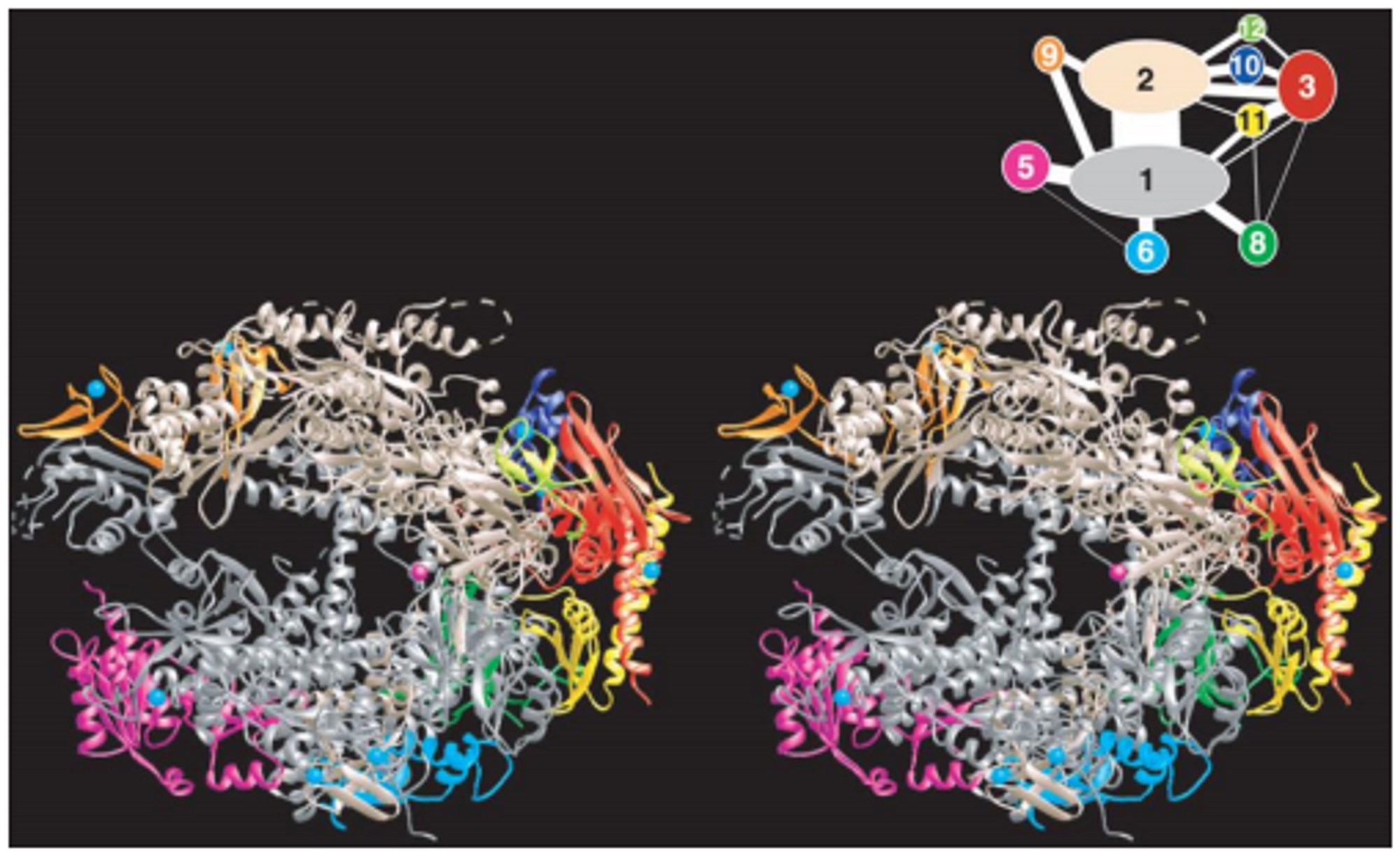
보면 RNA polymerase II(Rpb 4, 7 제외)의 모양이 마치 집게 모양으로 생겼다는 것을 알 수 있음. 이 집게의 안쪽에 DNA를 둔 채로 RNA 중합을 진행해나가는 것임.
참고로 이 때 바깥쪽을 향하고 있는 아미노산들은 대부분 - charge를 띄고 있음. 한편 안쪽을 향하고 있는 아미노산들은 대부분 + charge를 띄고 있음. 이는 backbone 때문에 음전하를 띄는 DNA, RNA를 집게 안쪽에 정전기적으로 잘 붙잡고 있기 위한 의도적인 전하배치임.
한편 위 그림상에서 보라색 작은 구슬로 표현된 것은 Mg2+ (혹은 Mn2+)를, 파란색 작은 구슬로 표현된 것은 Zn2+를 나타냄. 이 때 집게의 가운데 부분에 Mg2+가 존재하는 부분이 바로 catalytic activity를 띄는 catalytic center임. 또한 이 때의 Mg2+ ion을 metal A라 칭함.
한편 metal A 이외에 또 다른 Mg2+도 존재할 수 있는데, 이를 metal B라 칭함. metal A는 항상 원래 위치에 붙어있는데 반해 metal B의 경우 농도를 측정해 볼 시 metal A보다 훨씬 낮게 측정되고, 어떤 RNA polymerase II의 metal site에는 있으나 다른 곳에는 없는 경우가 많이 관찰됨. 이는 metal B가 substrate nucleotide가 들어옴과 동시에 같이 딸려들어오기 때문임.
여기서 그치지 않고 구조생물학자들은 elongation complex를 이루고 있을 때의 RNA polymerase II 구조를 관찰하고자 했음. 이를 위해서는 RNA polymerase II와 함께 DNA를 넣어줘야 했는데, 실제로 전사가 일어날 수 있는 DNA template를 선별해서 넣어줌. 그런데 이 때 넣어준 DNA 가닥은 전체가 이중가닥으로 이루어져있지 않고, 일부 가닥은 이중가닥이지만 그 뒤쪽 부분에는 단일가닥으로 존재하도록 가공된 녀석임. 이 때 단일가닥 부분을 RNA polymerase II가 인식하고 RNA 합성을 시작할 것임.
한편 우리가 관찰하고자 하는 것은 DNA template로부터 RNA를 완전히 합성한 이후의 RNA polymerase II가 아니라 RNA를 합성하는 도중의 RNA polymerase II이므로, RNA polymrease II의 RNA 중합을 도중에 stop시켜놓은 채 결정을 만들어야 함. 이를 위해 기질로 넣어준 DNA template의 단일 가닥을 C만으로 이루어져 있게 해 둔 후, 용액에 GTP를 넣어줘서 C에 상보적인 G가닥의 RNA가 만들어지도록 해 줌. 그러다가 C가 아닌 가닥을 만나게 되면 RNA polymerase II는 GTP 이외에 nucleotide source가 없으므로 더이상 RNA를 합성하지 못하고 전사가 중단된 채로 남아있게 됨. (즉, CCCCCCCCT로 해놓으면 GGGGGGGG까지만 RNA가 만들어지고 멈춰있게 됨) 이 때의 RNA polymerase II를 결정화하고 x-ray crystallography를 통해 얻은 구조가 아래와 같음.
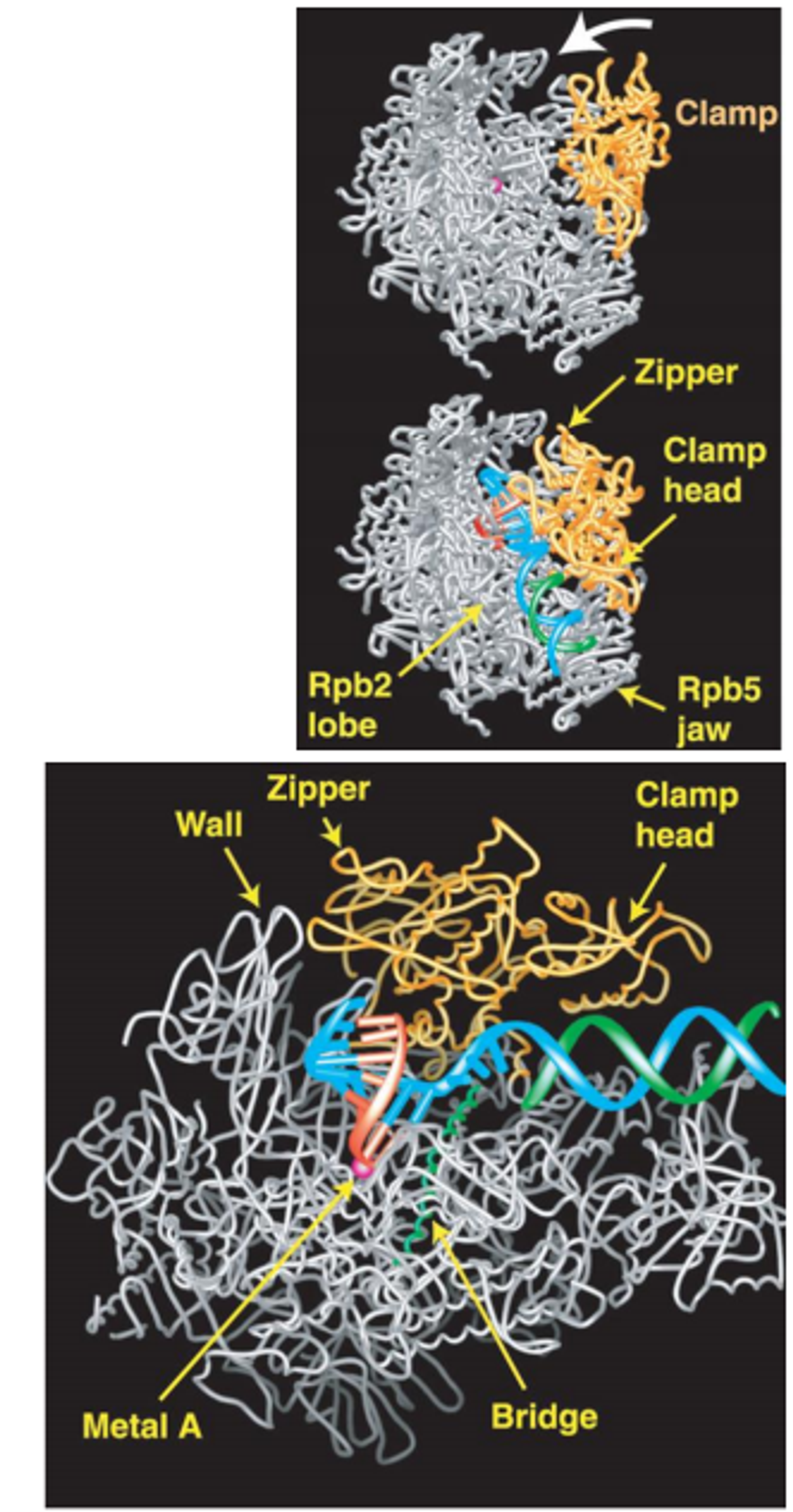
사실 이 clamp에 의해 RNA polymerase II가 독특하게 가지는 특성 중 하나인 processivity가 부여되는데, processivity는 RNA polymerase II가 한번 작동하기 시작하면 도중에 떨어져나가지 않고 끝까지 붙어 쭉 전사를 진행하는 성질을 말함. 즉, clamp가 DNA template를 꽉 붙들어주고 있기 때문에 RNA polymerase II가 도중에 빠져나가지 않고 계속 template을 따라 전사를 진행할 수 있는 것임.
한편 위 그림은 입체적이고 매우 정확한 단백질의 구조를 나타내주고 있기는 하지만 특징을 살펴보기 다소 어려움. 따라서 조금 더 알아보기 쉬운 모식도를 살펴보자.
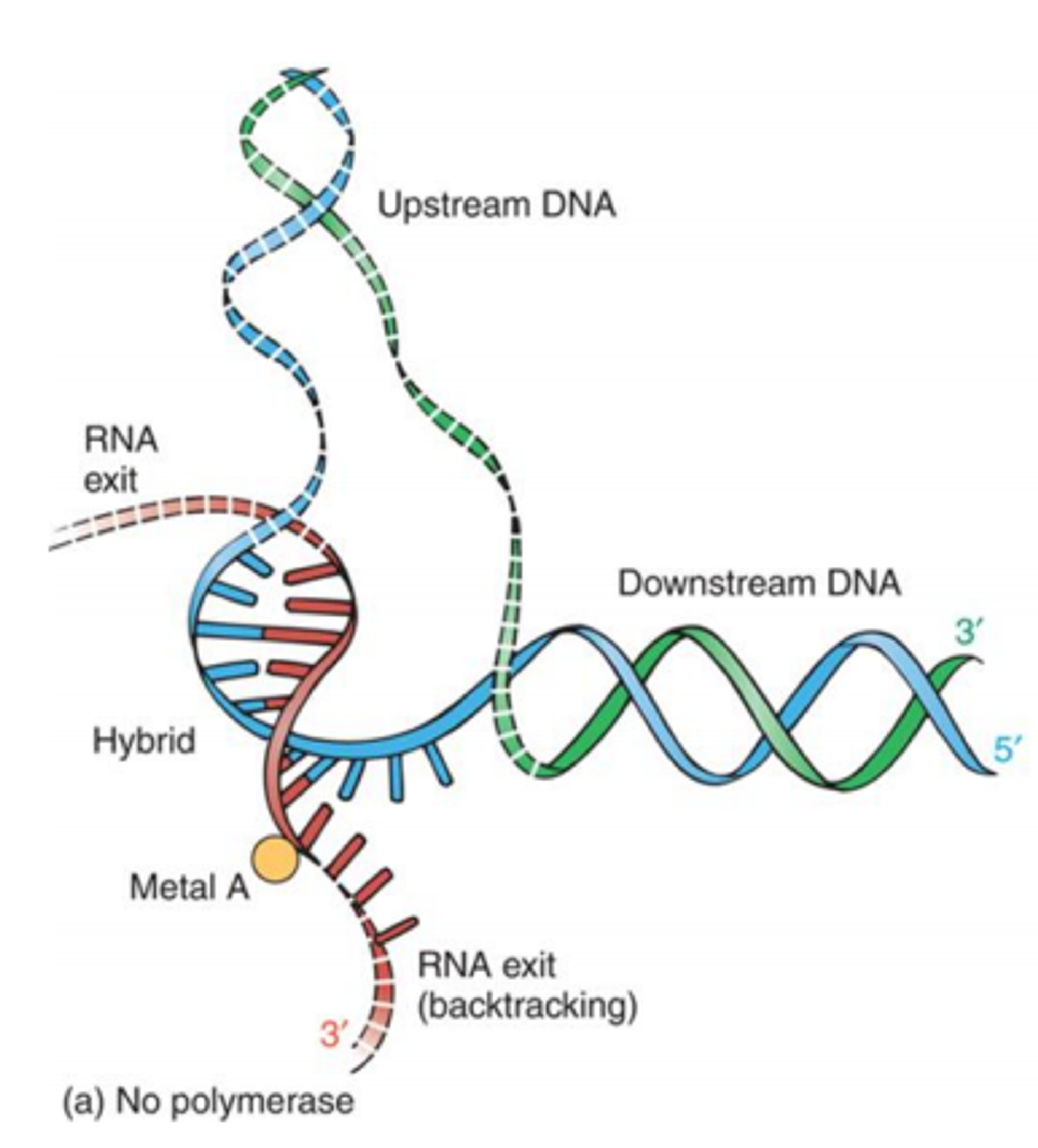
위 그림에는 RNA polymerase II의 element들이 묘사되어 있지 않으며 단지 template DNA(blue), nontemplate DNA(green), RNA(red)가 나타나 있음. 참고로 이 때 점선으로 표시된 DNA 서열들은 in vivo에서는 존재하는 서열이나, 앞서와 같이 우리가 인공적으로 단일 서열로 3' 부분을 만들어 넣어준 DNA에서는 존재하지 않는 부분임. (in vivo에서는 원래 이중가닥이던 DNA를 open한 후 드러난 단일가닥 DNA를 바탕으로 전사를 진행함) 이 때 노란색으로 표현된 metal A가 있는 site가 catalytic site임.

위 그림은 앞서의 그림에 더해 RNA polymerase II의 element들도 같이 표현한 것임. 이때 보면 DNA와 RNA가 RNA polymerase II와 가장 잘 반응하도록 optimize시켜주는 도우미 단백질들이 존재함. 위 그림에는 Zipper, Lid, Rudder가 나타나 있음. Lid의 경우 DNA와 RNA가 서로 분리된 채 존재하도록 도와주고, Rudder도 마찬가지로 DNA와 RNA가 서로 분리된 채 존재하도록 도와주며(Rudder가 DNA, RNA 결합이 분리된 채 존재하는 initiation point를 지정) Zipper는 template DNA와 non-template DNA가 다시 결합하는 것을 방지해줌.
한편 RNA polymerase의 구조를 자세히 들여다보면 위 그림과 같이 Pore 1과 Funnel에 의해 형성된 구조를 관찰할 수 있음. 이 때 이 Funnel을 통과해 Pore 1으로 새로운 nucleotide가 들어오며, 이 Pore 1 구조가 잘 design되어 있기에 Pore 1을 통해 RNA polymerase의 backtracking도 가능함. (backtracking은 mismatch repair를 하고자 할 때 일어남)
한편 위 그림에 그냥 둥그런 모양으로 표현된 Bridge helix는 사실 꽤나 중요한 역할을 할 것으로 추정됨. 바로 translocation임.

bridge helix는 위 그림 (b)에 나타난 것처럼 두 가지 상태로 존재할 수 있음. green은 bent state이고 orange는 straight state임. 이 때 결과적으로 이 녀석이 두 state를 번갈아가면서 가운데 부분의 구조가 변하여 (a)에서와 같은 translocation을 유발하지 않을까 예상되고 있음.
translocation의 mechanism을 조금 더 자세히 살펴보자. (a)를 보면 metal A가 있는 catalytic site에 의해 NTP가 새로 들어와 중합이 진행됨. 그런데 하나가 중합되고 나면 metal A의 위치와 새로 중합되어야 할 자리의 위치가 동일하지 않음. 따라서 translocation이 필요함. (이 때 RNA polymerase II 자체가 이동할 수 없으므로 서열이 이동해야 함) 이 때 bridge helix의 state변화가 translocation을 유발할 것으로 생각되고 있음. (a)를 보면 orange에서 green으로 bridge helix가 변화하며 metal A가 다시금 새로운 RNA 합성 site로 이동했음을 알 수 있음.
다음 포스트에서는 RNA polymerase II의 구조에 대해 이어서 살펴보도록 하자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 10.3 : 진핵생물의 promoter - 1 (0) | 2023.12.16 |
|---|---|
| [분자생물학] 10.2 : RNA polymerase II의 구조 - 2 (0) | 2023.12.16 |
| [분자생물학] 10.1 : 진핵생물의 RNA polymerase - 2 (0) | 2023.12.16 |
| [분자생물학] 10.1 : 진핵생물의 RNA polymerase - 1 (0) | 2023.12.16 |
| [분자생물학] 9.2 : prokaryote에서의 원거리 DNA, 단백질 상호작용 (0) | 2023.12.16 |