이번 포스트부터는 진핵생물에서 일어나는 번역 개시(translation initiation) 과정에 대해 알아보자.
eukaryote에서의 translation initiation은 bacterial translation initiation과 꽤나 차이가 있음. 대표적인 차이를 나열하자면 1) N-formyl-methionine 대신 그냥 methionine이 충전된 tRNA가 처음에 recruit되고 2) shine-Dalgarno sequence 대신 Kozak sequence가 있으며 3) mRNA의 5' end에 cap이 있어 initiation에 핵심적으로 작용한다는 것 등임.

그런데 1)에 나타난 특성에도 불구하고 initiating tRNA와 interior tRNA는 서로 다른 특성을 가짐. 위 그림의 왼쪽에 yeast의 initiator Met tRNA의 구조가, 오른쪽에 yeast의 elongator Met tRNA의 구조가 나타나 있음.
이들 둘 사이에는 미묘한 차이가 존재함. 그런데 이 차이가 몇몇 factor들과의 binding affinity를 dramatic하게 바꿀 수 있음. 예를 들어 initiation factor의 일종인 eIF2-GTP의 경우 elongator tRNA보다 initiator tRNA에 훨씬 더 tight하게 붙음. 한편 elongation factor의 일종인 eEF1A의 경우 initiator tRNA보다 elongator tRNA에 훨씬 더 tight하게 붙음.

진핵세포 내에서는 우선 40S ribosomal subunit이 5'-cap에 binding한 후 5' 끝부분부터 위 그림과 같이 3'쪽으로 쭉 scanning해나가다가 AUG를 만나게 되면 그곳부터 번역이 시작되게 됨. 이를 scanning model이라고 함. (참고로 이 때 40S ribosomal subunit이 잘 binding하기 위해서는 다양한 factor들, Met-tRNA, GTP 등이 필요함)
그런데 이 때 40S subunit scan 과정에서 그냥 아무런 AUG를 만나기만 하면 무조건 그곳으로부터 활발히 번역되는 것이 아니라 다른 AUG에 비해 선호되는 AUG가 존재함. 이 선호도를 부여하는 rule이 바로 Kozak's rule임. AUG의 A를 +1로 뒀을 때 -3 위치에 purine 염기가 오고, +4 위치에 G가 오는 것이 바로 kozak's rule에 의해 가장 favorable한 AUG를 만들어주는 서열 조합임. 예를 들어 AXXAUGG or GXXAUGG 등의 서열을 만들 시 이 sequence 내의 AUG에서 번역이 시작되기에 favorable한 환경이 갖춰져있다고 볼 수 있음. (보통 진핵생물에서 특정 단백질을 overexpression 시키고자 할 때는 ACCAUGG or GCCAUGG 조합의 sequence를 많이 사용함)
그렇다면 Kozak's rule은 어떻게 발견된 것일까.
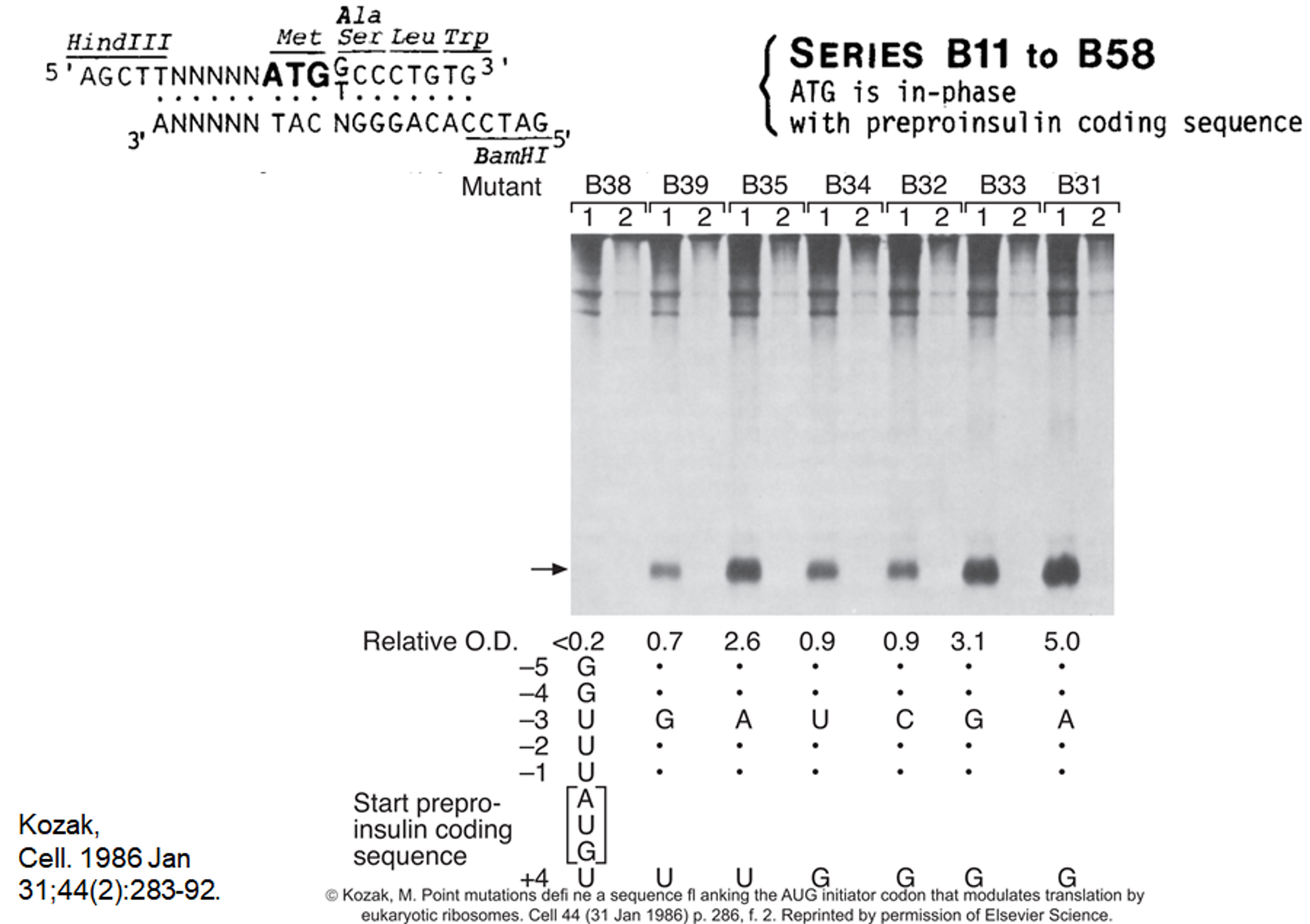
위에 이와 관련된 실험 결과가 나타나 있음. 우선 기본적으로 사용한 sequence는 위 그림 왼쪽에 나타나 있고, 이 때 발현되는 mRNA 부위가 preproinsulin을 coding하고 있으므로 -3부위, +4 부위를 위 그림 아래와 같이 바꿔가면서 preproinsulin의 발현양을 알아본 실험 결과가 위와 같음.
보면 나머지 서열은 같은 상태에서 -3, +4 부위만 다른 것들로 바꿔본 결과 -3자리에 G 또는 A, +4 자리에 G가 있을 때 가장 preproinsulin 발현이 잘 일어난다는 것을 알 수 있음.
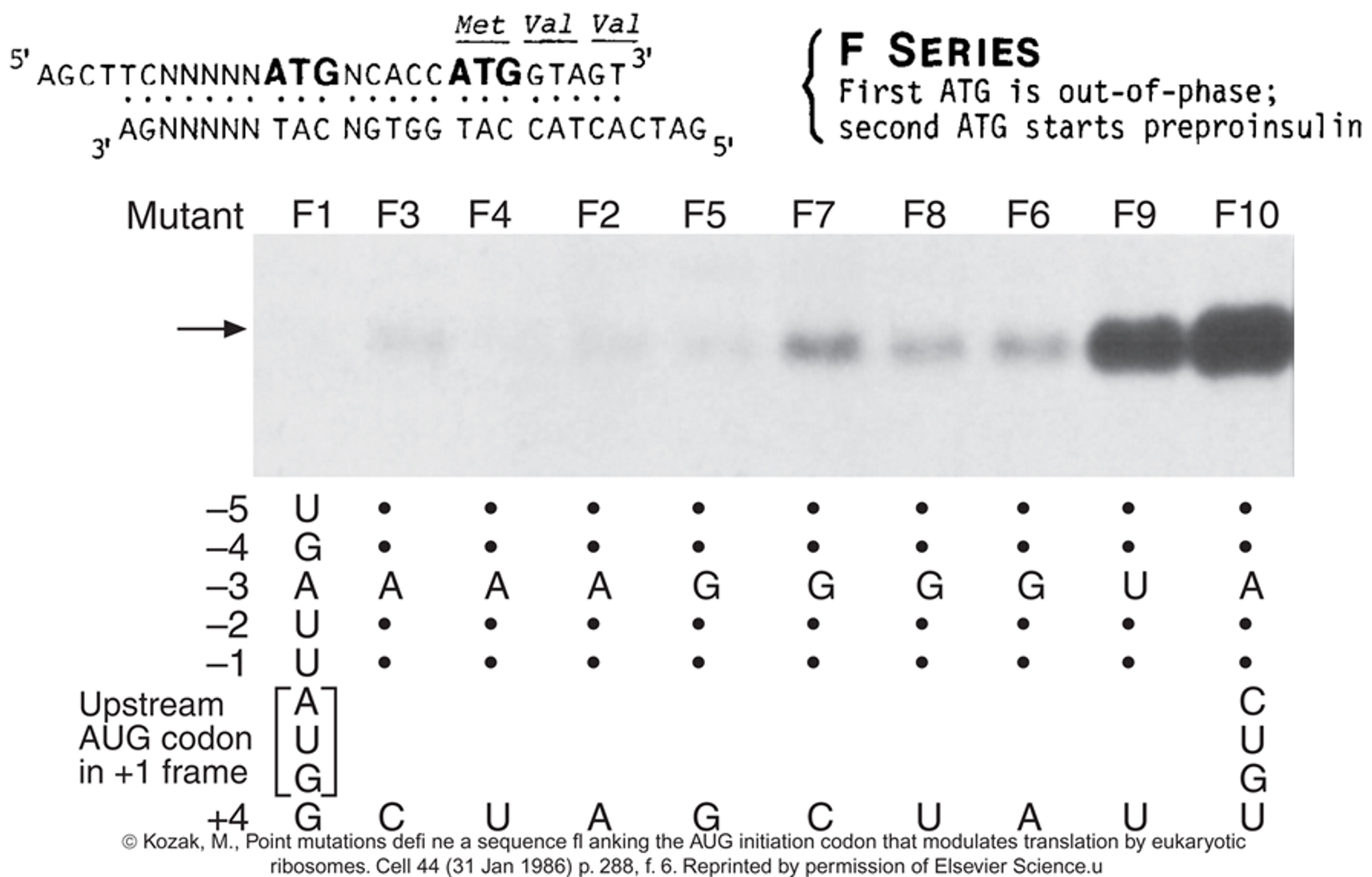
위 그림에 나타난 실험은 앞서와 유사한 실험임. 일단 이 실험에서 사용한 서열은 위 그림 왼쪽 위에 나타난 것과 같으며, 이번에는 ATG가 2개임. (이 두 ATG는 서로 3 codon frame에 맞지 않게, 즉 어긋지게 넣어줌) 이 중 뒤의 ATG에 의해서 preproinsulin이 발현되며, 앞의 ATG 부터 전사가 시작되면 조기에 종결코돈을 만나 제대로 된 단백질이 생성되지 않음. 이런 setting 하에서 앞의 ATG 주변 서열을 바꿔가며 결과적으로 첫 번째 ATG, 두 번째 ATG 간의 번역 preference가 어떻게 바뀌는지를 관찰해본 결과가 위와 같음.
보면 앞의 ATG 서열을 기준으로 -3 부위에 A or G, +4 부위에 G가 있는 경우에는 앞의 ATG가 번역될 가능성이 매우 높아서 상대적으로 뒤쪽 ATG부터 이어지는 preproinsulin은 제대로 발현되지 않는 것을 알 수 있음. 한편 앞쪽 ATG 주변에 favorable한 서열이 있지 않은 경우 뒤쪽 ATG부터 번역이 시작되어 preproinsulin 신호가 강하게 나타나는 것을 알 수 있음.
그렇다면 -3, +4 부위의 염기가 왜 중요할까.
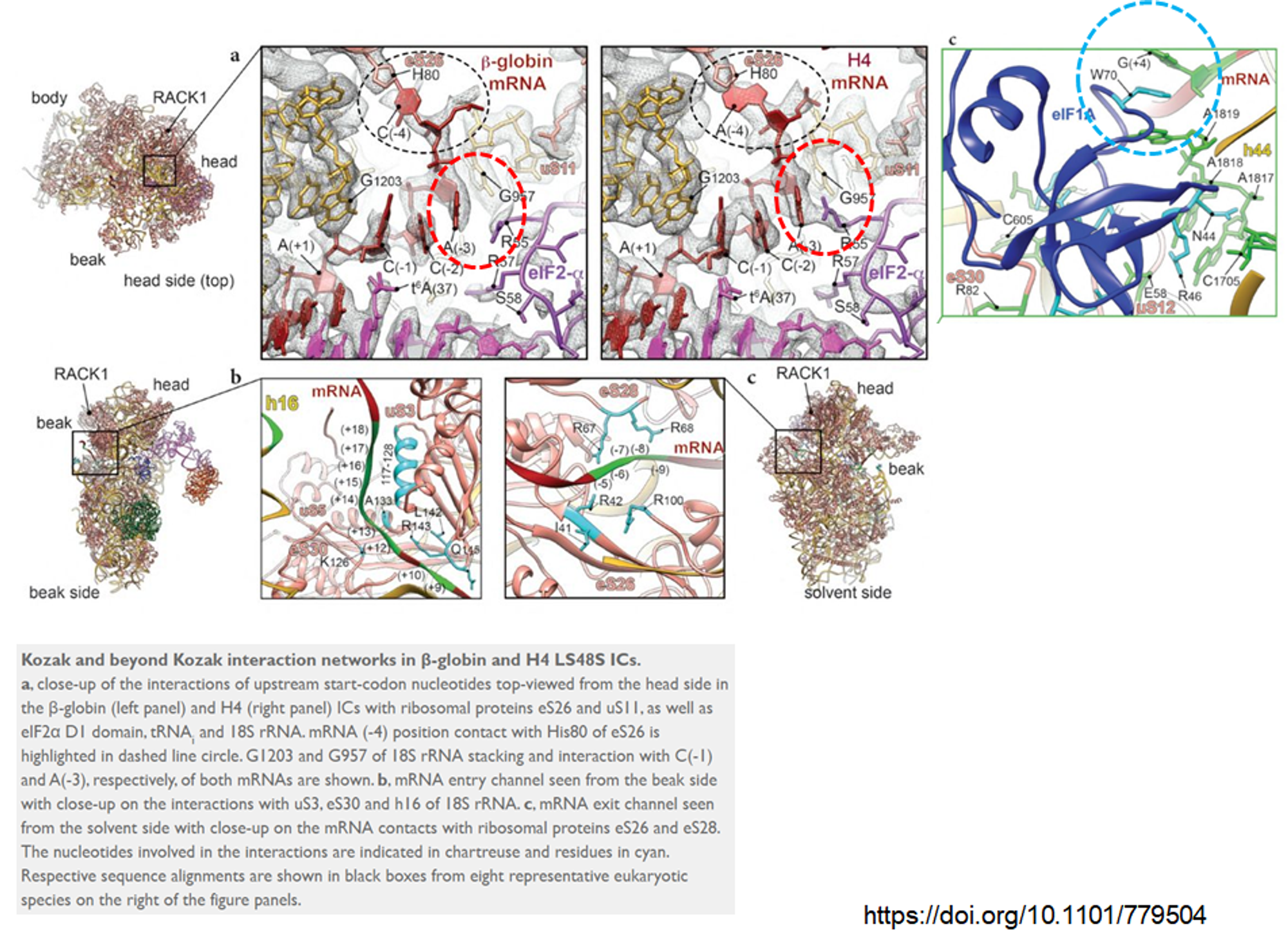
위 그림에 나타나 있는 것처럼 -3 부위의 염기는 eS26 근방의 단백질 등과 상호작용하고, +4부위의 염기는 eIF1A의 트립토판(W) 부분과 상호작용함. 그렇기에 이들 서열이 제대로 구성되어 있어야 비로소 favorable하게 전사 개시가 일어날 수 있음.
한편 mRNA의 sequence 그 자체 뿐만 아니라 mRNA가 형성하는 2차구조에 의해서도 번역 정도가 조절될 수 있음. 이와 관련된 실험이 아래와 같음.
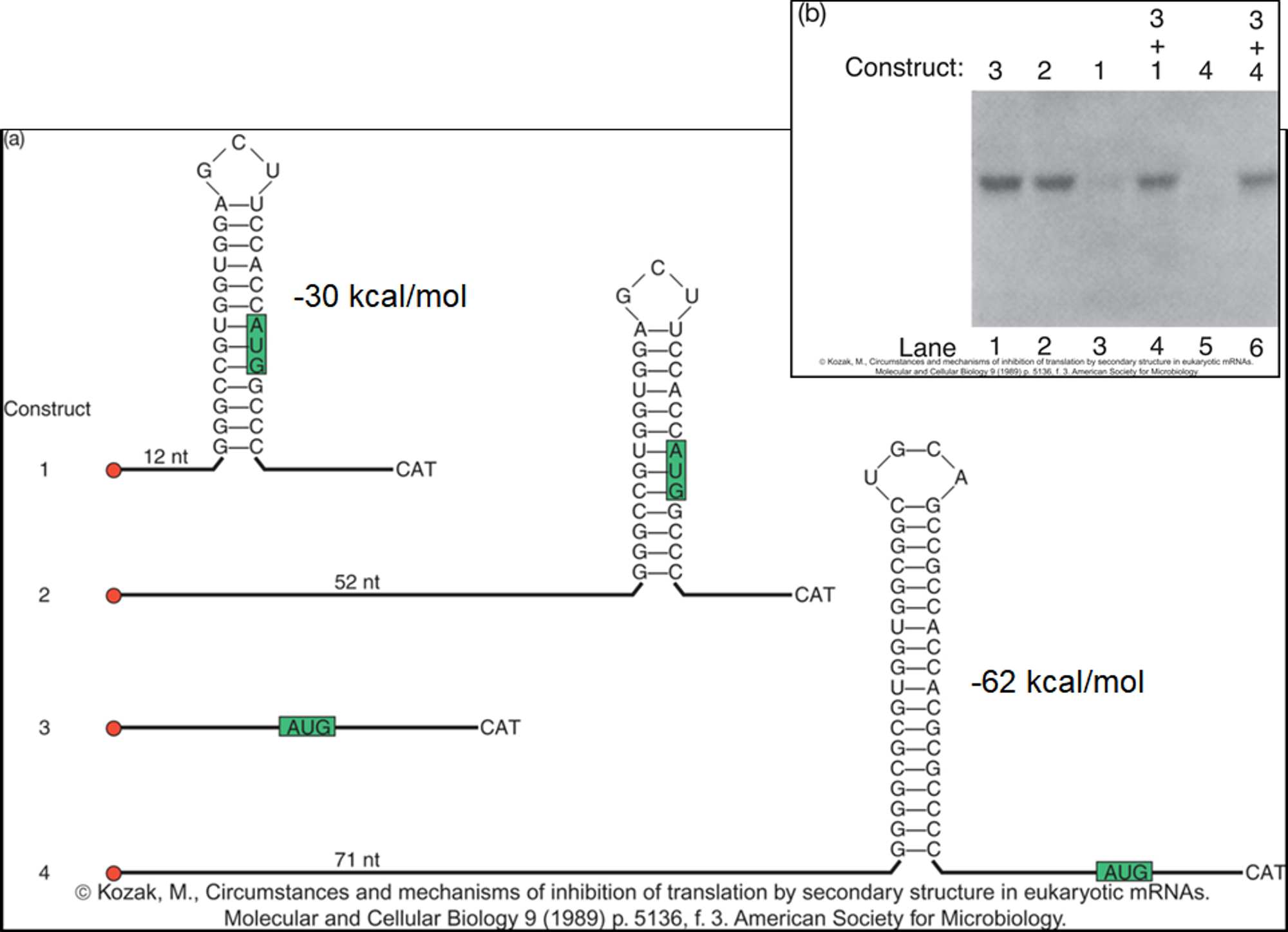
보면 1은 AUG가 포함된 비교적 짧은 hairpin을 가지고 있으며, 이 hairpin의 upstream에 짧은 서열이 존재하는 것이고 2는 1에서와 동일한 hairpin이 있는 대신 hairpin의 upstream에 비교적 긴 서열이 존재하는 것이고, 3은 아예 hairpin이 없는 채로 중간에 AUG가 존재하는 것이며 4는 거대한 hairpin의 downstream에 AUG가 존재하고 hairpin의 upstream에 많이 긴 서열이 존재하는 것임.
이 때 AUG 뒤쪽의 mRNA 서열에 의해 합성되는 CAT이라는 단백질의 발현 정도를 보여주는 것이 위 그림 오른쪽 위의 결과임. 보면 2, 3의 경우 번역이 잘 이루어짐. 그런데 1의 경우 번역이 잘 안 이루어짐. 이를 통해 hairpin 앞쪽 서열이 너무 짧을 시 ribosome이 제대로 붙지 못해서 번역이 잘 안 이루어진다는 것을 알 수 있음. 한편 4의 경우에도 번역이 잘 이루어지지 않는데, 이를 통해 hairpin이 너무 길 시 이 녀석이 일종의 barrier로 기능해서 AUG까지 ribosome이 도달하지 못하게 해버림. (위 그림의 1, 4 hairpin 오른쪽 부분에 각 hairpin을 해체하기 위해 필요한 에너지가 나타나 있는데, 이를 보면 왜 더 긴 hairpin이 barrier 역할을 하는지 더 잘 이해할 수 있을 것임)
다음 포스트부터는 진핵생물 번역개시에 필요한 각종 factor들에 대해 알아보도록 하자.