이번 포스트에서는 지난 포스트에 이어서 tRNA 충전 이후 일어나는 박테리아에서의 번역 개시 과정에 대해 알아보자.
Dissociation of ribosomes into two subunits
일단 tRNA의 충전이 일어나고 나면 그 다음에는 본격적으로 ribosome 수준에서의 반응이 일어남.

위와 같은 일이 처음에 일어나는데, 간단히 설명하자면 처음에는 붙어있었던 50S, 30S subunit들이 RRF, EF-G에 의해 분리되고, 뒤이어 30S subunit에 결합한 IF3 때문에 50S가 30S에 다시 붙는 것이 방지됨. (즉, reassociation이 방지됨)
이제 이와 관련된 실험에 대해 살펴보자.

위와 같이 4가지 동위원소를 사용한다고 생각해보자. 이 때 앞의 3개는 non-radioactive tracer로 단순히 ribosome의 무게를 heavy하게 만들어주는 역할이고, 뒤 1개는 radioactive tracer로 Uracil에 포함되어 radioactivity를 내게끔 해주는 녀석임.
기본적인 idea는 heavy한 medium에서 키워서 무거운 동위원소로 만들어진 ribosome이 포함된 E.coli를 다시금 light medium에 넣어서 배양시킨 후 이들로부터 ribosome을 분리해내서 gel filtration assay를 수행한 것임.
이 때 (a)처럼 한 번 붙은 ribosome 대단위체, 소단위체들이 다시 떨어지지 않는다면 heavy한 ribosome 따로, light한 ribosome이 따로 검출될 것임. 한편 subunit들 간에 exchange가 가능하다면 (b)처럼 서로 mix된 hybrid ribosome이 관찰될 것임.

실제 결과가 위와 같음. 우선 원래는 (a)처럼 heavy, light ribosome 각각의 subunit들에 의해 특징적인 peak들이 관찰됨. 그러나 앞서와 같이 medium을 교체해가며 실험을 수행하였더니 (b)와 같은 결과가 나타남. 보면 완전한 heavy peak도, 완전한 light peak도 아닌 어중간한 peak들이 관찰됨. 이를 통해 subunit간의 exchange가 일어난다는 것을 알게 됨. (즉, 평상시에는 dynamic하게 large subunit과 small subunit 간의 association, dissociation이 일어남)
한편 추가적인 연구를 통해 RRF(ribosome releasing factor)와 EF-G가 ribosome의 두 subunit을 떨어뜨려 준다는 것을 알게 되었고, 떨어진 두 subunit을 다시 붙지 못하게끔 해주는 것이 IF3라는 것도 알게 됨. (IF3는 특별히 30S subunit에 붙어 이런 역할을 수행함)
formation of the 30S initiation complex
30S, 50S subunit들이 분리되고 나면, 본격적으로 아래와 같은 일련의 과정에 의해서 30S initiation complex가 형성됨.

이 때 IF1, IF2는 IF3에 이어 들어오게 되며, 이들이 IF3와 30S subunit 간의 binding을 stabilize해줌. (IF1, 2, 3는 cooperative하게 30S rRNA와 결합함. 즉, IF2가 먼저 들어온 경우, IF1, 3가 뒤이어 들어오며 IF2와 rRNA간의 결합을 stabilize해줌) 참고로 IF2, 3는 처음에 혼자서 일단 binding을 할 수는 있지만 IF1은 아예 혼자서는 binding을 할 수 없음.
위 그림에 나타나 있는 것처럼 30S initiation complex의 형성 과정에는 mRNA, fMet-tRNA 성분이 반드시 첨가되어야 함. 지금부터 이들 각각에 대해 조금 더 알아보자.
30S initiation complex가 형성될 때 가장 먼저 결합하는 amynoacyl-tRNA는 무엇이며 이 녀석이 어떤 codon에 결합할까.
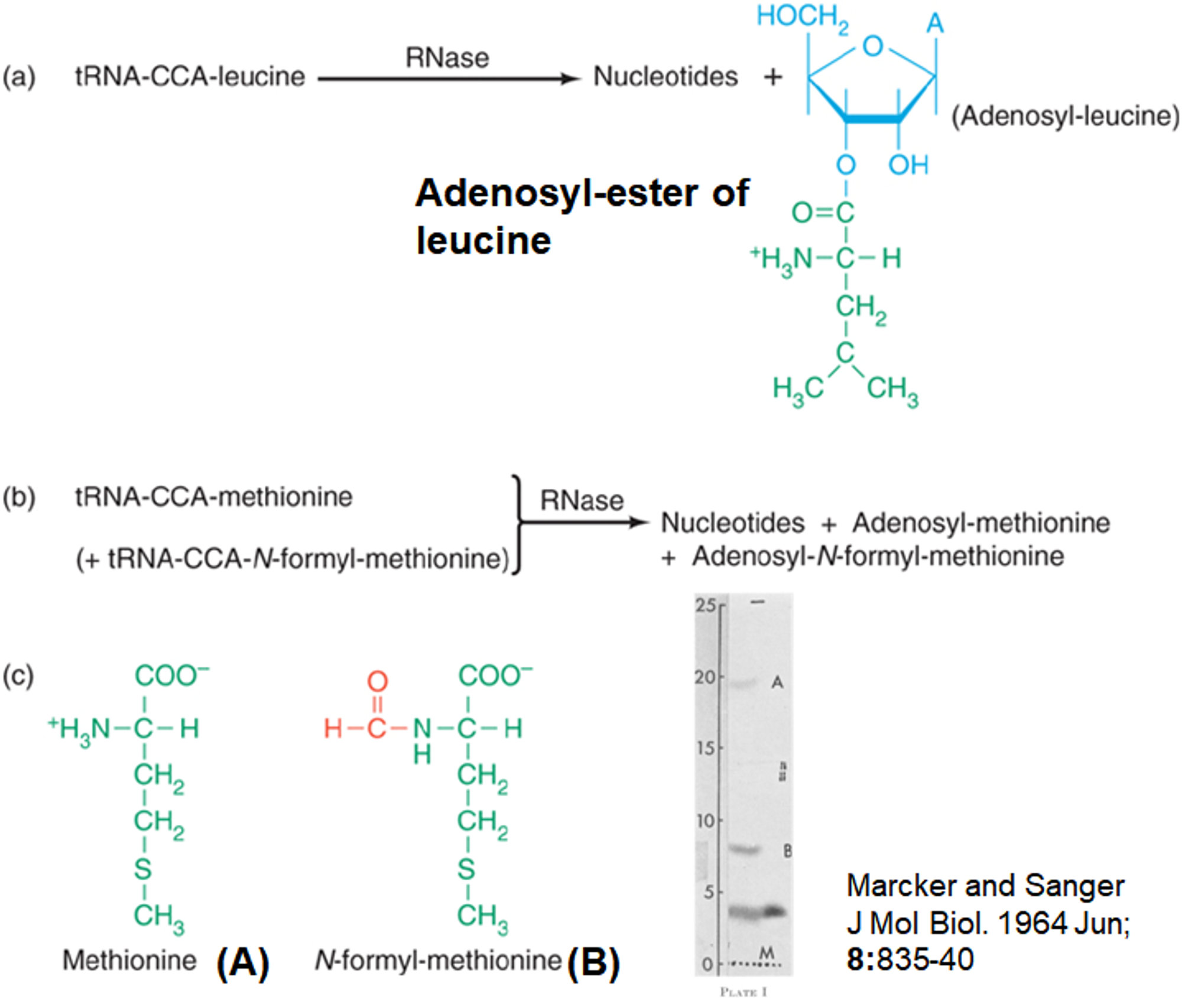
이와 관련된 실험 결과를 살펴보자. 우선 (a)의 경우 tRNA-CCA-leucine에 RNase를 처리했을 때 나타나는 생성물을 보여주고 있음. 보면 leucine과 연결된 adenosyl-ester가 생성물로 나오는 것을 알 수 있음. (이는 당연한데, CCA 중 A 부분과 leucine이 결합해 있었으므로 RNase 처리해서 tRNA의 나머지 성분이 다 분해될 시 adenosyl-ester만 나올 것임) 이러한 결과는 Met이 충전되는 tRNA를 제외한 나머지 complex에 RNase를 처리했을 때도 거의 동일하게 나옴.
그런데 (b)와 같이 tRNA-CCA-methionine에 RNase를 처리한 경우 (c)와 같이 두 종류의 생성물이 나옴. 실제로 (c) 오른쪽의 gel 전기영동 결과를 보면 두 종류의 band가 나오는 것을 확인할 수도 있음.
두 생성물의 구조를 파악해 본 결과 (c)에 나타난 것과 같이 하나는 methionine, 하나는 N-formyl-methionine이었음.

이후 생화학자들을 비롯한 많은 생물학자들의 연구 결과 위와 같은 과정을 통해 N-formyl-methionine이 형성된다는 것을 알게 됨. 이 때 핵심은 methionine이 tRNA에 처음에 그냥 붙은 후에 transformylase에 의해 formyl기가 달려 N-formyl-methionine이 형성된다는 것임. (다시 말해 처음부터 N-formyl-methionine이 tRNA에 충전되는 식의 일은 일어나지 않음)
그렇다면 그냥 methionine이 충전된 tRNA와 N-formyl-methionine이 충전된 tRNA는 도대체 뭐가 다른 것일까. 우선 각각의 tRNA들이 mRNA상의 어떤 codon들과 잘 결합하는지를 파악하기 위해 아래와 같이 filter binding assay를 수행함.

이 때 빨간색으로 나타나 있는 것이 tRNA, 파란색으로 나타나 있는 것이 codon(이 경우에는 UUU만 나타나 있지만 여러 실험 과정에서 각기 다른 codon들을 넣어줌), 검은색 덩어리가 ribosome임. 실험의 기본적인 idea는 ribosome과 함께 결합을 형성한 tRNA, codon만 filter에 남아있을 것이고, 만약 결합을 제대로 형성하지 못했다면 filter 아래로 떨어질 것이므로 이를 이용해 tRNA_m(일반 methionine이 충전된 tRNA)과 tRNA_f(N-formyl-methionine이 충전된 tRNA) 각각이 어떤 서열의 codon과 결합해 30S initiation complex에 recruit되는지를 보고자 함. (사실 이 실험 design을 이용하면 그냥 아무 tRNA를 넣어준 후 특정 codon도 함께 넣어줬을 때 어떤 tRNA가 recruit되는지를 바탕으로 codon이 indicate하는 amino acid가 어떤 종류인지를 알아내는 것도 가능하므로 참고할 것)

우선 tRNA_m의 결과는 위와 같음. 보면 AUG 서열을 사용했을 때에만 특징적으로 ribosome과 함께 filter 위쪽에 남아 있는 것을 알 수 있음.

다음으로 tRNA_f의 결과는 위와 같음. 보면 AUG 뿐만 아니라(물론 AUG가 가장 dominant하기는 함) GUG, UUG를 넣어줬을 때도 어느 정도 ribosome과 함께 filter 위쪽에 남아 있는 것을 알 수 있음. 이를 통해 tRNA_f가 tRNA_m과는 달리 AUG, GUG, UUG 모두에 대해 sequence preference를 가진다는 것을 알 수 있음. (다시 말해 두 type의 tRNA가 약간 다른 sequence specificity를 보임)
이후 tRNA_f와 tRNA_m에 의해 전달된 N-formyl-methionine, methionine이 결과적으로 합성된 protein의 어느 부분에 위치하는지를 알아내기 위해 in vitro translation 실험을 수행하였고, 그 결과가 아래와 같음.

이 실험은 polypeptide의 N-terminal에 tRNA_f와 tRNA_m을 각자 처리해 준 후 N-formyl-methionine, methionine이 N-terminal 부분에 첨가될 확률이 어느 정도인지를 측정하는 것을 목적으로 수행한 것임. 참고로 이 때 UG polymer 형태의 mRNA로 실험을 수행해 보기도 하고, UAG가 포함된 poly UAG mRNA로 실험을 수행해보기도 함. 이 때 poly UAG 같은 경우 AUG의 combination이 나올 수 있으므로 이 실험 결과에 더 주목해보자. 보면 tRNA_m을 넣어준 경우보다 tRNA_f를 넣어준 경우 훨씬 더 tRNA에 충전되어 있던 amino acid가 N-terminal에 붙을 확률이 높음. 결과적으로 tRNA_f가 바로 protein 합성 시 initiation 과정에 첨가되어 활용되는 initiator tRNA이며, tRNA_m의 경우 중간부에 있는 methionine 합성 시 관여하는 tRNA라는 것을 알게 됨.

위 그림에는 tRNA_f과 tRNA_m의 구조가 나타나 있으므로 참고할 것. 이 때 둘 다 anticodon 부위가 3'-UAC-5'으로 되어 있어서 결국 mRNA 중 5'-AUG-3' 부분과 결합하게 된다는 것은 동일함. 다만 세부적인 조성의 차이에 인해 tRNA_f의 경우 AUG(83% in E. coli gene) 뿐 아니라 GUG(14% in E. coli gene), UUG(3% in E. coli gene)와도 어느 정도 결합할 수 있다는 차이가 생기기는 함.
다음 포스트에서는 mRNA가 30S initiation complex에 어떤식으로 첨가될 수 있는지에 대해 이어서 알아보도록 하자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 17.1 : 세균에서의 번역 개시(bacterial translation initiation) - 4 (0) | 2024.02.04 |
|---|---|
| [분자생물학] 17.1 : 세균에서의 번역 개시(bacterial translation initiation) - 3 (0) | 2024.02.04 |
| [분자생물학] 17.1 : 세균에서의 번역 개시(bacterial translation initiation) - 1 (0) | 2024.02.04 |
| [분자생물학] 16.5 : piRNA, miRNA에 의한 조절 (0) | 2023.12.21 |
| [분자생물학] 16.4 : RNAi (RNA interference, RNA 간섭) (0) | 2023.12.21 |