이번 포스트에서는 지난 포스트에 이어서 30S ribosomal subunit에 mRNA가 어떻게 결합할 수 있는지에 대해 알아보도록 하자.
앞서 살펴봤던 것처럼 30S initiation complex 형성 시 mRNA도 같이 들어옴. 그렇다면 도대체 30S ribosome 부위는 mRNA의 어떤 부분을 인식하고 mRNA를 끌어들이는 것일까. 결론적으로 원핵생물에서 30S initiation complex 형성 시 인식되는 mRNA 부분은 Shine-Dalgarno sequence라는 것이 알려져 있음. 이 sequence의 존재를 처음 알게 된 실험 결과가 아래에 나타나 있음.

실험 idea는 매우 간단함. bacteria가 가진 16S rRNA(30S subunit을 이루는 요소)의 3' sequence와 bacteria에 감염되는 viral한 mRNA들 사이에 complementarity가 있는지를 비교해봄. 그랬더니 위 그림 오른쪽에 나타난 것처럼 대부분의 viral mRNA에서 16S rRNA 일부 서열과 상보적인 부분이 있다는 것을 알아냄. (결국 viral한 mRNA는 bacteria의 번역 system을 이용하기 위해서 이런 서열을 가지고 있는 것)
참고로 이 때 bacterial mRNA를 사용하지 않고 viral mRNA를 사용한 이유는 그 당시 bacterial mRNA의 sequence를 잘 모르고 있기도 했고, 쉽게 정제할 수 있는 mRNA가 viral mRNA다 보니 그랬을 것으로 추측 가능함.

결과적으로 위와 같은 recognition이 일어나는 것을 밝혀냄. 이 때 파란색으로 나타나 있는 것이 16S rRNA이고 빨간색으로 나타나 있는 것이 phage mRNA임. 보면 16S rRNA의 3' 끝부 근처 서열이 mRNA의 특정 서열(주로 AGGAGG 혹은 GGAGGA로 고정되어 있음)을 인지하게 되고, 결국 인식된 mRNA 부분보다 대략 10nt 내의 downstream에 AUG가 존재하여서 이 곳부터 translation이 일어나게 됨. 이런 발견의 공적을 사 16S rRNA가 인지하는 mRNA의 sequence를 Shine-Dalgarno sequence로 명명함.

Shine-Dalgarno sequence를 이용하면 위와 같이 expression vector를 만들 수도 있음. 위 그림은 expression plasmid의 한 종류를 나타내 주고 있으며 빨간색 화살표로 표시된 부분에 ribosomal binding site(Shine-Dalgarno sequence)가 들어가 있음을 확인 가능함. 이 site가 존재하게 되면 결국 이 site로부터 대략 10nt 안에 존재하는 AUG부터 쭉 단백질로 번역될 것임. (위 그림에도 실제로 rbs sequence보다 조금 아래에 ATG로 표시되어 있는 부분이 있음을 알 수 있음) 참고로 이런 expression vector를 사용해주면 bacteria에서 특정한 protein을 overexpression시킬 수 있으므로 이런 vector를 다양한 분야에서 활용 가능함.

참고로 이러한 Shine-Dalgarno sequence는 처음에는 물론 viral DNA에서 발견되었지만 추후 실험을 통해 위와 같이 bacteria DNA에서도 viral DNA에서와 유사한 sequence가 나타난다는 것을 확인할 수 있었음.
한편 이전 포스트에서 잠시 등장했던 것처럼 initiation factor로 IF1, 2, 3가 존재하는데, 이들은 앞서 봤던 mRNA의 binding, tRNA의 binding을 도와주는 역할을 함.

위 표에 나타난 결과는 간단히 말해서 ribosome, mRNA와 함께 다양한 조합의 initiation factor들을 넣어줘가면서 mRNA, fMet-tRNA와 ribosome 간의 binding affinity가 어떻게 달라지는지를 나타내준 것임. 하나하나 살펴보자.
우선 IF1, 2를 넣었을 때, IF2만 넣었을 때는 거의 mRNA, fMet-tRNA와 ribosome 간의 binding이 일어나지 않음. 한편 IF3만 단독으로 넣었을 경우 특징적으로 mRNA와 ribosome 간의 결합 정도만 증가함. 한편 IF1과 IF3, IF2와 IF3를 같이 넣어줬을 때는 mRNA와 ribosome 간의 결합 정도가 조금 더 증가하다가 비로소 IF1, 2, 3를 다 넣어주면 mRNA와 ribosome 간의 결합 세기가 가장 세진다는 것을 알 수 있음. 이를 통해 mRNA와 ribosome 간의 결합에 있어서 IF3가 key factor이고 그 밖에 IF1, 2는 IF3의 역할을 보조한다는 것을 알 수 있음.
한편 fMet-tRNA와 ribosome 간의 결합에 있어서는 위 data만 봤을 때는 다소 애매함. 물론 IF2, IF3를 같이 넣어줬을 때 다른 때에 비해서 fMet-tRNA의 binding affinity가 올라갔으므로 IF2가 major한 역할을 하지 않을까 짐작할 수는 있지만 본 실험 결과만으로 확신하기 힘듦. 물론 IF1, 2, 3가 다 있을 때는 fMet-tRNA와 ribosome 간의 결합이 가장 잘 이루어지므로 IF1, 2, 3가 fMet-tRNA가 ribosome에 붙도록 도와주는 녀석임은 확실함.

앞서의 unclear한 부분을 조금 더 명확히 하기 위해 이번에는 다른 system으로부터 유래한 mRNA를 가지고 동일한 실험을 수행함. 그 결과 IF2만 넣어줬을 때 갑자기 fMet-tRNA와 ribosome 간 결합력이 확 증가하는 것을 볼 수 있음. 이를 통해 IF2가 fMet-tRNA와 ribosome 간의 binding에 있어 핵심적으로 작용하는 key binding factor라는 것을 알 수 있음. (이 경우에도 앞서와 마찬가지로 IF1, 3는 IF2의 역할을 보조해준다는 것을 알 수 있음)
방금의 실험 결과에서 알 수 있는 사실 중 하나는 IF2가 fMet-tRNA가 30S initiation complex에 binding할 수 있도록 도와주는 major factor라는 것임. 한편 다른 실험에서는 IF2에 GTP가 존재하며, 이 GTP가 IF2의 30S subunit binding에 있어 핵심적이라는 것을 밝혀냄. 이후 과학자들은 IF2에 있는 GTP가 hydrolysis되면서 fMet-tRNA를 recruit하는 것은 아닐지에 대해 의심하게 됨. (보통 NTP가 associate되어 있으면 이런 의심을 해보는 것이 일반적임) 이를 확인하기 위해 hydrolysis가 일어나지 않는 변형된 GTP를 만들어서 IF2와 결합시켜봄. 보통 ATP의 경우에는 triphosphate에서 O 대신 S를 치환시켜준 화합물을 넣어줘서 hydrolysis를 안일어나게 해주기도 하지만 GTP의 경우(특히 이 경우) 아래와 같은 화합물을 만들어 hydrolysis를 block함.
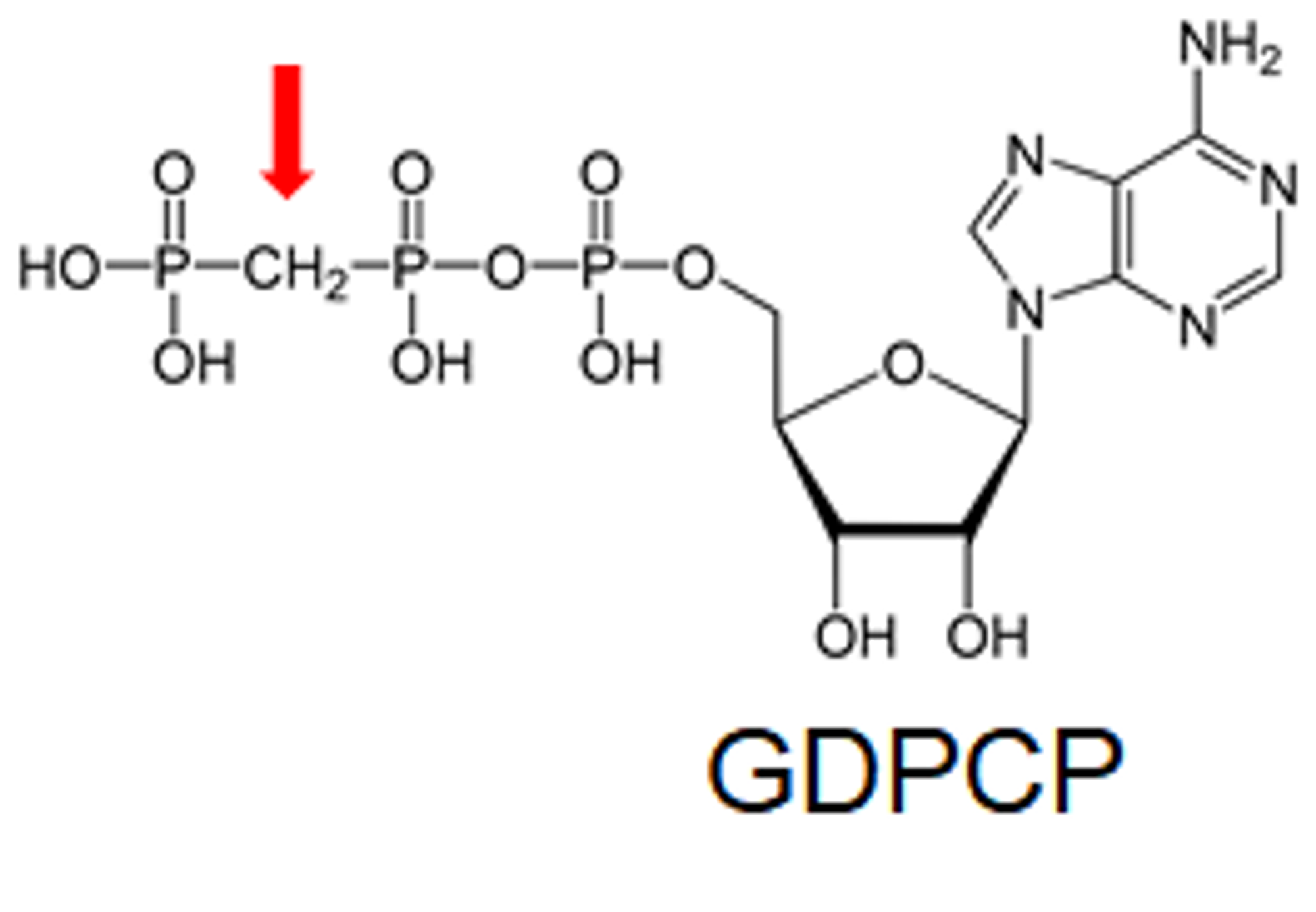
이 화합물의 이름은 GDPCP이며 gamma phosphate와 beta phosphate 사이에 O 대신 methyl기를 넣어줌. 이럴 시 이 녀석은 hydrolysis되지 못함.
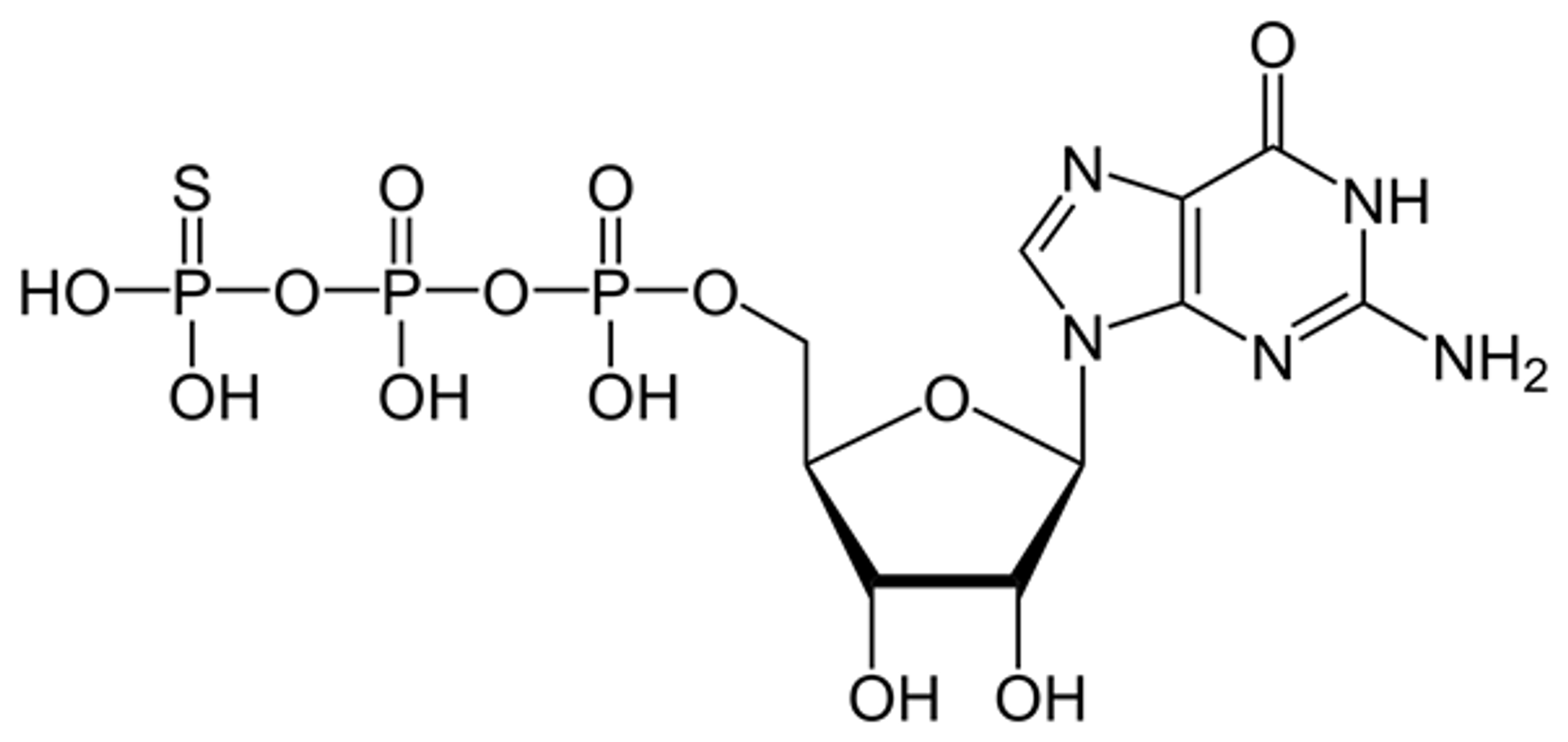
그 밖에 위 그림과 같이 gamma phosphate에 O대신 S를 치환시켜준 화합물을 사용할 수도 있으며, 이 때 이 화합물은 GTPgammaS라고 부름. 이 녀석을 사용해도 GTP의 hydrolysis를 block해줄 수 있음.
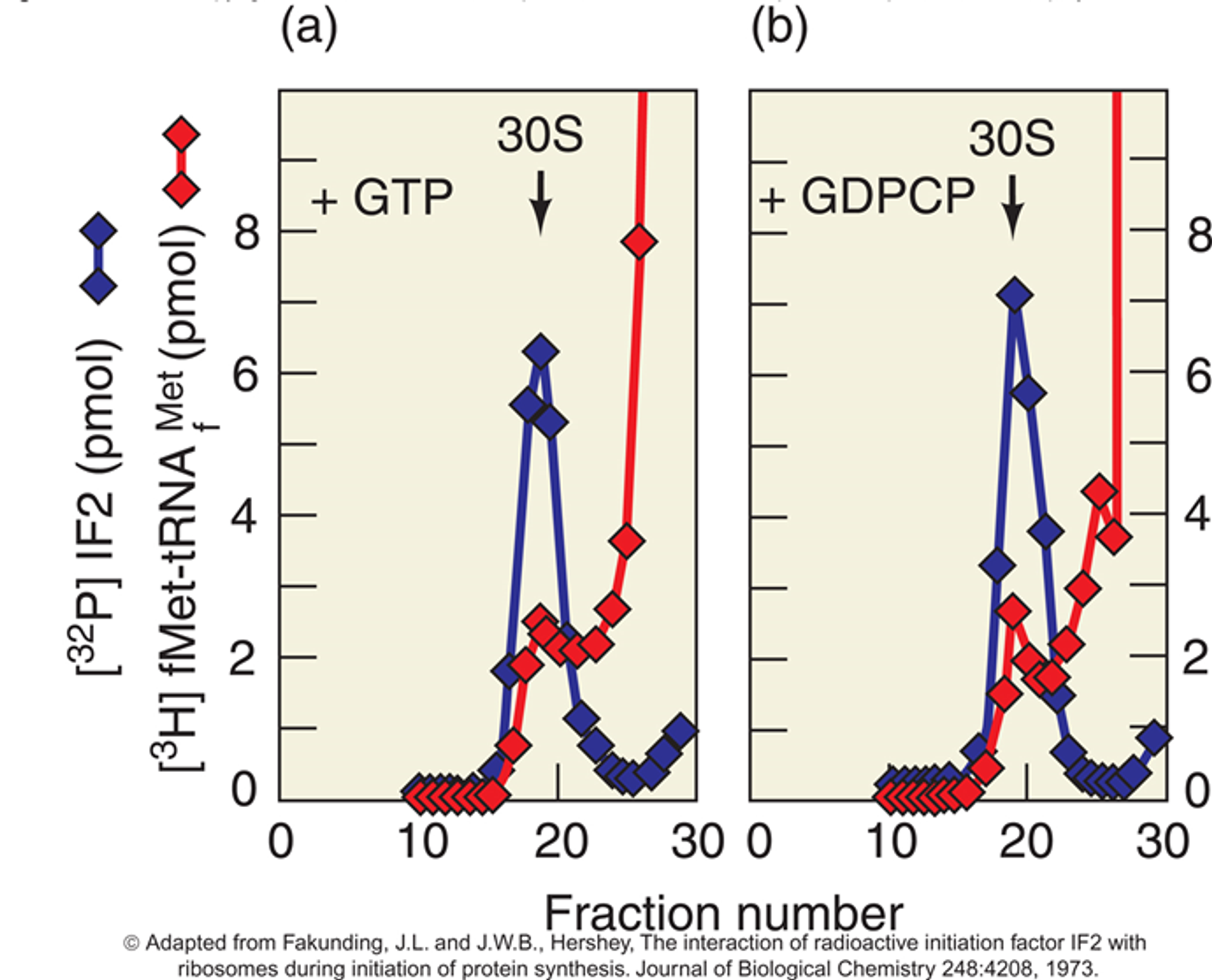
GDPCP를 이용해 수행한 gel filtration assay 결과가 위와 같음. (0 부분이 작은 size, 30부분이 큰 size) 우선 (a)는 정상적인 GTP를 사용했을 때의 결과이고 (b)가 GDPCP를 사용했을 때의 결과임. 이 때 붉은색으로 표시된 것은 3H를 이용해 fMet-tRNA를 labeling한 것이고 파란색으로 표시된 것은 32P를 이용해 IF2를 labeling한 것임.
결과를 보면 30S 위치(30S subunit이 위치하고 있는 곳)에서의 fMet-tRNA 비율이 GTP를 사용했을 때와 GDPCP를 사용했을 때 거의 차이가 나지 않음. (peak가 유사하게 나타남) 이를 통해 IF2에 붙은 GTP가 굳이 hydrolysis되지 않아도 initiator tRNA가 30S subunit에 recruit되는 데는 문제가 없다는 것을 알 수 있음.
결론적으로 IF2에 붙어 있는 GTP는 그 존재 자체가 중요하지 이 녀석이 hydrolysis되는 것이 30S initiation complex 형성에는 중요하지 않음.
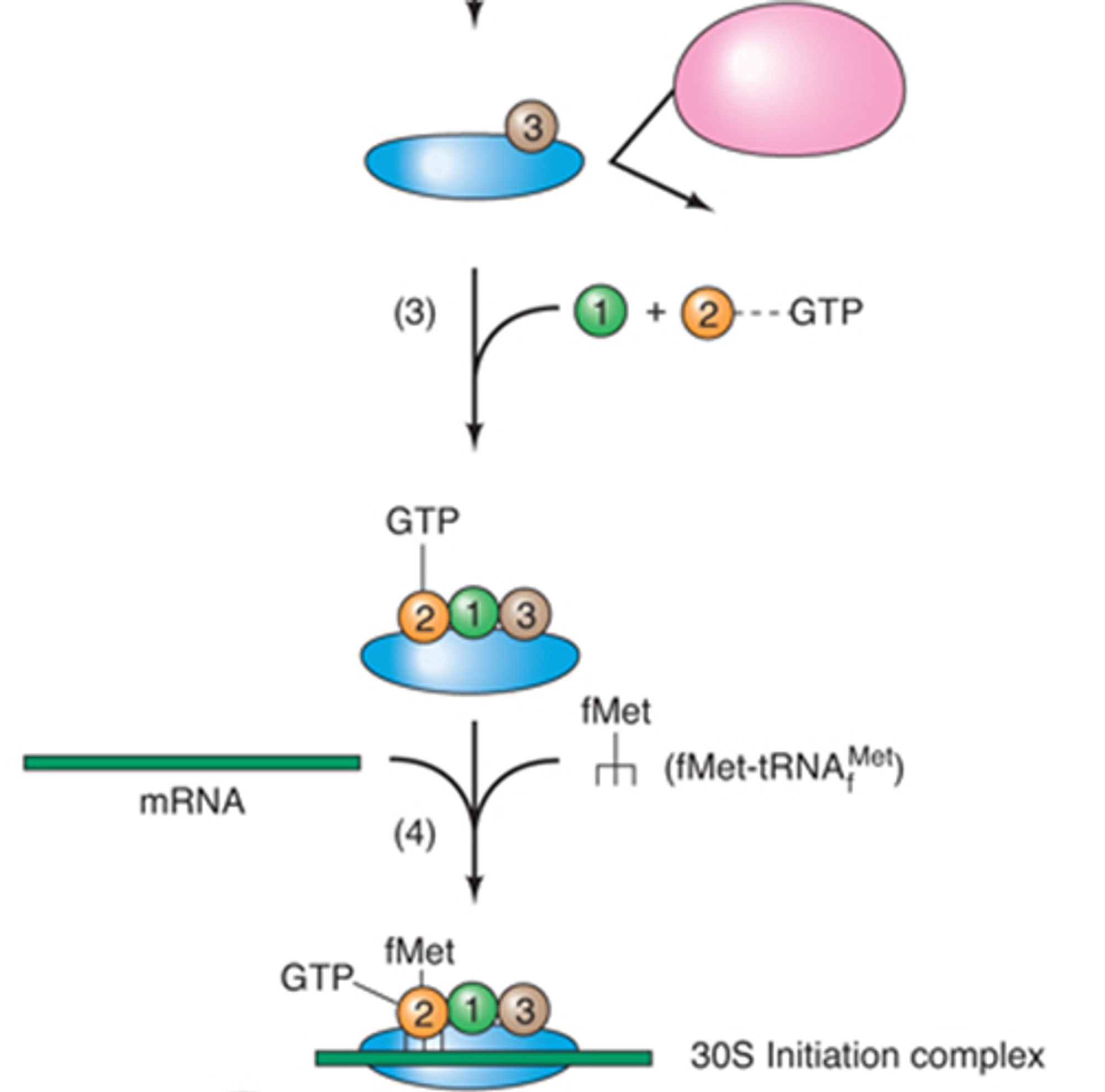
결국 이런 일련의 연구를 거쳐서 위와 같은 sequential한 과정이 밝혀졌음. 이 때 중요한 것은 30S initiation complex를 형성할 때 각종 factor, element들이 sequential하게 들어온다는 것임. 그 밖에 IF1, 2, 3가 모두 하나씩 모여서 30S initiation complex를 형성한다는 것도 기억해둘 것. (이런 개수와 관련된 정보는 stoichiometry를 측정해 알아냄)
다음 포스트에서는 30S initiation complex 형성 이후에 일어나는 일들에 대해 알아보도록 하자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 17.2 : 진핵생물에서의 번역 개시(eukaryotic translation initiation) - 1 (0) | 2024.02.04 |
|---|---|
| [분자생물학] 17.1 : 세균에서의 번역 개시(bacterial translation initiation) - 4 (0) | 2024.02.04 |
| [분자생물학] 17.1 : 세균에서의 번역 개시(bacterial translation initiation) - 2 (0) | 2024.02.04 |
| [분자생물학] 17.1 : 세균에서의 번역 개시(bacterial translation initiation) - 1 (0) | 2024.02.04 |
| [분자생물학] 16.5 : piRNA, miRNA에 의한 조절 (0) | 2023.12.21 |