이번 포스트에서는 세균을 포함한 원핵세포에서 일어나는 RNA 전사에 있어서 매우 중요한 역할을 수행하는 RNA polymerase와 promoter에 대해 알아보자.
RNA polymerase (RNA 중합효소)
원핵생물이 사용하는 RNA polymerase의 물성에 대해 SDS-PAGE를 통해 처음으로 밝혀낸 것은 1969년의 일임. 그 당시의 SDS-PAGE 결과는 아래와 같음.

이 때 이 실험 결과를 통해 한 polypeptide로 구성된 줄 알았던 RNA polymerase가 실제로는 alpha, beta, beta', sigma의 subunit(소단위체)들로 이루어져 있음을 알 수 있었음. 한편, 다른 실험을 통해 이 밖에 omega 소단위체도 존재함이 밝혀짐.
이 때 alpha, beta로만 이루어진 protein을 core enzyme이라 하고 alpha, beta, sigma로 이루어진 protein을 holoenzyme이라고 하는데, 이 둘의 transcription activity(전사 활성) 비교를 통해 sigma subunit의 역할을 어느 정도 이해할 수 있었음.

위 표에서 intact에 해당하는 것은 손상이 되어 있지 않은 DNA를, nicked는 손상된 DNA를 나타냄. 우선 intact의 경우 holoenzyme activity가 core enzyme activity에 비해 월등히 높음. 한편 nicked의 경우 core enzyme의 activity가 intact에 비해 조금 더 증가함.
이 때 일반적으로 RNA polymerase는 nick이 있으면 이 nick에 비특이적으로 붙어 전사를 진행하는 경향이 있음. 이 두 데이터를 비교해봤을 때 nick이 없을 때 core enzyme에 비해 holoenzyme의 transcription activity가 높다는 것은 sigma subunit이 DNA의 특정 서열(promoter)에 RNA polymerase를 결합시켜주어서 전사를 개시하는 역할을 할 것이라는 것을 암시함. 실제로 sigma subunit은 그런 역할을 수행하며, 그렇기에 이 subunit을 specificity factor라고도 부름.
promoter (프로모터)
그렇다면 RNA polymerase와 DNA 중 promoter 부분의 결합은 어떤 방식으로 측정할 수 있을까. 이전 포스트에서 배운 nitrocellulose filter binding assay를 사용할 시 아래와 같은 결과를 얻을 수 있음.

이 때 실험 맨 처음에는 labeled DNA를 사용하지만 시간이 점차 지남에 따라 과량의 non-labeled DNA를 넣어주게 됨.
결과를 보면 holoenzyme의 경우 시간이 경과되어도 labeled DNA와 그대로 붙어있지만 core enzyme의 경우 시간이 경과되면 labeled DNA와 떨어져서 non-labeled DNA와 붙게 됨. 이는 holoenzyme의 경우 처음부터 쭉 DNA와 단단히 결합해서 계속 그 결합이 유지되는 반면 core enzyme의 경우 처음에는 DNA와 꽤 잘 결합되는 듯 보이지만 결국 제대로 결합이 일어나지 않고 붙었다 떨어졌다를 반복하게 된다는 것을 의미함. (참고로 이 때 graph의 y축은 filter 부분의 radioactivity 정도임) 이는 앞서 살펴본 sigma factor의 역할을 생각하면 충분히 납득 가능한 실험결과임.

한편 nitrocellulose filter binding assay를 이용해서 RNA polymerase와 DNA의 결합력을 온도에 따라 측정해본 결과 위 그림과 같이 37도에서 DNA와 RNA polymerase간의 결합이 가장 잘 일어남을 확인 가능함.
그렇다면 실제로 RNA polymerase는 promoter에 어떤 식으로 결합할까.
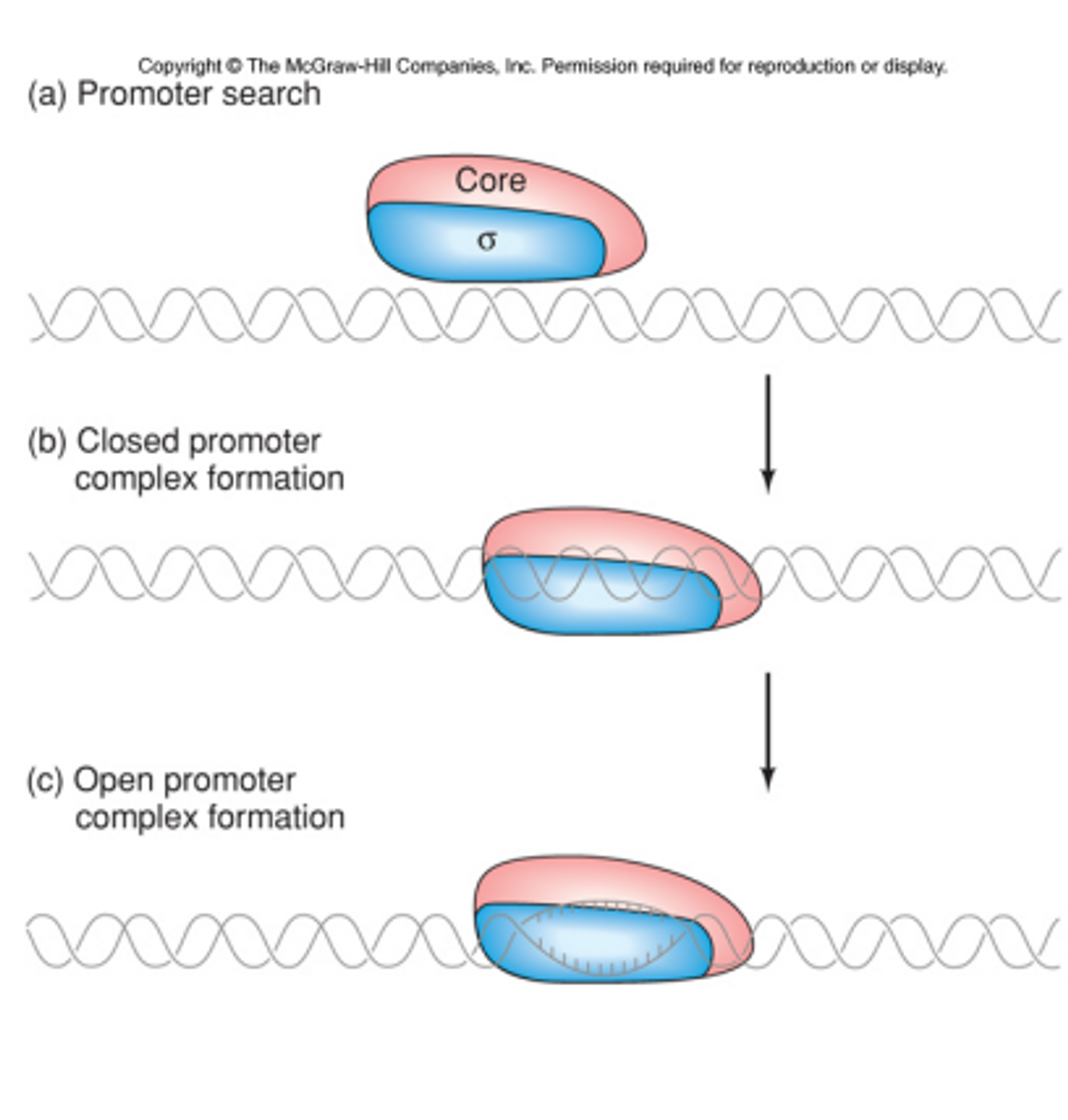
위 그림에 그 과정이 대략적으로 나타나 있음. 이 때 (a) 단계는 RNA polymerase holoenzyme이 DNA와 매우 느슨하게 결합해서 쭉 DNA를 훑으면서 promoter를 찾는 과정임.
다음으로 (b) 단계에서는 sigma factor에 의해 어느 정도는 DNA와 RNA polymerase 간의 결합이 단단해지지만, 아직 DNA가 단일가닥으로 열리지는 않은 closed promoter complex가 생성됨.
마지막으로 (c) 단계에서는 sigma factor가 DNA를 열어주게 되고 그 결과 open promoter complex가 생성됨. (이 때가 DNA, RNA polymerase 간의 결합이 가장 한 상태)
그렇다면 sigma factor는 구체적으로 promoter의 어떤 부분에 붙는 것일까. sigma factor가 붙는 promoter 내부 element를 core promoter element라 부르며, 아래 그림과 같음.
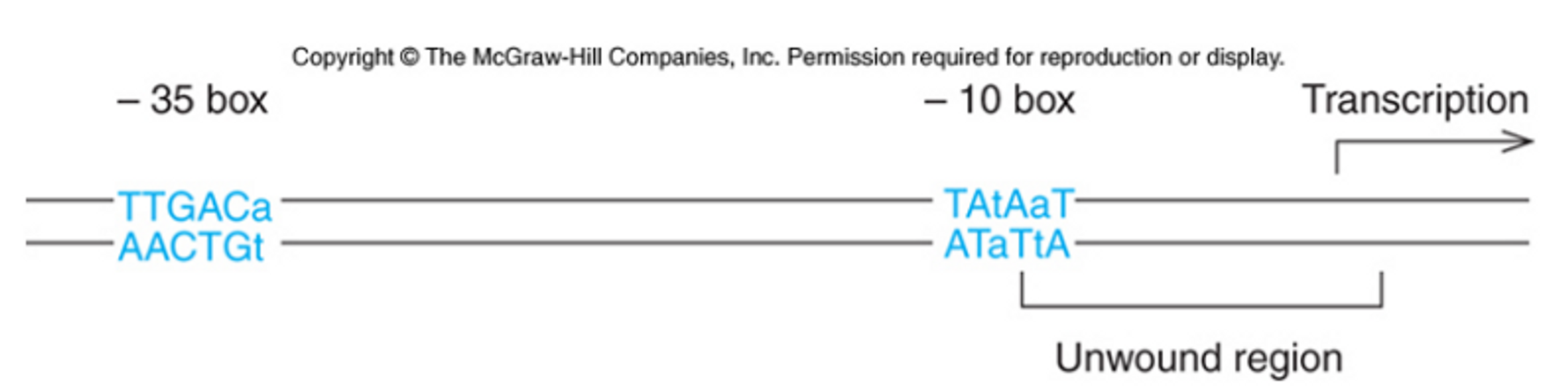
참고로 이 때 일반적으로 transcription이 시작되는 부분을 +1로 표현함. 따라서 -10, -35는 transcription 시작부위로부터 얼마나 upstream(위쪽)에 있는지를 나타내주고 있음. 이 때 위 그림에 파란색으로 나타난 것과 같은 서열은 일반적으로 매우 보존되어 있으며 그 중에서도 대문자는 더욱 보존되어 있음. (소문자로 나타낸 서열은 이에 비해 덜 보존되어 있음)
결과적으로 그림상에 파란색으로 나타난 부분에 sigma factor가 붙게 됨. 따라서 대문자에 해당하는 부분이 mutation되어 다른 서열로 바뀌게 되면 sigma factor가 promoter에 제대로 못 붙게 될 것임. (반면 이미 이상해져있던 서열을 다시 위에 나타난 것과 같은 파란색 서열로 바꿔주는 mutation은 오히려 sigma factor의 결합능을 올려줄 수 있음)
한편 core promoter element보다도 더 upstream에 위치한 UP element도 있는데, 이 부분은 transcription-activator protein인 Fis라는 녀석이 붙는 site로 작용해서 전사를 촉진시켜줌.
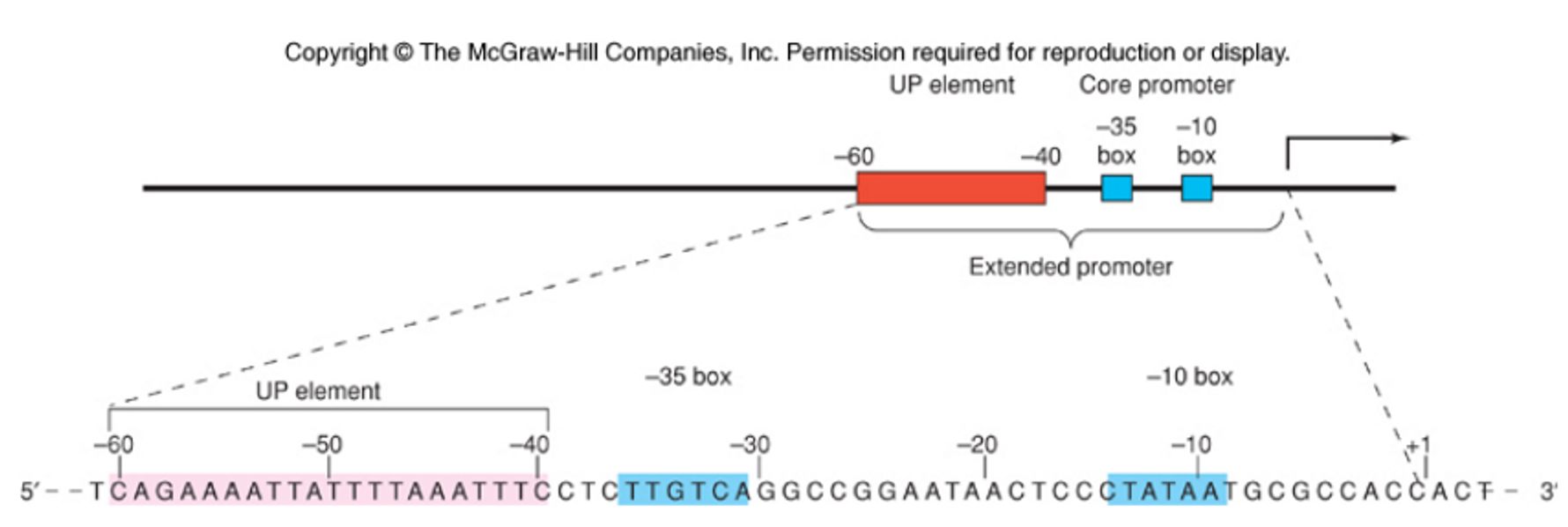
위 그림은 rRNA gene(rrn)에서의 UP element와 core promoter element를 나타내주고 있음. 그런데 rrn gene은 그 역할을 생각해보면 당연하게도 단백질을 매우 많이 합성해야 되는 상황에서 매우 강하게 활성화됨.
그렇다면 이러한 주변 상황이 어떻게 전사에 영향을 미칠 수 있을까. 우선 이러한 조절은 주로 promoter를 대상으로 이루어짐. 구체적으로, i(initiating)NTP가 있을 경우에는 rrn promoter가 활성화되며, alarmone ppGpp(tRNA에 붙은 amino acid가 부족하다는 신호)가 있을 경우에는 rrn promoter가 억제됨.
다음 포스트부터는 본격적으로 원핵세포에서의 transcription initiation(전사 개시)에 대해 자세히 알아보자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 6.2 : 원핵생물의 transcription initiation(전사 개시) - 2 (0) | 2023.08.30 |
|---|---|
| [분자생물학] 6.2 : 원핵생물의 transcription initiation(전사 개시) - 1 (0) | 2023.08.27 |
| [분자생물학] 5.9 : knockout(낙아웃), transgene (0) | 2023.07.04 |
| [분자생물학] 5.8 : IP (면역침강법), yeast two hybrid assay (효모단백질잡종법) (0) | 2023.07.04 |
| [분자생물학] 5.7 : DNA-단백질 상호작용 - nitrocellulose filter, gel mobility shift, footprinting, ChIP (0) | 2023.07.02 |