이번 포스트에서는 저번 포스트에 이어서 원핵생물의 전사개시 과정에 대해 알아보자.
지난 포스트의 마지막에서는 sigma factor가 다시 재사용되기 위해 거치는 sigma cycle에 대해 알아봤었음.
그렇다면 sigma-cycle은 어떻게 일어날까. 이와 관련된 가설이 두 가지 존재함. 첫 번째는 obligate release 가설이고 두 번째는 stochastic release 가설임. 첫 번째 가설은 initiation이 일어나자마자 sigma factor가 바로 떨어져버린다는 것이고 두 번째 가설은 시기에 상관없이 random하게 sigma factor가 떨어진다는 것임. 현재는 두 번째 가설이 학계에서 더 인정받고 있는 상황임.
그렇다면 이러한 가설들에 대한 검증은 어떤 방식으로 이루어졌을까.
우선 Fluorescence Resonance Energy Transfer(FRET)를 이용하면 이를 검증할 수 있음.

FRET의 기본적인 idea는 위 그림에 나타나 있는 것처럼 donor 형광물질, acceptor 형광물질을 이용해 donor가 흡수 가능한 excitation wave(흡수 파장)를 가해주었을 때 acceptor가 donor와 충분히 가까워서 energy transfer에 의해 acceptor 고유의 emission wave(방출 파장)를 방출하는지를 보는 과정을 통해 특정 두 분자가 가까운 곳에 위치해 있는지, 멀리 위치해있는지를 판단하는 것임.
위 그림에서는 크게 Trailing-edge FRET과 leading-edge FRET 방식이 나타나 있음.
우선 Trailing edge FRET부터 살펴보자. 그림에도 나타나 있는 것처럼 D(donor)는 sigma factor에, A(acceptor)는 DNA의 5' 말단에 붙어 있는 상황임. 이 때 obligate release, stochastic release 각각에 대해 어떤 결과가 나올지를 생각해보자. 보면 obligate release의 경우 처음에는 D와 A의 거리가 가까워 FRET이 일어나지만, initiation 이후 sigma factor가 바로 떨어진다면 D와 A의 거리가 멀어져서 FRET이 일어나지 않을 것임. 다음으로 stochastic release의 경우에도 마찬가지로 처음에는 D, A의 거리가 가까워 FRET이 일어나지만, 점차 RNA 전사가 진행됨에 따라 A와 D의 거리가 멀어져 FRET이 일어나지 않게 될 것임. 따라서 trailing-edge FRET을 통해서는 두 가설에서의 결과가 동일하게 나타나므로 둘 중 어느 것이 맞는지를 검증하지 못함.
다음으로 leading-edge FRET에 대해 알아보자. 이 때 D는 sigma factor에, A는 DNA의 3' 말단에 붙어있음. 이 경우 만약 obligate release 가설이 맞다면 처음에도 A, D의 거리가 멀어서 FRET이 일어나지 않고, 나중에는 아예 sigma factor가 떨어져버리므로 FRET이 마찬가지로 일어나지 않을 것임. 한편 stochastic release 가설이 맞다면 처음에는 A, D 간의 거리가 멀기 때문에 FRET이 일어나지 않지만 점차 DNA의 3' 말단과 RNA polymerase의 거리가 가까워지고, 우연히 sigma factor가 떨어지지 않은 경우라면 A, D 간의 거리가 충분히 가까워져 FRET이 일어날 것임.

위 data는 실제 실험 결과임. 보면 Trailing-edge FRET을 통해서는 RNA 중합이 진행됨에 따라 FRET 활성이 감소했으며(이것만으로는 어느 가설이 맞는지 모름) Leading-edge FRET을 통해서는 RNA 중합이 진행됨에 따라 FRET 활성이 증가된 경우가 확인됨. 따라서 이 결과는 stochastic release model을 지지해줌.
이 외에 stochastic release model에 힘을 실어주는 또 다른 증거도 있음.

위 그림은 polymerase를 세포에서부터 얻어낸 다음 in vitro 환경에서 transcription을 시킨 뒤 gel electrophoresis를 통해 polymerase 각각에 대한 probe(탐침)를 사용해서 각 subunit들이 존재하는지 여부를 확인한 것임. 이 때 Holo로 표시된 것은 holoenzyme을 다 넣어준 것을 뜻하며, EC는 elongation complex를 뜻하고, 위첨자 S는 stationary cell(정체된 cell), 위첨자 E는 분열이 활발한 cell을 의미함.
data를 살펴보면 Holoenzyme에 비해 조금 연해지기는 했지만 elongation complex에서도 일부 sigma factor가 관찰된다는 것을 알 수 있음. 따라서 이 결과는 stochastic release model을 지지해줌.
이제 다음으로 promoter 부근에서의 local한 DNA melting에 대해 알아보자. 일단 아래와 같은 실험을 통해 DNA가 melting되는(즉, single strand가 되는) size를 알 수 있음.

이 때 위 그림의 (a)는 실험에 사용하는 agent인 DMS가 염기 A에 methylation을 유발한다는 것을 나타내주고 있음. (b)는 실험의 과정을 나타내고 있는데, 우선 DNA에 labeling을 수행한 다음, RNA polymerase와 이 DNA를 결합시키고 (그러면 melting이 일어날 것) 이 후 DMS를 처리해 A에 methylation을 유발함. 이후 RNA polymerase를 다시 제거해주게 됨. 그러면 나머지 부위는 다시 잘 붙게 될텐데 하필 melting된 부분 중 methylation된 A가 있는 부분은 T와 다시 상보적인 결합을 형성하지 못하고 single strand로 남아있음. 이후 S1(single strand를 잘라주는 효소)을 처리해주게 되면 methylated A를 기점으로 다양한 fragment들이 나타나게 됨. 이를 전기영동을 통해 확인해보게 되면 가장 짧은 길이에 의해 나타난 band와 가장 긴 길이에 의해 나타난 band의 거리차를 통해 melting region의 크기(bubble size)를 알아낼 수 있음.
실제로 이런 방법을 통해 bubble의 크기를 측정해 본 결과 일반적으로 12bp, 조금 더 길게는 17~18bp정도의 크기를 가짐을 알게됨.
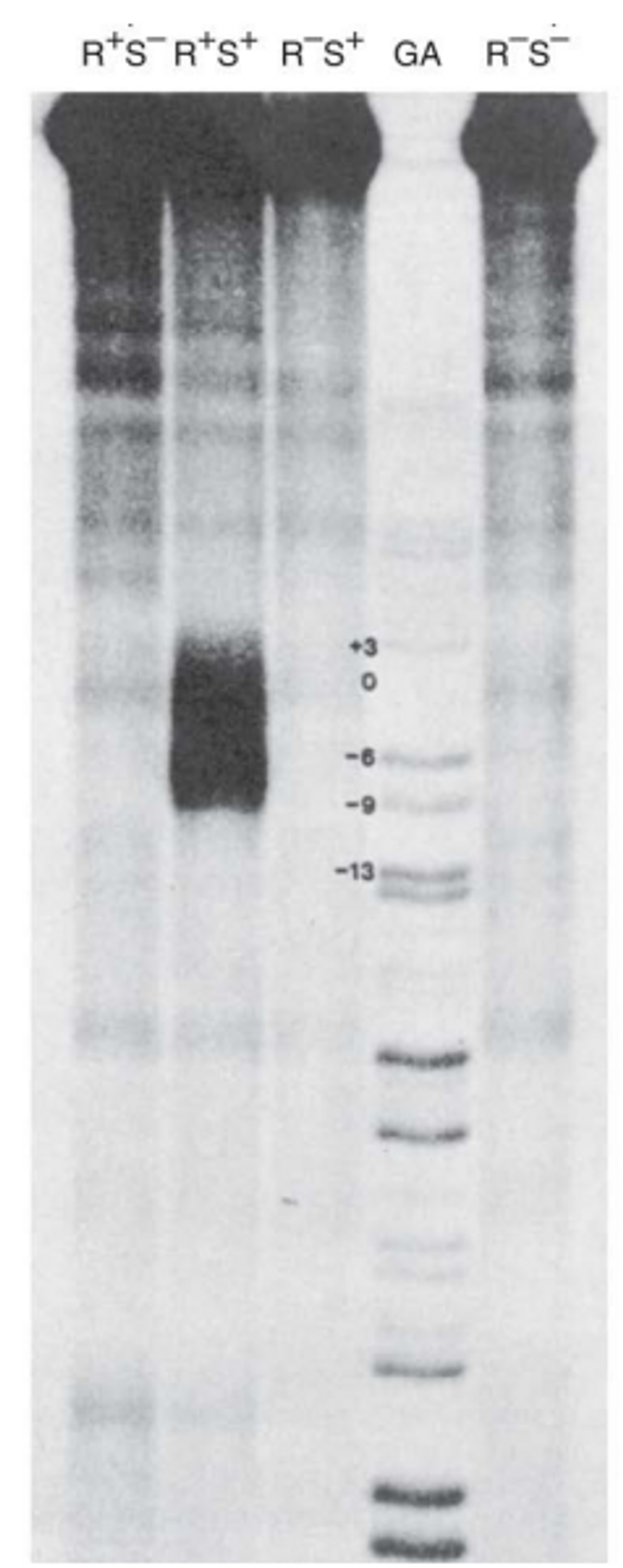
위 그림은 실제 data임. 이 때 S+는 S1을 처리해준 sample을, R+는 RNA polymerase를 사용해준 sample을 의미함. 실제로 결과를 보면 R+S+에서 가운데 부분에 뚜렷한 band가 나타남이 확인됨. 이 band의 두께에 해당하는 만큼이 bubble의 크기임. (여기서는 대략 13bp정도로 보임)
한편 이 밖에 bubble의 size를 파악하는 또 다른 방법도 존재함.
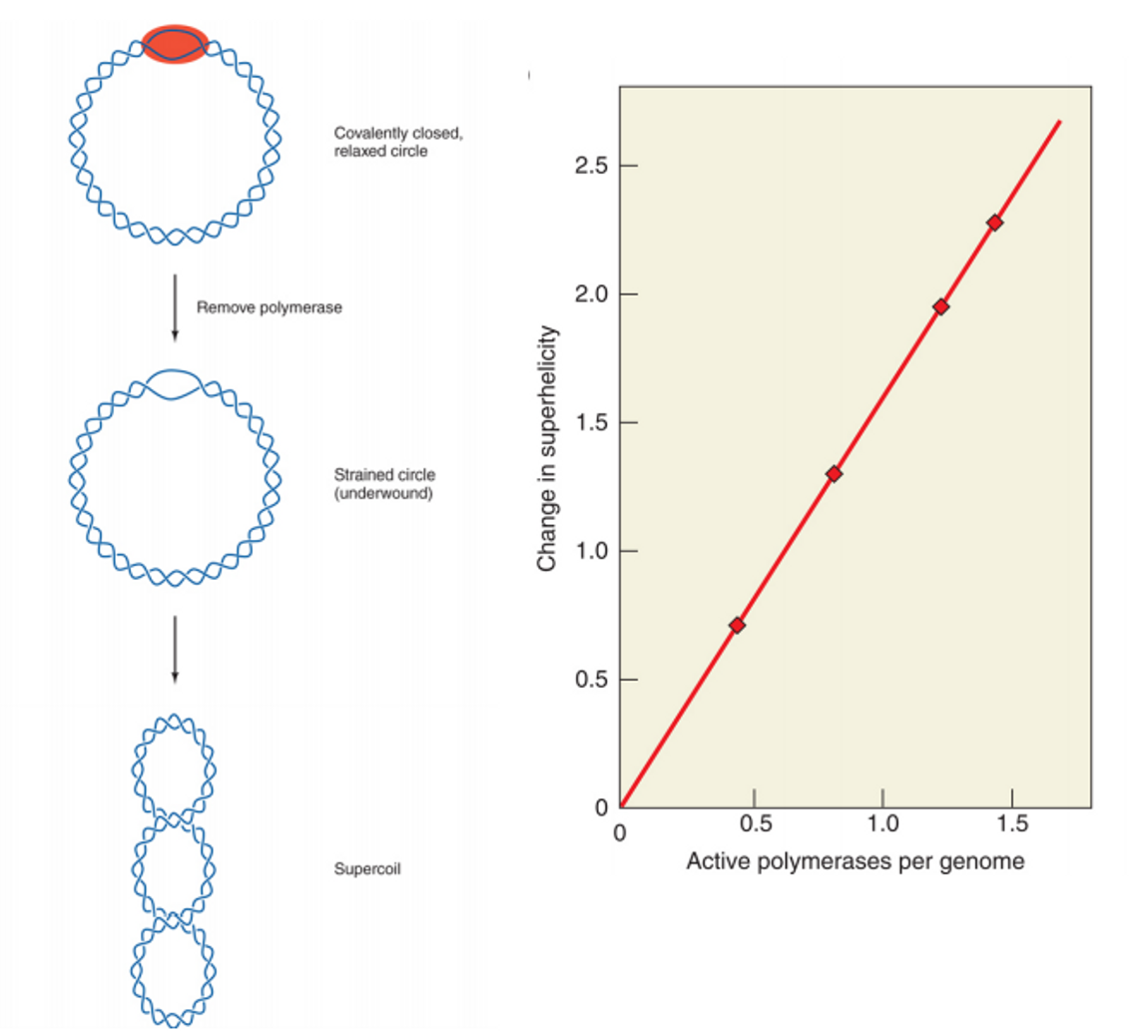
이 방식은 위 그림에 나타난 것과 같이 원형 DNA에 적용 가능한데, 일단 DNA가 일부 풀리게 되면 (그 이후 DNA melting의 주 원인인 polymerase를 제거해주고 나면) DNA가 풀린 정도에 따라 위 그림 왼쪽 아래와 같이 전체 원형 DNA의 supercoiling이 일어나게 됨. 이 때 supercoiling이 일어난 정도에 따라 어느 정도의 DNA가 melting되었는지를 알 수 있음.
그러면 위 그림에 주어진 data를 이용해 대략 계산해보자. 우선 이 때 genome 당 active한 polymerase가 1개일 때를 생각해보자. (active한 polymerase가 1개여야 polymerase 1개에 의해 melting된 DNA의 길이를 알 수 있을 것임) 이 때의 supercoiling 변화 정도는 대략 1.6임. 따라서 이를 바탕으로 DNA가 대략 1.6바퀴정도 풀렸다는 것을 유추할 수 있음. 한편 DNA 이중나선이 한 바퀴 꼬일 때 총 10.5bp의 DNA가 포함되므로, 1.6에 10.5를 곱한 16.8개가 바로 DNA melting size임.
다음 포스트에서 이어서 원핵생물의 전사 과정에 대해 알아보도록 하자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 6.2 : 원핵생물의 transcription initiation(전사 개시) - 4 (0) | 2023.08.30 |
|---|---|
| [분자생물학] 6.2 : 원핵생물의 transcription initiation(전사 개시) - 3 (0) | 2023.08.30 |
| [분자생물학] 6.2 : 원핵생물의 transcription initiation(전사 개시) - 1 (0) | 2023.08.27 |
| [분자생물학] 6.1 : 원핵생물의 RNA polymerase (RNA 중합효소), promoter(프로모터) (0) | 2023.07.04 |
| [분자생물학] 5.9 : knockout(낙아웃), transgene (0) | 2023.07.04 |