이번 포스트에서는 지난 포스트에 이어 진핵생물에서의 번역 개시 조절과정에 대해 알아보자.
진핵생물에서의 translation control 과정과 관련된 가장 유명한 단백질 중 하나는 바로 Heme-controlled repressor(HCR), 혹은 heme-regulated inhibior(HRI)로 불리는 녀석들임. 이 녀석은 EIF2AK1이라는 gene에 의해 발현되는데, gene의 이름을 봐도 알 수 있는 것처럼 eIF2α를 인산화시켜주는 4가지 효소 중 하나임. HRI는 Heme(혹은 Heme을 구성하는 heavy atom)이 부족한 조건에서 발현되며, 이 녀석에 의해 결과적으로 global한 translation이 억제되게 됨.

대략 위 그림과 같은 과정에 의해 translation이 억제됨.
앞서 말했듯 HRI는 위 그림에 나타나 있는 것처럼 eIF2α(GTP가 달려 있는 부분)에 인산기를 달아줌. 그렇게 될 시 바로 그 다음 반응이 block되지는 않고, 위 그림에서와 같이 Met-tRNA도 잘 recruit되고 40S subunit도 잘 recruit되며 이후 GTP가 GDP로 가수분해되면서 eIF2가 release되는 것까지도 정상적으로 잘 일어남. 그런데 이 때 GDP를 다시 GTP로 바꿔주는 과정에서 eIF2B가 작용하게 되는데, eIF2B가 GDP를 GTP로 바꿔주기 위해 eIF2에 binding하고 나면 α에 있는 phosphate에 의해 eIF2B가 다시 떨어지지 못하게 됨.
결과적으로 다시 GDP가 GTP로 바뀌었음에도 계속 붙어있는 eIF2B에 의해서 또 다른 cycle 내에 eIF2가 투입되지 못하게 되면서 (initiation complex가 제대로 만들어지지 않아)결과적으로 global한 translation이 억제되게 되는 것임.

이 밖에 위 그림에 나타나 있는 것과 같이 다양한 외부 자극들에 의해서 eIF2α 에 인산화가 될 수 있으며 그 결과 global한 translation이 block되는 결과가 초래됨. (dsRNA의 존재에 의해 PKR이 활성화되어, UPR 혹은 hypoxia에 의해 PERK가 활성화되어, 아미노산 부족 혹은 UV에 의해 GCN2가 활성화되어 이런 결과가 초래될 수 있음)
그런데 무작정 모든 종류의 translation이 다 turn off되면 세포가 그냥 죽어버릴 것임. 따라서 세포는 위 그림에 나타난 것과 같은 외부 스트레스 자극을 받을 시 global한 translation은 turn off시키는 대신 ATF4와 같은 녀석의 translation은 turn on시켜줌. 이 때 ATF4는 stress 저항성 activity를 부여하는 단백질들을 encoding하는 gene들의 전사를 활성화시켜줄 수 있는 transcriptional factor임.
그렇다면 어떻게 이렇게 특정 mRNA의 translation만 촉진시키고 나머지는 다 block하는 것이 가능할까.

위 그림에는 ATF4 gene으로부터 만들어진 mRNA가 나타나 있음. 그런데 이 mRNA는 굉장히 다양한 AUG를 가져서 multiple site에서 translation이 initiation될 수 있음.
normal condition에서는 매우 많은 ternary complex들이 만들어지게 되고 그 결과 이상한 site로부터 translation이 시작되어서 정작 초록색으로 표시된 ATF4가 만들어지는 ORF(open reading frame)는 translation되지 못함. 한편 stress condition이어서 eIF2α가 인산화되어 있는 조건에서는 ternary complex의 수가 적고, 그 결과 여러 AUG로부터 만들어지는 ORF간의 경쟁이 적어져서 초록색 ORF가 비교적 잘 translation되게 됨. 그 결과 이런 기작에 의해 stress 조건 하에서 ATF4가 더 많이 만들어질 수 있음.

이렇게 만들어진 ATF4는 위와 같이 antioxidation, protein folding, protein processing, autophagy 등의 후속 반응들을 촉진시켜 stress 저항성 증대시키게 됨.
한편 이러한 stress 반응을 sensing하는 mechanism뿐만 아니라 세포 내의 energy가 충분한지 여부를 sensing하는 system도 필요함. 이런 system의 대표적인 예시가 mTORC1과 AMPK pathway임.
(사실 mTORC1은 노화와도 상당한 관련이 있는데 mTOR의 full name이 Mammalian target of rapamycin인 것을 보면 이를 알 수 있음. 현재 rapamycin은 노화를 지연 시킬 수 있을 것으로 기대되는 후보 약물 중 하나이므로 참고할 것)
mTORC1 내부에는 kinase 역할을 수행하는 mTOR kinase가 있는데, 이 녀석은 대표적으로 4E-BP1과 S6K1이라는 단백질을 인산화시킴.
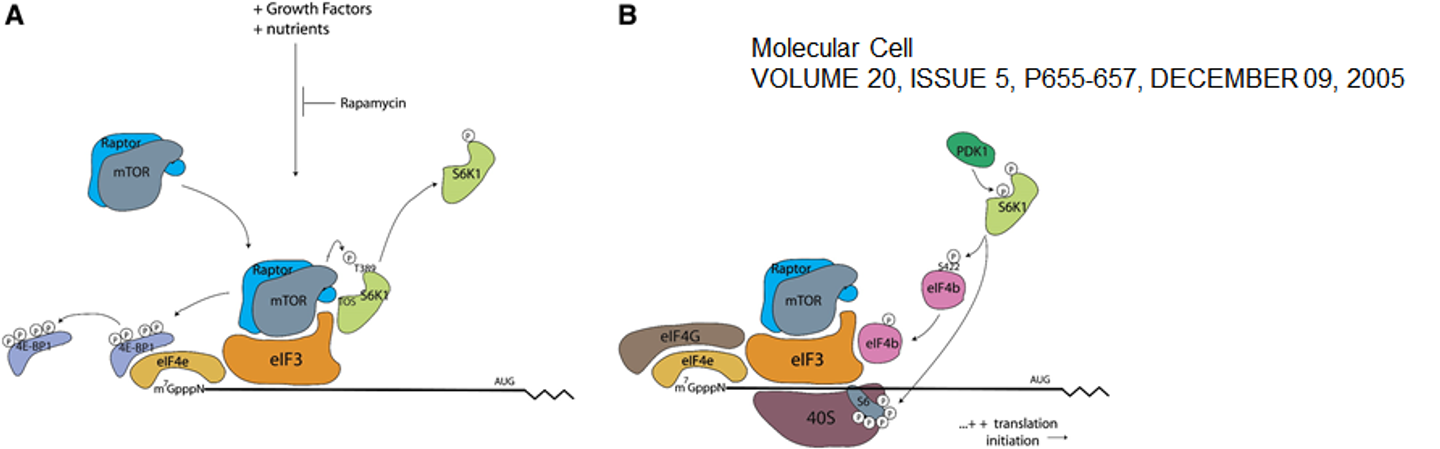
그 결과 translation initiation 측면에서 위 그림과 같은 조절이 일어나게 됨.
원래 4E-BP1은 eIF4E에 붙어서 eIF4E와 eIF4G(kind of scaffold protein)가 binding하지 못하게 block하는 역할을 하고 있음. 그런데 이 녀석이 mTOR kinase에 의해 인산화되게 되면 이 녀석이 떨어져 나가고, 결과적으로 eIF4E에 eIF4G가 결합할 수 있게 됨.
한편 S6K1도 원래는 eIF3에 결합해 있는 상태로 존재함. (일종의 translation initiation inhibitor) 그런데 S6K1이 mTOR kinase에 의해 인산화되게 되면 S6K1이 eIF3로부터 떨어져나가게 됨. 한편 떨어져 나간 S6K1에 PDK1이 작용해 하나의 인산기를 때주게 되면 비로소 S6K1이 kinase 활성을 가지게 되고 S6K1이 eIF4B를 인산화시킴. 결과적으로 인산화된 eIF4B가 initiation complex에 recruit되어서 translation initiation이 촉진되게 됨.
이 밖에 활성화된 S6K1은 PDCD4라는 단백질도 인산화시켜줄 수 있는데, 결국 이 인산화에 의해 PDCD4가 destruction되게 됨. 원래 PDCD4는 eIF4A inhibitor로 작용하는 녀석이었는데 이 녀석이 분해되므로 결과적으로 translation initiation이 촉진되게 됨.
다음 포스트에서는 남은 조절 기작들에 대해 이어서 알아보도록 하자.