이번 포스트부터는 단백질 번역에서의 신장 과정에 대해 알아보도록 하자.
기본적으로 elongation 과정은 bacteria와 eukaryote에서 매우 비슷함. (둘 다 뒤에서 다시 살펴볼 EF-T, EF-G factor가 중요하게 사용됨)
translation elongation에 대해 잘 모르던 과거에는 polypeptide가 N→C 방향으로 합성되는지, C→N 방향으로 합성되는지를 잘 몰랐고, ribosome이 RNA를 읽어나가는 방향도 몰랐으며, 어떻게 해서 mRNA로부터 단백질이 만들어지는지의 과정도 잘 몰랐었음.
그런데 연구를 통해 ribosome이 mRNA를 5'→3' 방향으로 읽어나가고, 이 과정에서 합성되는 단백질은 N→C방향으로 합성된다는 것을 알게 됨.

위 그림에 나타나 있는 것이 단백질이 어느 방향으로 합성되어 나가는지를 밝혀준 실험 과정임. 일단 위 그림 (a)와 같이 mRNA의 5'부터 3'까지 여러 ribosome이 붙어있게 되고, 결국 5'에 가까이 위치한 ribosome일수록 아직 단백질을 많이 합성하지 못한 상태, 3'에 가까이 위치한 ribosome일수록 단백질을 상대적으로 많이 합성한 상태일 것임.
이 때 특정 시점에 3H(중수소)로 labeling된 leucine을 첨가해봄. 그러면 labeled leucine이 tRNA에 충전된 결과 새로이 합성되는 단백질 내에 삽입될 것임. 그 결과가 (b)에 나타나 있음.
보면 이미 많이 합성이 진행된 단백질의 경우에는 끝부분에만 labeled leucine이 첨가된 반면 합성이 아직 거의 진행되지 않았던 단백질의 경우에는 전반적으로 다 labeled leucine이 첨가되어 있음.
이제 (b)에서 얻은 sample에 trypsin을 처리하여 peptide들만을 isolate하고, peptide positioning을 시켜보며 N terminal부터 C terminal 사이의 각각의 amino acid 영역에서 labeled leucine 함량이 어느정도인지를 전반적으로 조사해봄. 그 결과 위 그림 (c)와 같이 N terminal 부분에서는 labeled leucine이 거의 발견되지 않은 반면 C terminal 부분에서는 labeled leucine이 엄청나게 많이 발견됨.
이 실험 결과를 통해 우리는 단백질이 N→C방향으로 합성되어 나간다는 것을 알 수 있음. (C에 labeled leucine이 많았다는 것은 결국 (b)에서도 나타나 있듯이 C부위가 가장 나중에 합성되는 부위 중 하나라는 것을 의미함. 따라서 단백질의 합성 방향은 N→C라는 것을 알 수 있음)
한편 우리가 익히 알고 있는 것처럼 64(4 X 4 X 4)개의 codon이 20개의 amino acid를 지정함. 그렇다면 처음으로 3개의 염기가 모여 만들어지는 codon이라는 개념이 제안된 것은 언제일까.

처음으로 codon의 개념을 안한 논문이 위 그림 위쪽에 나타나 있으며, 이 중 두 저자가 각각 1962년, 2002년에 노벨생리의학상을 수상함.
이제 위 논문에 대해 조금 더 자세히 알아보자. 이 논문에서 사용한 것은 E. coli이며, 우리가 다양하게 조작해준 DNA를 bacteriophage를 통해 E. coli로 주입시킨 후 특정 단백질에 의한 phenotype이 나타나는지 여부를 파악함.

.
위 그림과 같이 연구자들은 염기 하나를 빼보기도 하고 넣어보기도 했으며, 두개, 세개씩을 넣거나 빼보기도 했음. 그 결과 frameshift mutation을 일으킨 경우에는(1 or 2개의 염기씩을 넣거나 뺀 경우) 정상 단백질에 의한 phenotype이 관찰되지 않은 반면 3의 배수로 mRNA를 넣거나 빼준 경우(- - - or + + +)에는 WT에서 나타나던 것과 유사한 단백질 활성이 관찰됨. 이를 통해 mRNA 중 3개가 단위로 묶여 단백질 합성에 관여한다는 사실을 알게 됨.
다시 말해 3개의 base가 1개의 amino acid를 지정한다는 것을 알게 되었으며, 이 때의 codon들이 overlapping되지는 않는다는 것을 확인함. (띄엄띄엄 deletion을 시켜준 경우에도 3의 배수개만큼 deletion시키기만 하면 WT에서와 유사한 단백질 활성이 관찰됨) 이 밖에 고정된 start point가 있으며, 그 start point 이후로는 건너뛰거나 쉬어가는 point 없이 연속적으로 3개씩의 codon에 의해 아미노산들이 recruit된다는 것을 알게 됨.
한편 이 때 본 논문의 저자들이 처음으로 codon들이 degeneration될 가능성에 대해 conclusion에 언급함. 즉, 여러 개의 codon들이 하나의 amino acid에 matching될 것으로 예상함. (64개의 codon들이 중복되어 20개의 amino acid와 matching될 것이라고 제안함)
이제 triplet code에 대해 어느정도 알게 되었으니 그 다음으로는 어떤 sequence가 어떤 amino acid에 대응되는지를 알아야 함.

이 때 수행한 역사적인 실험결과가 위와 같음. 보면 UUUUUU... 혹은 AAAAAA.... 혹은 CCCCCCC... 등을 넣어주는 한편, 이들과 함께 14C로 labeling된 phenylalanine을 넣어줌. 이후 단백질로 합성되어 축적되는 phenylalanine의 양을 관찰함. 그랬더니 결과적으로 polyuridylic acid를 넣어줬을 때 phenylalanine이 압도적으로 많이 만들어진다는 것을 알게 됨. 이를 통해 poly U가 어떤 식으로든 Phe를 지정하고 있다는 사실을 알게 됨.

한편 Khorana 등의 연구자들은 더 나아가서 poly UC, poly AG, poly UG, poly AC 등의 두 단위로 반복되는 polynucleotide와 poly UUG, poly AAG와 같이 세 단위로 반복되는 polynucleotide를 이용해 어떤 단백질들이 만들어지는지를 확인해봄.
결과적으로 두 단위로 반복되는 polynucleotide로부터는 두 종류의 amino acid들이 번갈아가며 나타나는 형태의 polypeptide가 만들어짐. 이를 통해 적어도 codon이 홀수개로 이루어진 base로 구성되어 있다는 사실을 알 수 있음. (codon이 짝수개의 염기로 구성되어 있었다면 한 종류의 amino acid밖에 나오지 않았을 것임)
다음으로 세 단위로 반복되는 polynucleotide로부터는 세 종류의 amino acid들 중 하나로 이루어진 homopolypeptide가 만들어짐. 이를 통해 codon이 3의 배수개의 base로 이루어져 있다는 것을 알 수 있음. (예를 들어 ABC가 반복되는 서열에서 ABC, BCA, CAB에 의해 서로 다른 amino acid가 만들어질 수 있어야 3종류의 amino acid가 나타날 것임)
결과적으로 이 실험을 통해서는 3, 9, 15, ...와 같은 갯수의 base들로 codon이 이루어질 것임을 알 수 있음. 이후 추가적인 연구를 통해 codon은 3개의 base로 이루어져 있다는 것을 확신하게 됨.
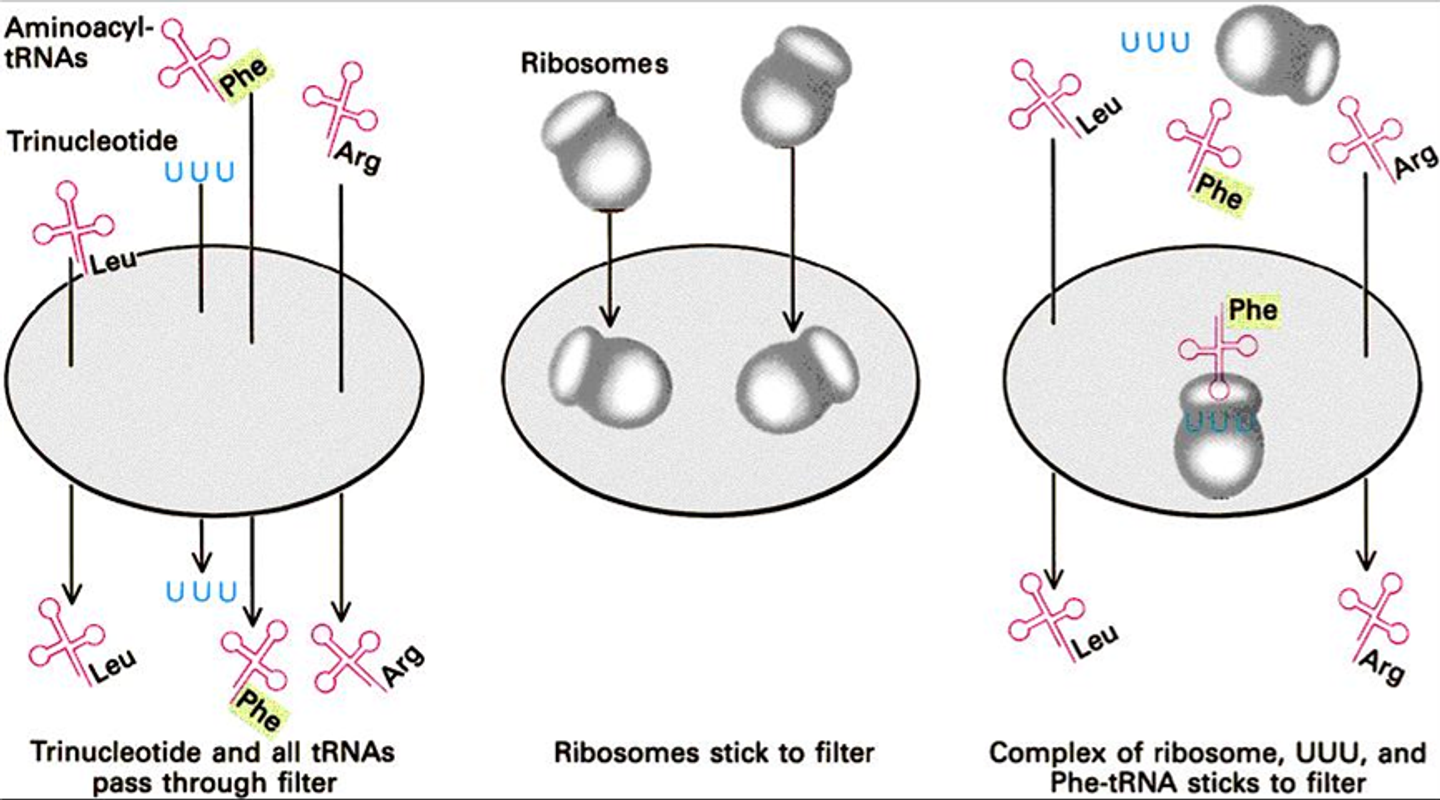
본격적으로 각각의 codon이 어떤 amino acid와 matching되는지를 밝혀낸 실험은 앞서 살펴봤던 filter binding assay임. 보면 특정 trinucleotide와 함께 다양한 amino acid가 충전된 tRNA를 넣어준 상태에서 ribosome과 함께 filter 위에 어떤 종류의 tRNA가 남아있는지를 알아보는 방식으로 실험이 수행됨. 그 결과 특정 codon에 의해 어떤 amino acid, 어떤 tRNA가 recruit되는지를 알 수 있었음.

한편 위 그림에 나타나 있는 실험 결과를 보면 codon들의 degeneration에 대해서도 알 수 있음. 이 경우에는 특히 lysine이 charge되어있는 tRNA가 어떤 trinucleotide와 결합하는지를 확인하는 assay를 수행하였고, 그 결과 lysine과 matching되는 codon으로 AAA 뿐만 아니라 AAG도 있다는 것을 알 수 있음.

이런 다양한 실험의 결과 위와 같이 64개의 codon이 각각 어떤 amino acid를 지정하는지와 관련된 genetic code를 알게 됨.

이에 대한 공로로 위 그림에도 나와있는 것처럼 1964년에 노벨 생리의학상이 수여됨.
한편 앞서 degeneracy에 대한 내용이 등장했었음. 그러면 도대체 degeneracy는 어떻게 해서 발생하는 것일까. 이는 Wobble hypothesis로 설명됨. 간단히 설명하자면 codon의 3번째 염기(3' 부근)와 anticodon의 1번째 염기(5' 부근) 간에는 G-C, A-T pair 뿐 아니라 그 밖의 wobble base pair도 허용된다는 내용임.

위 그림에 wobble base pair의 대표적인 예가 나타나 있음. 보면 A와 U 뿐만 아니라 G와 U간에도 수소결합이 형성될 수 있고 I와 A 사이에도 마찬가지로 수소결합이 형성될 수 있음.

그 결과 위 그림과 같이 anticodon의 5' 부근 염기와 codon의 3' 부근 염기 사이에 watson-crick base pair 뿐 아니라 wobble base pair도 가능해져서 결과적으로 한 amino acid가 charging된 tRNA가 여러 codon에 와서 결합하는 것이 가능해짐.
앞서 universal한 genetic code들이 나타나 있는 표를 첨부했었음. 그런데 그 표에 나타난 matching이 항상 옳은 것은 아님. 즉, system별로 예외적인 match가 관찰되는 경우도 있음.

대표적인 예시가 위 표에 나타나 있으므로 참고할 것.
다음 포스트에서는 이제 본격적인 translation elongation cycle에 대해 알아보도록 하자.