이번 포스트부터는 본격적으로 번역 신장 과정에 대해 자세히 알아보도록 하자.
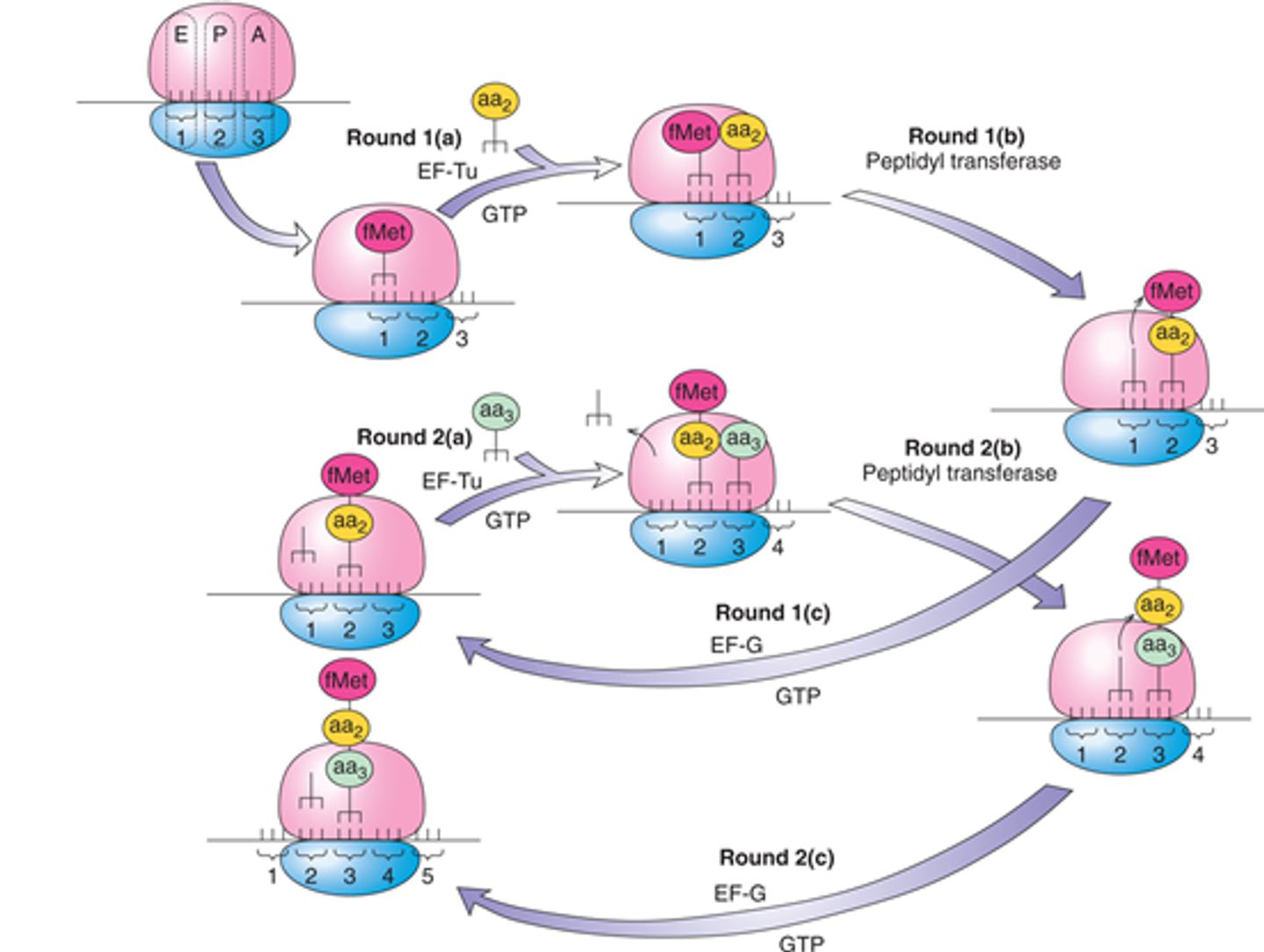
전체적인 elongation cycle 모식도는 위와 같음. elongation cycle은 크게 3개의 step으로 나누어지는데, 아래와 같음.
1. EF-Tu(with GTP)에 의해 aminoacyl-tRNA가 ribosome의 A site에 binding
2. ribosome 내 RNA를 center로 해서 구성되는 peptidyl transferase에 의해 P site에 있는 peptide와 A site에 새로 들어온 aminoacyl-tRNA의 amino acid 사이에 peptide bond가 형성 (ribosome 내 protein들은 대다수 ribosome의 구조 유지 자체에만 도움을 줌)
3. EF-G(with GTP)에 의해 peptidyl-tRNA와 codon을 포함하는 전체 ribosome frame의 translocate 발생. (이후 deacylated tRNA는 떨어져나감)
이 때 2번에 등장한 peptidyl transferase 활성이 도대체 어떻게 rRNA로부터 생기는지에 대해 궁금할 수 있음. 이와 관련된 그림이 아래에 나타나 있음.

위 그림 중 C에 특히 주목해보자. 보면 23S rRNA 중 2451번 A의 2' 위치 OH가 P site에 있는 tRNA 발단부의 A부분 2' 위치 OH를 공격하는 반응을 시작으로, A site에 있는 tRNA와 붙어있는 amino acid의 N 부분이 P site에 있는 tRNA와 붙어있는 peptide 중 C terminal 부분의 C를 공격해서 결국 이 둘이 결합하게 됨.
이런 식의 반응 mechanism을 가지고 있기 때문에 실제로 23s rRNA의 2451번 A의 2' 부위를 그냥 H로 바꿔버리게 되면 ribosome에서의 peptidyl transferase 활성이 관찰되지 않음.
그렇다면 ribosome에 A, P, E와 같은 site들이 있다는 사실은 어떻게 알게 되었을까.
이를 알아내기 위해 사용한 항생제의 일종이 바로 puromycin임. puromycin의 구조는 아래 그림에 나타나 있음.

위 그림 (a)의 오른쪽 구조가 puromycin임. 보면 puromycin이 위 그림 왼쪽에 나타난 것과 같은 tyrosyl-tRNA와 매우 유사하다는 것을 알 수 있음. (참고로 이 때 tyrosyl-tRNA의 위쪽에 나타난 구조는 tRNA의 말단부인 A 구조임)
결국 puromycin의 맨 아래 NH2는 tRNA에 charging되어있는 peptide를 공격할 수 있어서 ribosome 상에서 새로 합성되는 peptide 내에 puromycin이 포함될 수 있음. 동시에, 그림 (b)에 나타나 있는 것처럼 C 말단부분에 puromycin이 포함되어버리면 더 이상 단백질 합성이 계속되지 않고 ribosome 밖으로 방출되어버림. (즉, puromycin은 아미노산이 충전된 tRNA의 analog임) 이 puromycin을 이용해 설계된 실험이 아래에 나타나 있음.
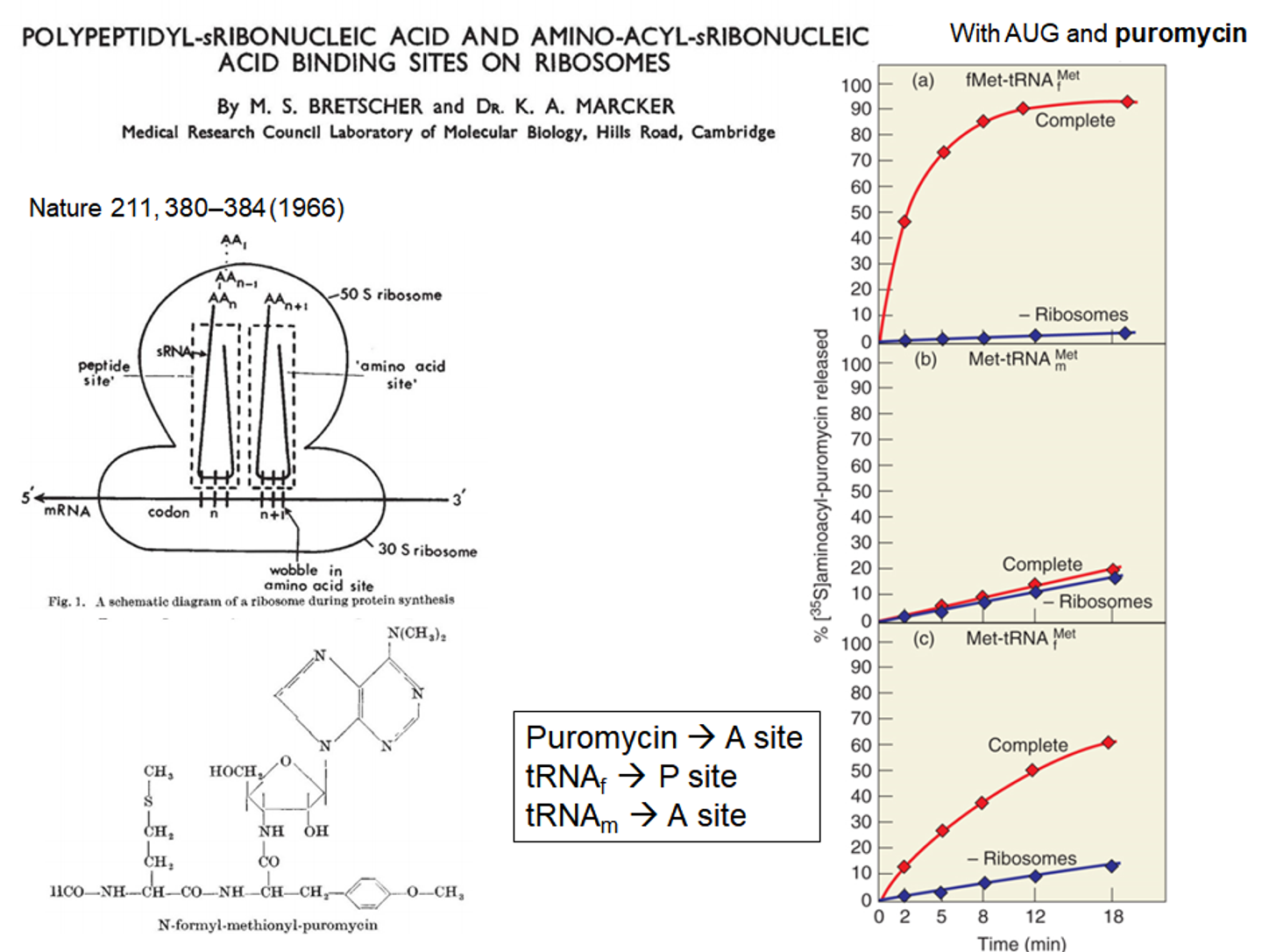
이 실험에서는 특히 AUG에 의해 recruit되는 Met에 이어 곧바로 puromycin이 결합된 후 위 그림 왼쪽 아래와 같은 생성물이 만들어진 채로 ribosome에서 방출되는 system을 구성함.
이제 본격적으로 위 그림 오른쪽에 나타나 있는 결과를 살펴보자. 우선 (a), (b), (c) 모두 y축은 aminoacyl-puromycin이 release되는 정도를 나타내주고 있으며, x축은 time을 나타내주고 있음. 이 때 각각의 실험마다 모든 contents를 다 넣어준 setting과 ribosome을 넣어주지 않은 setting에 대해서 결과가 어떻게 달라지는지를 비교함.
우선 (a)부터 살펴보자. 이 실험시 사용된 ribosome은 원핵생물로부터 유래한 녀석이므로 결국 ribosome에 의해 translation이 일어나려면 fMet-tRNA가 필요함. 따라서 (a)에서는 일단 fMet-tRNA를 넣어줌. 그랬더니 ribosome이 없을 때에 비해 ribosome이 있을 때 압도적으로 많은 양의 단백질 생성물이 만들어져 나옴.
다음으로 (b)를 살펴보자. 이 경우에는 Met-tRNA를 넣어줌. 그럴 시 그냥 Met-tRNA가 ribosome의 A site를 차지하게 되고, 결과적으로 A site에 puromycin이 binding되지 못함. 이 때문에 ribosome이 있으나 없으나 동일한 결과가 나타남.
마지막으로 (c)를 살펴보자. 이 경우에는 Met이 충전된 tRNA이기는 하나 이 때의 tRNA가 initiator tRNA임. (즉, initiator tRNA에 Met을 인위적으로 붙여준 것) 이런 setting 하에서는 (a)와 유사하게 ribosome이 없는 경우에 비해 ribosome이 있을 때 단백질 생성물이 훨씬 더 많이 만들어져 나옴.
이 실험결과를 통해 결국 ribosome 중 puromycin이 결합하는 자리와 initiator tRNA가 결합하는 자리는 서로 다르다는 것을 알 수 있으며, 한편 initiator tRNA와 그냥 tRNA가 결합하는 자리 또한 다르다는 것을 알 수 있음. (특히 (b) 결과를 통해 puromycin과 그냥 tRNA가 결합하는 자리가 동일하다는 것도 추측할 수 있음) 실제로는 우리가 익히 알고 있다시피 puromycin, 혹은 일반적인 tRNA가 들어오는 자리는 A site, initiator tRNA가 들어오는 자리는 P site임.
앞서의 실험은 ribosome 내에 적어도 두 개의 site가 존재할 것임을 알려줬다면 아래에 나타나 있는 실험은 ribosome 내에 tRNA가 결합할 수 있는 site가 3개 존재한다는 것을 확인시켜줌.

이 실험에서는 크게 3가지 tRNA를 사용해주었으며, 각각의 tRNA를 서서히 많이 넣어줌과 동시에 과량의 mRNA를 넣어줌. (특히 본 실험에서는 mRNA 서열 중 poly U를 사용했고, 이에 맞는 Phe-tRNA를 사용함)
우선 poly U mRNA와 함께 acetyl-Phe-tRNA를 서서히 많이 넣어줬을 때의 결과를 살펴보자. 우선 이 때 넣어준 acetyl-Phe-tRNA는 아래와 같이 생김.

이 녀석은 peptydyl-tRNA analog로 기능함. 다만 이 녀석이 들어갈 시 더 이상 ribosome에 tRNA가 추가되며 후속 translation 반응이 일어나지는 않음. 이런 상태에서 ribosome 1개당 acetyl-Phe-tRNA가 몇 개 결합하는지(stoichiometry)를 확인해본 결과 위 그림 왼쪽 위에서 나타난 것과 같이 1개라는 것을 알게 됨. 이러한 결과가 나온 이유는 acetyl-Phe-tRNA가 ribosome의 P 혹은 A 중 한 곳에서만 결합한 채로 남아있기 때문임.
다음으로 poly U mRNA와 함께 Phe-tRNA를 서서히 많이 넣어줬을 때의 결과를 살펴보자. 이 때의 stoichiometry를 확인해 본 결과가 위 그림 왼쪽 아래에 나타나 있으며, 결과적으로 ribosome 1개당 2개의 Phe-tRNA가 결합한다는 것을 알게 됨. 이런 결과가 나온 이유는 Phe-tRNA가 P site와 A site에 동시에 결합하기 때문임. (이 때 E site에 의한 effect가 나타나지 않은 이유는 E site에 Phe-tRNA가 아닌 그냥 tRNA가 들어가기 때문임)
마지막으로 poly U mRNA와 함께 그냥 tRNA(Phe를 충전할 수 있는 녀석)를 넣어준 경우를 살펴보자. 이 때의 stoichiometry를 확인해 본 결과가 위 그림 오른쪽에 나타나 있으며, 결과적으로 ribosome 1개당 3개의 tRNA가 결합한다는 것을 알게 됨. 이러한 결과가 나온 이유는 그냥 tRNA가 E site, P site, A site에 동시에 결합할 수 있기 때문임.
이 실험을 통해 결과적으로 ribosome 내에 tRNA가 결합 가능한 서로 다른 site가 3개 존재하고 있다는 것을 알게 됨. 그리고 추가적인 실험을 통해 E site에는 deacylated tRNA가 위치하게 되고 결과적으로 E site에 잠시 머물던 deacylated tRNA가 밖으로 release된다는 것을 알게 됨.
참고로 이 때 E site, P site, A site 각각은 exit site, peptidyl site, aminoacyl site를 의미함.

위 그림은 translation elongation cycle에서 어떤 protein factor들이 작용하는지를 나타내주고 있음. 이 때 중점적으로 살펴봐야 할 factor들은 크게 EF-T와 EG-G이며 뒤이어 이들 factor들의 역할에 대해 자세히 알아볼 것임.
이 그림에서는 EF-G와 EF-T의 ribosome에의 binding site가 유사하다는 것 정도를 눈여겨볼 필요가 있음. 그 밖에 위 그림에 전반적으로 나타나 있는 cycle에 대해서도 어느 정도는 알아둘 필요가 있는데, 처음에는 P site에 initiator tRNA만 붙어있는 상태에서 일반적인 aa-tRNA와 함께 EF-Tu-GTP가 같이 cooperated된 상태로 이 큰 덩어리가 ribosome의 A site에 들어오게 됨. 이후 GTP hydrolysis가 일어나면서 EF-Tu-GDP가 방출됨.
그러고 나면 ribosome 내의 RNA에 의해 peptidyl transfer가 일어남. 그 이후에는 EF-G-GTP가 들어오면서 앞서 EF-Tu가 위치하던 곳과 비슷한 ribosome 위치에 결합함. 이후 EF-G-GTP 내의 GTP가 분해되면서 EF-G-GDP가 ribosome에서 release됨과 동시에 ribosome의 translocation이 일어남. 그렇게 해서 E site로 이동한 E-RNA는 release되게 되고, 이후 앞서와 동일한 일이 반복됨.
(참고로 위 그림상에서 각각의 factor에 붙어있던 GTP가 GDP로 바뀌는 과정이 나타나 있는데, 이 과정은 기본적으로 factor 자체가 가지고 있는 활성에 의해 일어나기는 하지만, 이 활성이 ribosome에 결합했을 때에만 turn on된다는 것을 기억해둘 필요가 있음)
다음 포스트에서는 이제 각 elongation의 과정에 대해 조금 더 상세하게 알아보도록 하자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 18.2 : 번역 신장 (translation elongation) - 2nd step (0) | 2024.08.09 |
|---|---|
| [분자생물학] 18.2 : 번역 신장 (translation elongation) - 1st step (0) | 2024.08.09 |
| [분자생물학] 18.1 : 번역 신장 (translation elongation) - 1 (0) | 2024.08.09 |
| [분자생물학] 17.3 : 진핵생물에서의 번역 개시 조절 (eukaryotic translation regulation) - 3 (0) | 2024.08.09 |
| [분자생물학] 17.3 : 진핵생물에서의 번역 개시 조절 (eukaryotic translation regulation) - 2 (0) | 2024.08.09 |