이번 포스트에서는 원핵생물에서의 repressor 단백질과 DNA 간의 상호작용에 대해 알아보도록 하자.

DNA와 결합하는 단백질들은 위 그림 (a)에 나타난 것과 같이 거의 대부분 공통적으로 helix-turn-helix motif를 가지고 있음.
이 중에서도 위 그림에서 나타난 빨간색 helix가 DNA의 major groove를 인식해 결합하게 되는데, 따라서 이 빨간 helix를 recognition helix라고 부름.
그러므로 helix-turn-helix motif를 통해 DNA와 결합하는 단백질들의 경우 recognition helix의 amino acid sequence와, 특정 DNA의 base sequence들의 조합에 의해 결합이 이루어지고, 이 때 amino acid와 base끼리의 상호작용 정도에 따라 specificity가 결정됨.
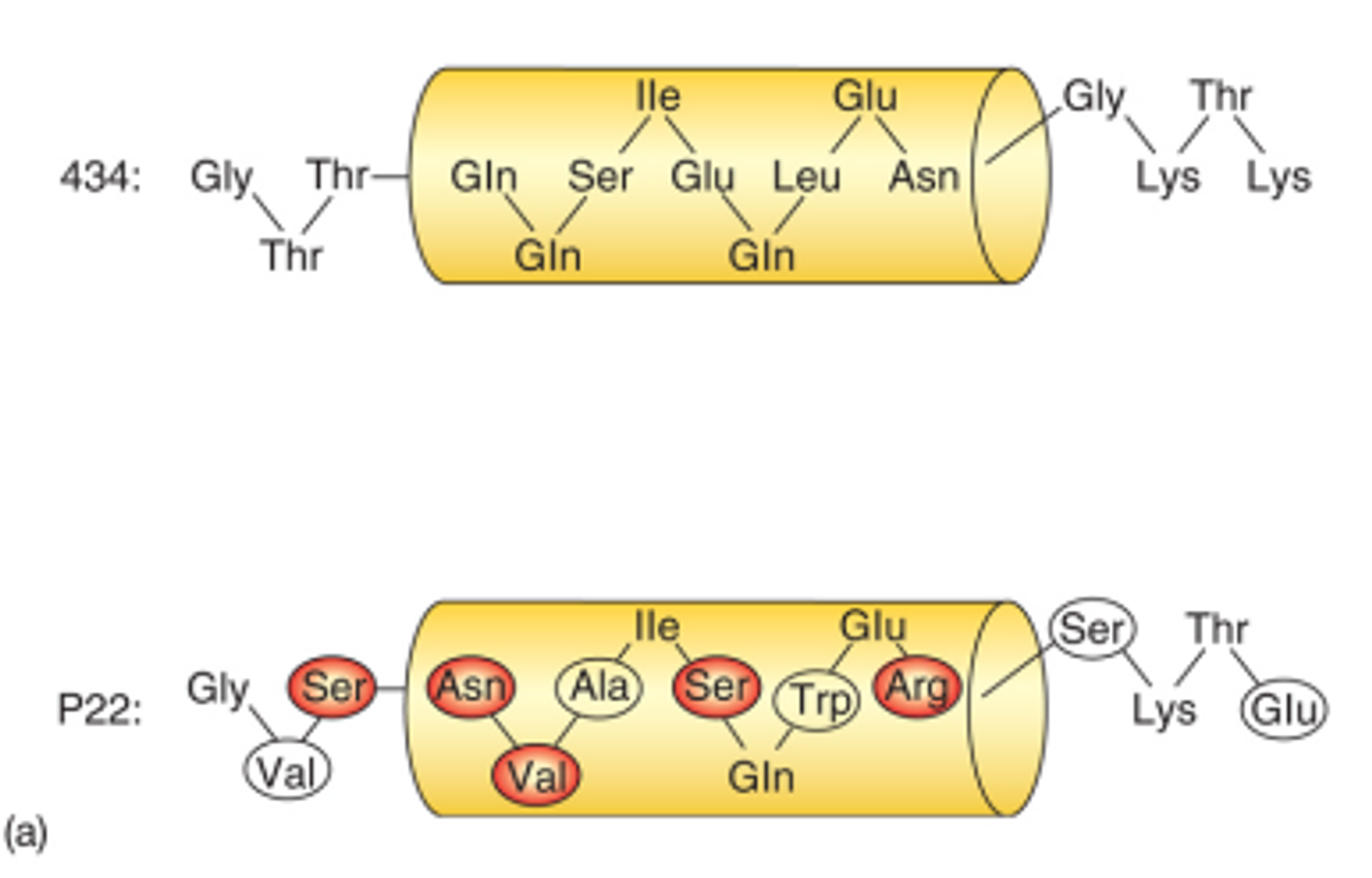
위 그림은 대표적으로 발견되는 2 종류의 repressor(434와 P22)를 나타내주고 있음. 이 때 노란색으로 표현된 부분이 recognition helix임.
그 중 P22에 대해 조금 더 자세히 알아보자. 이 녀석의 경우 위 그림상에 나타나 있는 특정 amino acid가 DNA와의 binding에 특히나 핵심적인 역할을 수행함. 이 때 실제로는 helix 구조를 이루고 있으므로 이렇게 옆으로 보는 것보다 위에서 내려다보는 것이 구조적 이해를 하는데 더 용이할 것임.

위 그림이 동일한 P22의 recognition helix를 위에서 바라본 것임. 이 때 붉은색으로 표시된 N, S, R, V가 앞서 (a)에서 붉게 표시되었던 부분과 동일한 부분인데, 보면 왼쪽에 다 몰려있는 것을 알 수 있음. 따라서 이 왼쪽 부분이 DNA와 binding하는 핵심적인 amino acid라는 것을 알 수 있음. (즉, 이 부분이 P22의 specificity를 결정)
한편, 정말 key amino acid(붉은색)가 DNA binding에 있어 핵심적인 역할을 수행하는지 알아보기 위해, 434 protein에 mutagenesis를 일으켜 딱 P22의 key amino acid 부분의 amino acid만 P22의 것으로 바꿔봄. (즉, 나머지 부분은 다 동일한데 P22의 key amino acid 자리만 딱 N, S, R, V로 바꿔봄) 이후 변형된 434를 P22가 붙는 DNA와 함께 첨가해 DNA footprinting을 진행해봄.
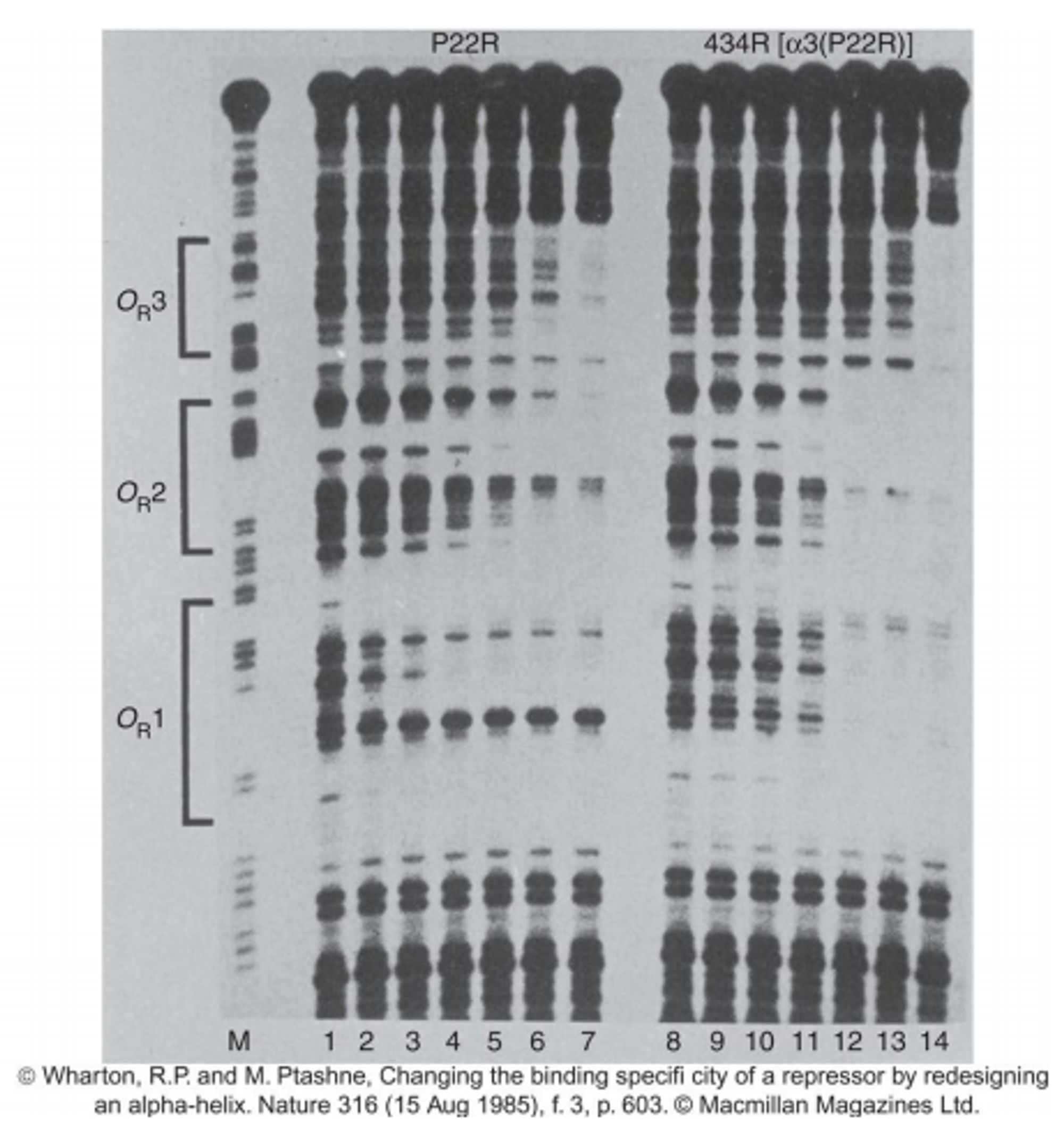
그랬더니 그 결과가 위와 같이 나옴. 이 때 왼쪽 P22R에 해당하는 band들은 그냥 P22를 서서히 많이 넣어주었을 때의 DNA footprinting 결과임. 보면 P22의 농도가 증가함에 따라 점차 OR1, OR2, OR3 부분에 해당하는 band가 흐려짐을 알 수 있음. 즉, 이 부분에 P22가 결합하는 것.
한편 위 그림 오른쪽에는 앞서 말한 방법으로 mutagenesis시킨 434 repressor를 가지고 동일한 실험을 수행한 결과임. 이 때 P22 repressor를 서서히 많이 처리했을 때와 거의 동일한 결과가 나타남을 알 수 있음. 즉, 이를 통해 key amino acid만 P22의 것으로 치환해도 P22가 붙는 DNA자리에 동일하게 붙을 수 있음을 알 수 있음. (따라서 key amino acid가 DNA-repressor binding의 specificity를 결정함)
한편 이전 chapter에서 배운 lambda repressor의 경우 amino-terminal arm으로 이루어진 extra motif를 추가적으로 가지며, 이 motif가 DNA를 감싸고 있음.
이 때 이전에 언급한 것처럼 lambda repressor는 OR, 그 중에서도 OR1, OR2에 더 affinity가 강하고(물론 양이 너무 많을 시 OR3에도 붙을수는 있음) Cro는 OR3에 더 affinity가 강함. 따라서 Cro와 lambda repressor는 기본적으로 동일한 operator인 OR에 affinity를 가지지만, 세부적으로 보면 조금 다른 microspecificity를 가짐.
이렇듯 조금 다른 microspecificity를 가질 수 있는 것은, recognition helix 내 amino acid의 미세한 차이로 인해 결합 가능한 DNA와의 affinity가 달라지는 현상이 일어나기 때문임.

위 그림은 lambda repressor와 operator가 붙는 것을 모식적으로 보여주고 있음. 이 때 1~5까지의 덩어리가 lambda repressor monomer이며, 앞서 본 것처럼 실제로는 lambda repressor 2개가 위 그림과 같이 dimer를 이루어서 작용하게 됨.
이 때 3, 3'에 해당하는(붉은색) 부분이 recognition helix임. 보면 3, 3'이 operator에 해당하는 major groove와의 binding에 핵심적인 역할을 하고 있음. 한편 5와 5'은 서로서로 붙게 되는데, 따라서 이 부분은 protein-protein interaction을 유발하는 helix에 해당함.
참고로 위 그림을 자세히 보면 lambda repressor dimer가 결합함에 따라 DNA가 미세하게 휘는 것도 관찰 가능함.
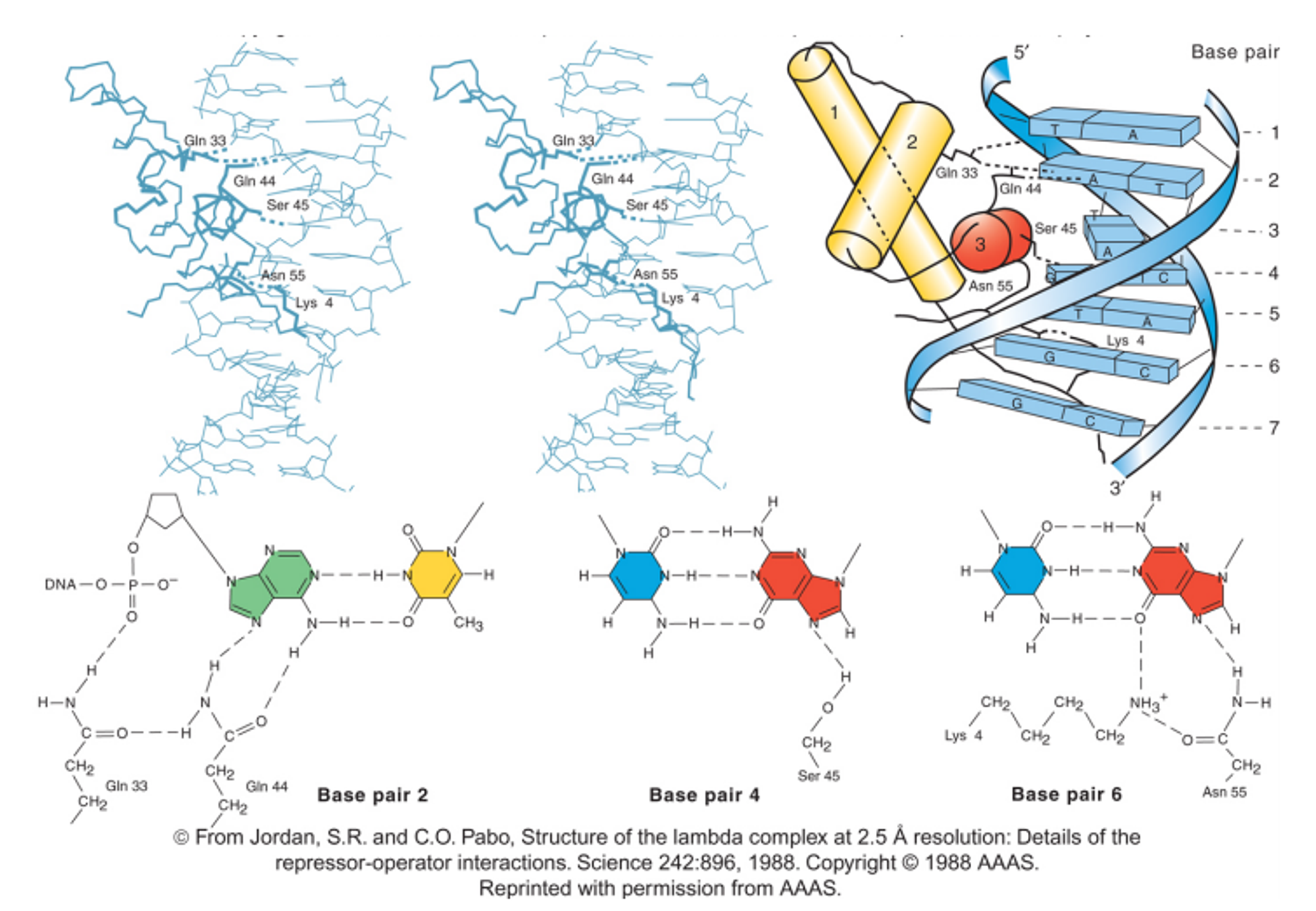
이제 조금 더 high-resolution에서 살펴보자. 위 그림을 보면 어떤 amino acid가 어떤 base와 상호작용하는지에 대해 잘 알 수 있음.
이 때 위 그림에 나타난 것처럼 특히 Gln, Gln, Ser, Asn이 DNA와의 binding에 있어 매우 핵심적으로 작용함. 조금 더 구체적으로 표현하자면 위 그림 아래에 나와있는 것처럼 특정 base들과 Gln, Gln, Ser, Asn이 H-bond(수소결합)를 형성함. 이 때 Gln, Gln, Ser, Asn와 수소결합을 형성하는 염기들도 정해져 있는데, 위 그림 상에서는 1, 2, 3, 4, 5, 6, 7의 염기 중 2, 4, 6의 염기들이 Gln, Gln, Ser, Asn과 H-bond로 결합함.
한편 이런 H-bond는 amino acid와 DNA backbone과의 electrostatic attraction을 극대화하는 데 큰 역할을 함.

위 그림에서는 Lys과 같은 positive charge amino acid가 나타나 있으며, 이 밖에도 Arg, His과 같은 + charge를 띄는 amino acid들이 DNA의 backbone과 electrostatic attraction을 형성함. (DNA backbone은 인산기에 의해 - charge를 띄므로 이는 당연한 현상) 이 때 앞서 설명한 H-bond에 의해 이 electrostatic attraction이 더 잘 일어날 수 있게끔 모양이 최적화됨.
이런 방식을 사용해서 DNA와 protein이 단단히 결합할 수 있게 되는 것임.
사실 이러한 DNA-protein간의 구조적인 결합은 X-ray crystallography를 이용해 시각화할 수 있음.

이 결과를 이용하면 H bonding site, van der Walls contact site(in the operator and in the repressor)등을 구조적으로 특정하는 것이 가능해짐. (위 그림과 같이 퍼즐 맞추듯 모양이 딱 맞아들어가는 각도를 찾게 되면 DNA와 protein이 binding하는 부분을 1:1로 matching할 수 있게 됨) 추가적으로 (b)를 보면 실제로 DNA가 bending 하고 있어서 (c)와 같이 protein과 잘 결합할 수 있도록 구조를 형성하고 있다는 사실 또한 알 수 있음.
이런 식으로 구조생물학적 실험기법을 통해 얻은 지식들은 genetic test를 통해 한번 더 교차검증할 수 있음. (그리고 사실 이 것이 일반적인 수순임. 이 때문에 구조생물학자들과 유전학자들이 협업하는 경우가 꽤나 흔함) 예를 들어 구조생물학적으로 DNA operator의 특정 염기에 결합하는 것으로 밝혀진 amino acid residue가 있다고 하자. 그러면 유전학자들은 mutagenesis를 통해 이 residue를 변형시켜서 그 특정 염기와의 결합이 잘 안일어나는 쪽으로 phenotype이 바뀌는지를 보게 됨. 만약 특정 염기와의 결합이 잘 안일어나거나 DNA-protein 결합력이 떨어진다면, 구조생물학적으로 예상한 것이 정말 사실이라는 것을 확실히 confirm할 수 있게 됨.
다음 포스트에서는 원거리에 떨어져있는 DNA, 단백질들이 어떻게 상호작용할 수 있는지에 대해 알아보자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 10.1 : 진핵생물의 RNA polymerase - 1 (0) | 2023.12.16 |
|---|---|
| [분자생물학] 9.2 : prokaryote에서의 원거리 DNA, 단백질 상호작용 (0) | 2023.12.16 |
| [분자생물학] 8.3 : lambda phage(람다 파지), antitermination - 3 (0) | 2023.12.16 |
| [분자생물학] 8.3 : lambda phage(람다 파지), antitermination - 2 (0) | 2023.12.16 |
| [분자생물학] 8.3 : lambda phage(람다 파지), antitermination - 1 (0) | 2023.12.16 |