이번 포스트에서는 전사체(transcript, RNA)를 분석할 때 사용 가능한 각종 분석 방법들에 대해 알아보자.
nothern blot (노던 블랏, 노던 블롯, 노던 블로팅)
우선 nothern blot에 대해 알아보자.
이 방식은 서던 블롯과 매우 유사하지만 실험 대상이 RNA라는 점만 다름.
우선 다양한 조직들로부터 RNA를 얻은 후 RNA를 gel에 loading한 다음 서던 블롯에서와 동일한 방법으로 membrane으로 옮겨줌. 이후 우리가 관찰하고자 하는 RNA가 어느 정도 존재하는지를 보아야 하므로 probe(탐침)를 써야 하는데, 이 때는 일반적으로 우리가 관심있는 RNA에 상보적으로 결합할 수 있는 cDNA를 probe로 사용함.

이후 autoradiograph 등을 이용하면 위 그림과 같은 데이터를 얻을 수 있음. 이 때 band의 두께가 두꺼울수록 발현되는 RNA의 양이 더 많음을 의미하게 되며, band의 두께는 앞서와 마찬가지로 densitometer를 이용해 측정 가능함.
S1 mapping
다음으로 S1 mapping에 대해 알아보자. S1 mapping은 유전자에서 전사가 시작되는 부위, 혹은 전사가 끝나는 부위가 어디인지를 확인하고자 할 때 아주 유용하게 사용될 수 있음.
이 때 'S1'이라는 이름은 mapping과정에서 S1 nuclease(single strand nucleotide를 잘라주는 효소)를 사용하기에 붙은 것임.
S1 mapping에는 크게 두 가지 종류가 있음.
첫 번째 종류는 5' end S1 mapping임.
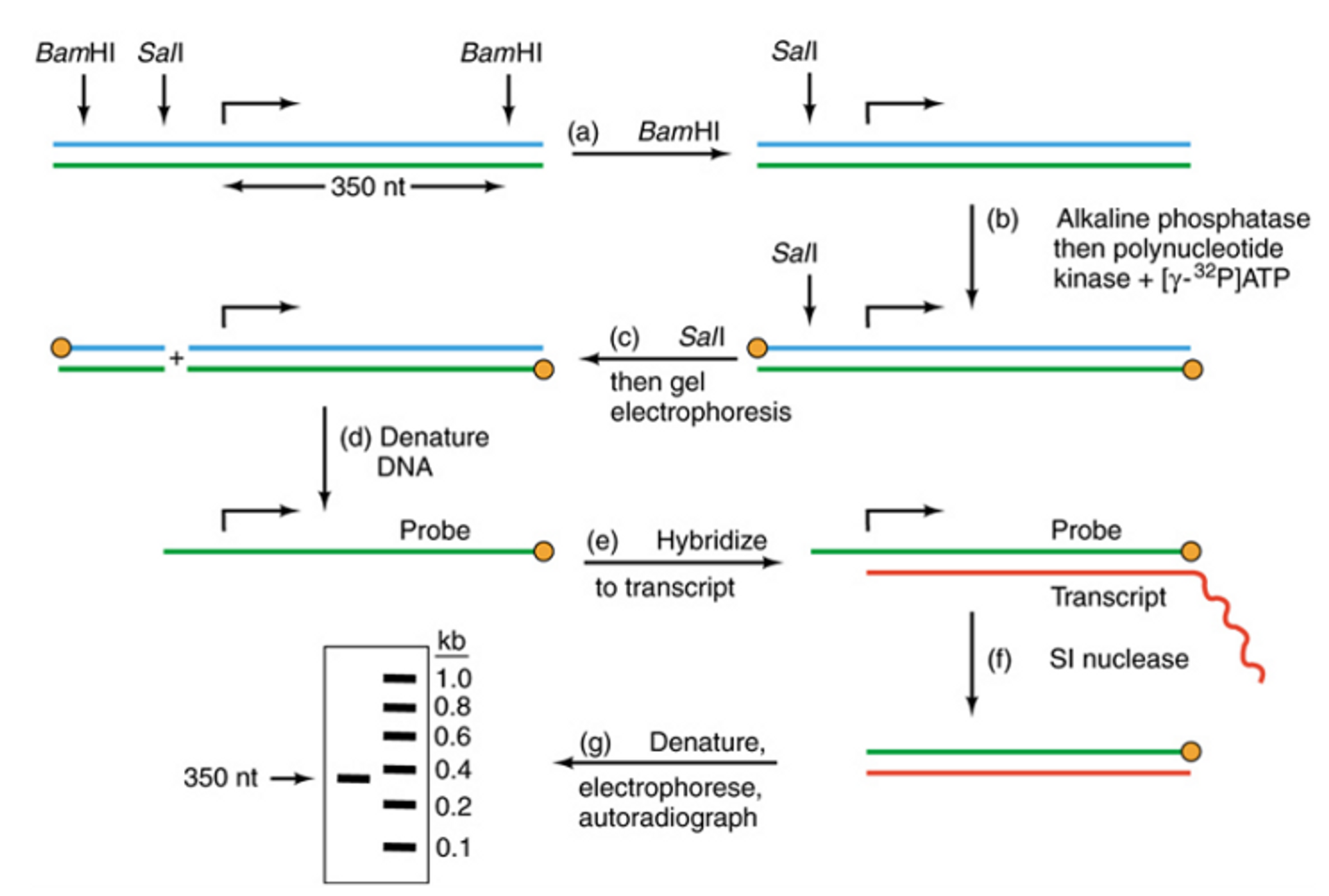
위 그림에 5' end S1 mapping의 과정이 나타나 있음. 하나하나 살펴보자.
(참고로 처음 4개 단계는 모두 probe를 만들기 위한 단계임) 우선 맨 처음 그림에 DNA 전사의 시작 지점이 대략적으로 나타나 있음. 그러나 이 위치에 대해 정확히는 모르는 상태임. 한편 주변에 위치한 제한효소 절단부위의 위치는 정확히 알고 있음.
이런 상황에서 우선 BamH1과 같은 제한효소로 절단을 해줌. (a)
다음으로 DNA fragment의 5' 부위를 labeling해주어야 하는데, 이를 위해 (b)와 같은 과정을 거침. 이 때 alkaline phosphatase를 처리해주는 이유는 이미 5' 맨 끝에 붙어있던 인산기를 때주기 위해서이고 이후 polynucleotide kinase와 함께 [gamma -32P] NTP를 넣어주는 이유는 5' 끝을 다시 인산화시켜주는 과정에서 동위원소로 표지하기 위해서임.
이후 Sal1과 같은 제한효로 다시 DNA를 절단시켜준 후 denature 시켜주면 DNA probe를 만들 수 있음. (이 때 제한효소에 의해 대략적인 전사 시작부위의 일부분이 잘리지 않게끔 제한효소를 잘 골라줘야 함)
이후 이렇게 만들어진 probe를 우리가 관심있게 보고자 하는 transcript와 hybridize시켜주게 됨. 다음으로 S1 nuclease로 single strand(단일가닥)로 남아있는 녀석들을 다 분해시켜주게 되면 probe와 결합해 이중가닥을 이룬 transcript의 일부 영역만 분해되지 않고 남아있게 됨.
이 녀석을 전기영동시켜주게 되면 위 그림 왼쪽 아래와 같은 결과를 얻을 수 있음. 이 때 이 녀석의 크기가 350 nucleotide이므로, 결과적으로 transcription의 starting point는 BamH1에 의해 절단되는 절단부위로부터 350bp만큼 위쪽에 있음을 알 수 있음.
다음으로 살펴볼 종류는 3' end S1 mapping임.
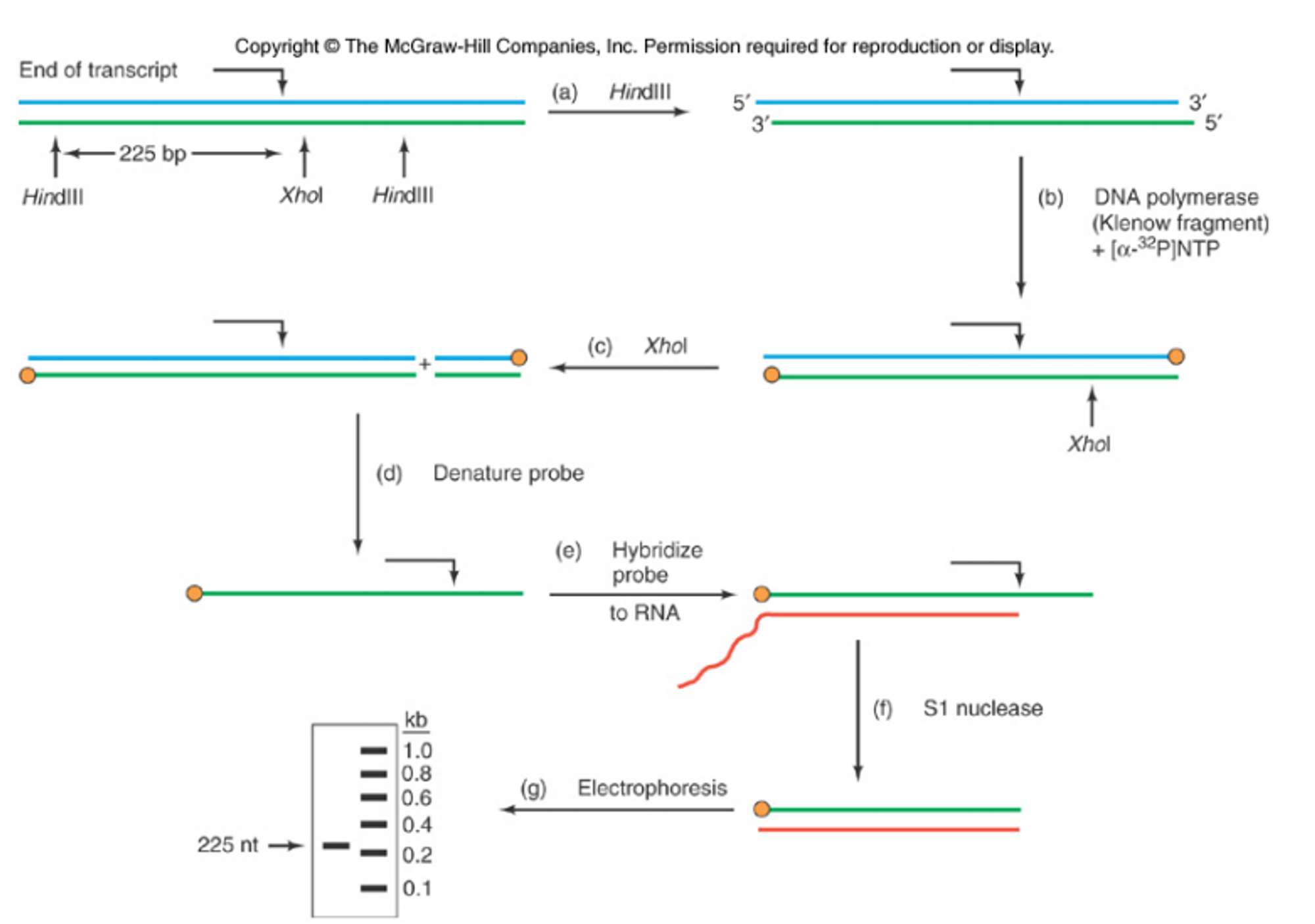
위 방법도 앞서와 거의 동일하나, 이 경우 transcript의 end point(끝점)를 파악하기 위해 사용됨. (이 경우에는 대략적인 end point보다 위쪽에 있는 잘 알려진 제한효소절단부위를 기준으로 probe를 제작하게 됨)
나머지 과정들은 거의 동일하나 한 가지 다른 점이 있는데, 3' end를 labeling하는 과정이 그것임. 이 경우 DNA polymerase와 함께 [alpha-32P] NTP를 사용하면 됨. (중합효소에 의해 중합이 진행될 때는 NTP의 gamma, beta phosphate가 떨어지고 난 상태의 NMP가 결합하기 때문에 alpha-32P를 사용해 3'에 동위원소 표지된 NTP를 첨가해줄 수 있는 것임)
위 그림에서 나타난 예시에서는 결과적으로 HindIII 제한효소 절단부위보다 225bp 아래에 end point가 존재함을 확인함.
primer extension
primer extension은 S1 mapping보다도 더 정확하게 전사시작부위를 알고자 할 때 사용 가능한 방법임. (이 방법을 이용하면 전사시작부위를 단일 뉴클레오타이드 단위로 알아낼 수 있음)
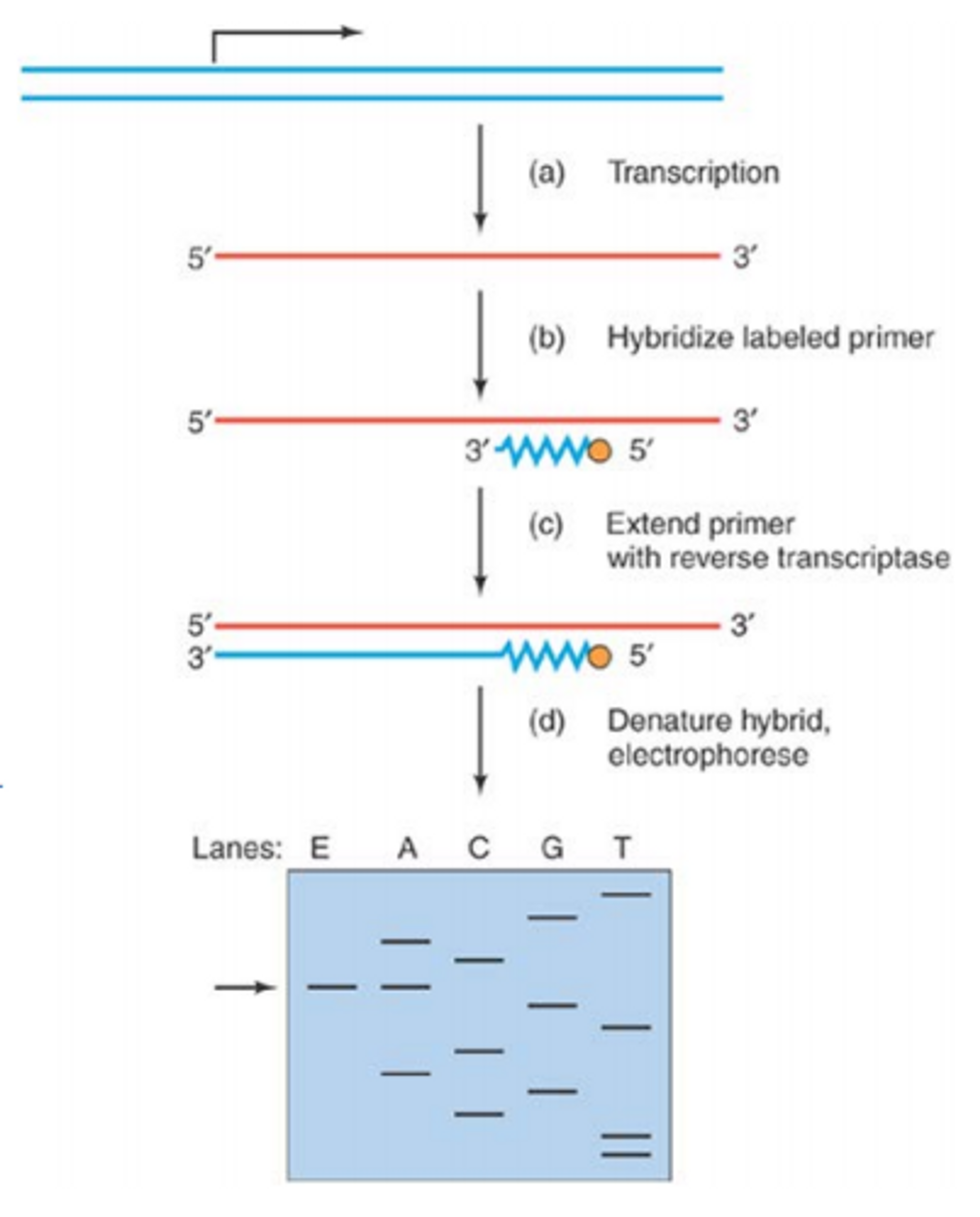
이 경우 처음에는 세포 내에서 transcription을 진행시킨 후, 만들어진 세포 내 RNA를 얻어 실험을 수행함. (이 때 이 RNA들 속에는 우리가 알고자 하는 RNA도 포함되어 있을 것임)
이렇게 뽑아낸 RNA들에, 표지된 oligonucleotide (18bp) primer를 hybridize시켜줌. 이 상태에서 reverse transcriptase를 처리해주게 되면 역전사가 일어나 RNA를 기반으로 DNA 중합체가 만들어지게 됨. (이 때 쭉 역전사가 일어나다가 RNA의 끝부분을 만나면 더이상 참고할 주형가닥이 없으므로 역전사가 종료됨)
이후 RNA-DNA hybrid를 denature시키고 새로 합성된 DNA를 DNA gel에 로딩함. 그 결과가 위 그림 아래에 나타나 있음.
보면 E lane에 표시된 것이 바로 파란색 사슬의 전기영동 결과임. 한편 이 때 새로 합성된 DNA의 말단 부분 서열, 즉 3' 말단 서열이 곧 transcript의 시작 서열이므로 이를 알기 위해서 맨 처음에 주어진 파란 부분 DNA에 대해 sanger sequencing을 진행함. (그 결과가 위 그림 맨 아래 오른쪽에 나타나 있음)
이후 이 둘을 비교하게 되면 새로 합성된 DNA의 3' 서열이 무엇인지, 즉, transcript의 시작 서열이 무엇인지 알 수 있게 됨. (이 경우 DNA의 3' 서열은 A이고, 이에 상보적인 transcript의 starting nucleotide는 U임)
run-off transcription
run-off transcription 또한 전사 시작부위를 알아내기 위해 사용 가능한 방법 중 하나임.

이 방법은 간단히 말해 in vitro, 즉 시험관에서 transcription을 시키는 것임. 우선 위 그림에서는 SmaI을 이용해서 DNA를 잘라줌. (물론 이 녀석에 의해 잘린 부위 내부에 전사가 시작되는 부위가 있어야 함)
이후 RNA polymerase와 표지된 NTP를 넣어주고 인위적으로 transcription을 시켜줌. 그러면 전사가 진행되다가 더이상 합성할 서열이 없어 RNA polymerase가 떨어져버리게 됨. 이렇게 중간에 중합효소가 떨어진다는 점 때문에 이 방식을 run-off transcription이라 함. (이 때 RNA polymerase가 떨어지는 근본적인 이유는 SmaI에 의해 잘린 절단부위때문임)
그렇다면 왜 이런 일을 하는가. 우선 이렇게 만들어진 transcript 조각들은 전기영동을 통해 관찰 가능한데, 이 때 band의 두께를 통해 이 부위에 대한 전사가 얼마나 활발히 이루어졌는지를 알 수 있음. 그 밖에 RNA의 길이를 통해 SmaI 절단부위로부터 얼마정도 upstream에 transcription이 개시되는 부위가 존재하는지에 대해서도 알 수 있음.
다음 포스트에서는 세포 내에서 전사의 정도를 측정하는 방법들에 대해 알아보도록 하자.