이번 포스트에서는 DNA sequencing(DNA 시퀀싱)에 대해 알아보고, 뒤이어 restriction mapping, site-directed mutagenesis에 대해 알아보도록 하자.
DNA의 sequencing에 있어 가장 기본에 해당하는 방법은 바로 sanger sequencing임. sanger sequencing에서는 dideoxy nucleotide를 사용한다는 것이 가장 큰 특징 중 하나임.
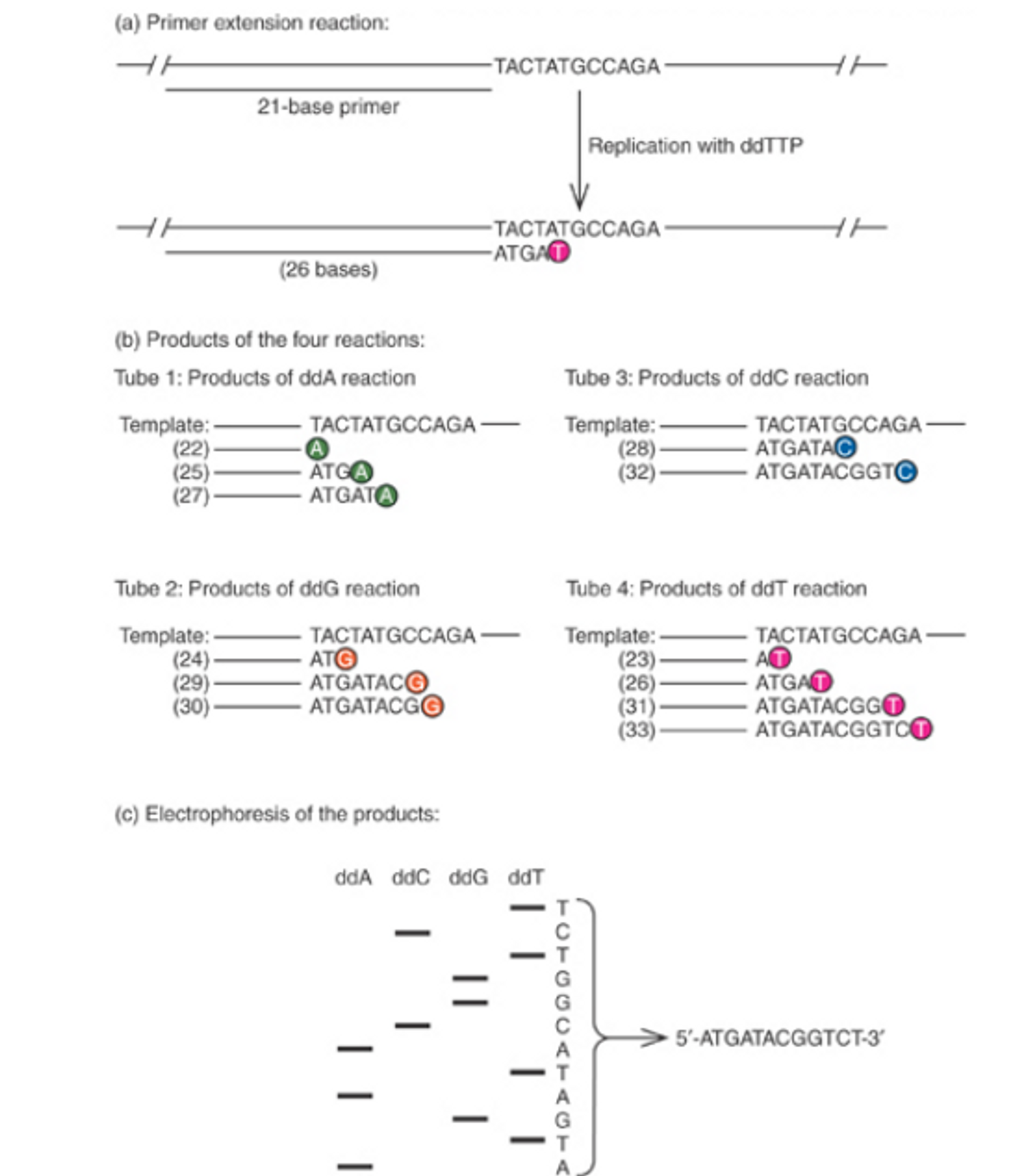
위 그림에서도 나타나 있듯 우리가 알고자 하는 서열 바로 앞쪽에 primer를 결합시켜 준 후 dNTP와 함께 4종류의 ddNTP를 하나씩 넣어주면서 결합을 시켜봄. 그럴 시 랜덤하게 다양한 위치에 ddNTP가 결합되게 되는데, 이 때문에 생성되는 DNA 서열의 길이가 다 다를 것임.
이후 electrophoresis를 통해 가장 짧은 서열부터 긴 서열 순으로 ddNTP를 읽는 방식을 통해 sequencing이 이루어짐. (참고로 당연히 ddNTP를 넣어줄 시 3' C에도 OH가 없으므로 더이상 DNA 합성이 진행되지 않고 중단됨)
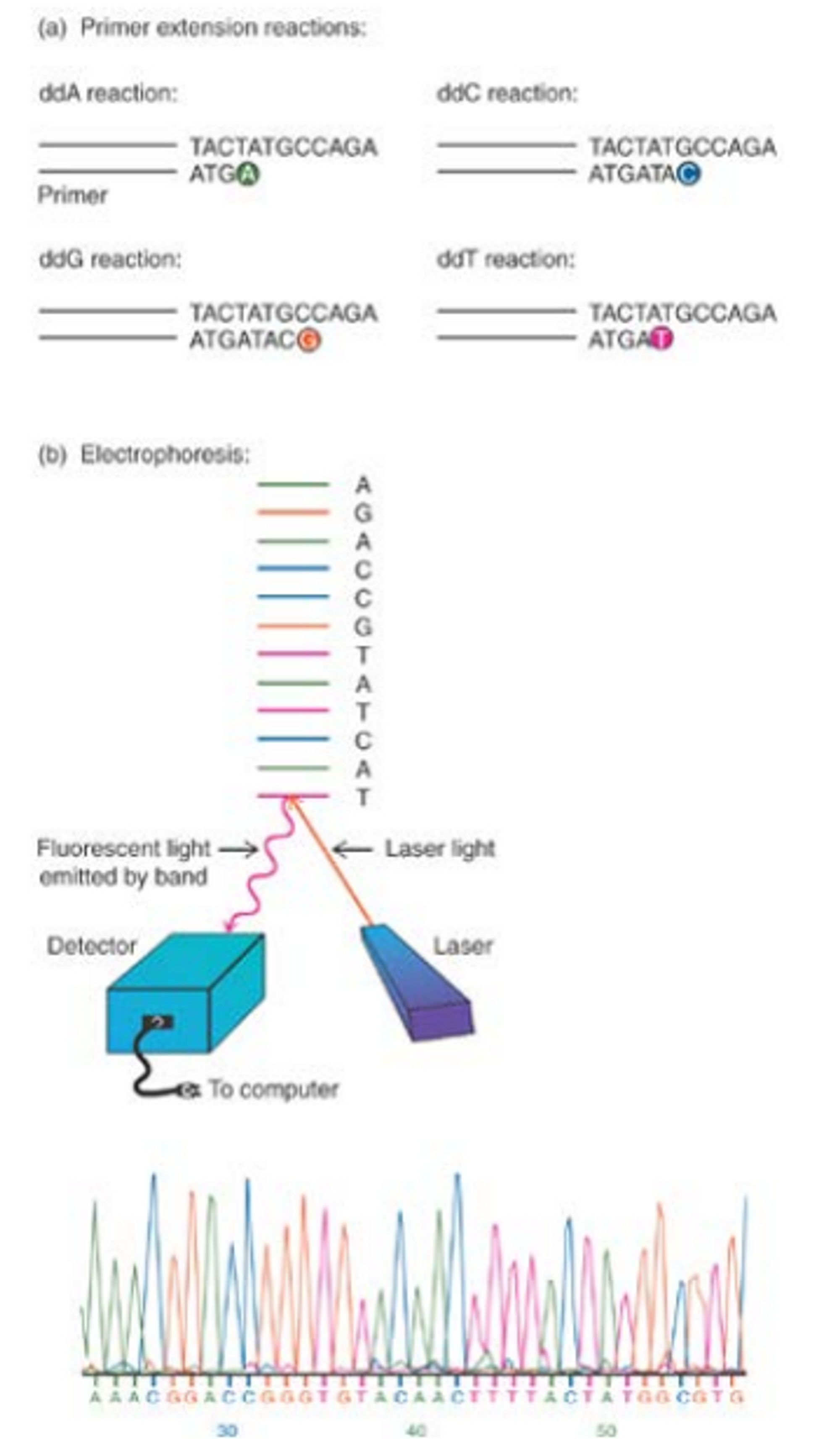
한편 앞서의 방법은 ddNTP 4종류를 한 종류씩 넣어가면서 수동적으로 분석해야 함. 그러나 기술의 발전에 따라 위 그림과 같 각 ddNTP를 서로 다른 형광으로 염색한 후 이 녀석들을 한 번에 다 넣어준 후 sanger method를 통해 반응을 진행시킬 수 있게 됨. 이후 형광을 detection할 수 있는 detector(감지기)를 통해 형광을 관찰함. 이러한 방법을 통해 automated DNA sequencing이 가능해짐.
이후 기술 발전에 따라 훨씬 빠르고 갚싸게 DNA sequencing을 할 수 있게 되었는데, sanger sequencing 이후 개발된 고효율의 sequencing 방법들을 통틀어서 next generation sequencing(NGS)이라 부름.
대표적인 예가 Illumina sequencing, pyrosequencing 등임. (참고로 이 때 pyrosequencing에서는 DNA 합성 시 나오는 PPi가 방출될 때마나 나오는 형광을 측정하는 방식을 이용해 sequencing을 진행함)
다음으로 restriction mapping에 대해 알아보자.
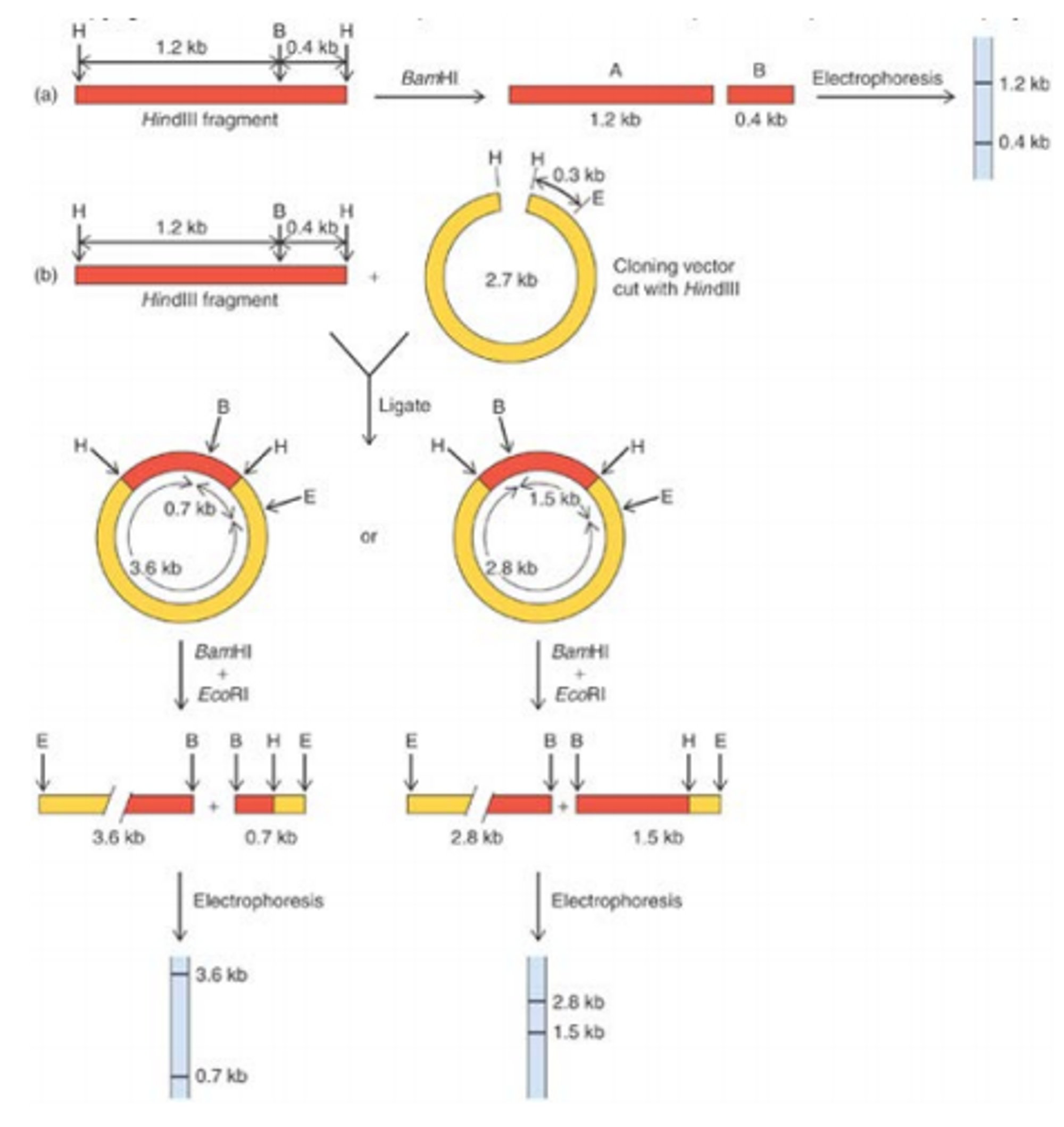
위 그림에 restriction mapping의 과정이 나타나 있음. 이 실험법의 목적은 우리가 sequencing하기 전에 cloning을 할 때 cloning이 제대로 된 녀석만을 선별해내는 것임. 이에 대해 조금 더 자세히 알아보자.
위 그림에서 나타나 있는 것처럼 우리가 sequencing하고자 하는 서열에 있어 H, B와 같은 제한효소에 의해 잘리는 부위가 비대칭적임. (따라서 (a)에서 나타난 것과 같이 BamH1으로 잘라줄 경우 서로 다른 크기의 조각으로 잘림) 한편 이 서열을 cloning해주는 과정에서, 양 말단이 모두 HindIII에 의해 잘리므로 정방향으로도, 역방향으로도 plasmid에 들어갈 수 있음. 이 때 plasmid 내부에도 다른 제한효소 절단자리가 있음. 따라서 위 그림과 같이 정방향으로 들어갔는지, 역방향으로 들어갔는지에 따라 BamH1, EcoR1으로 잘라줬을 시 형성되는 서열의 길이가 다름. (이 길이는 물론 electrophoresis로 확인함) 따라서 이 결과를 바탕으로 정방향으로 cloning된 녀석들만 선택해 sequencing할 수 있음.
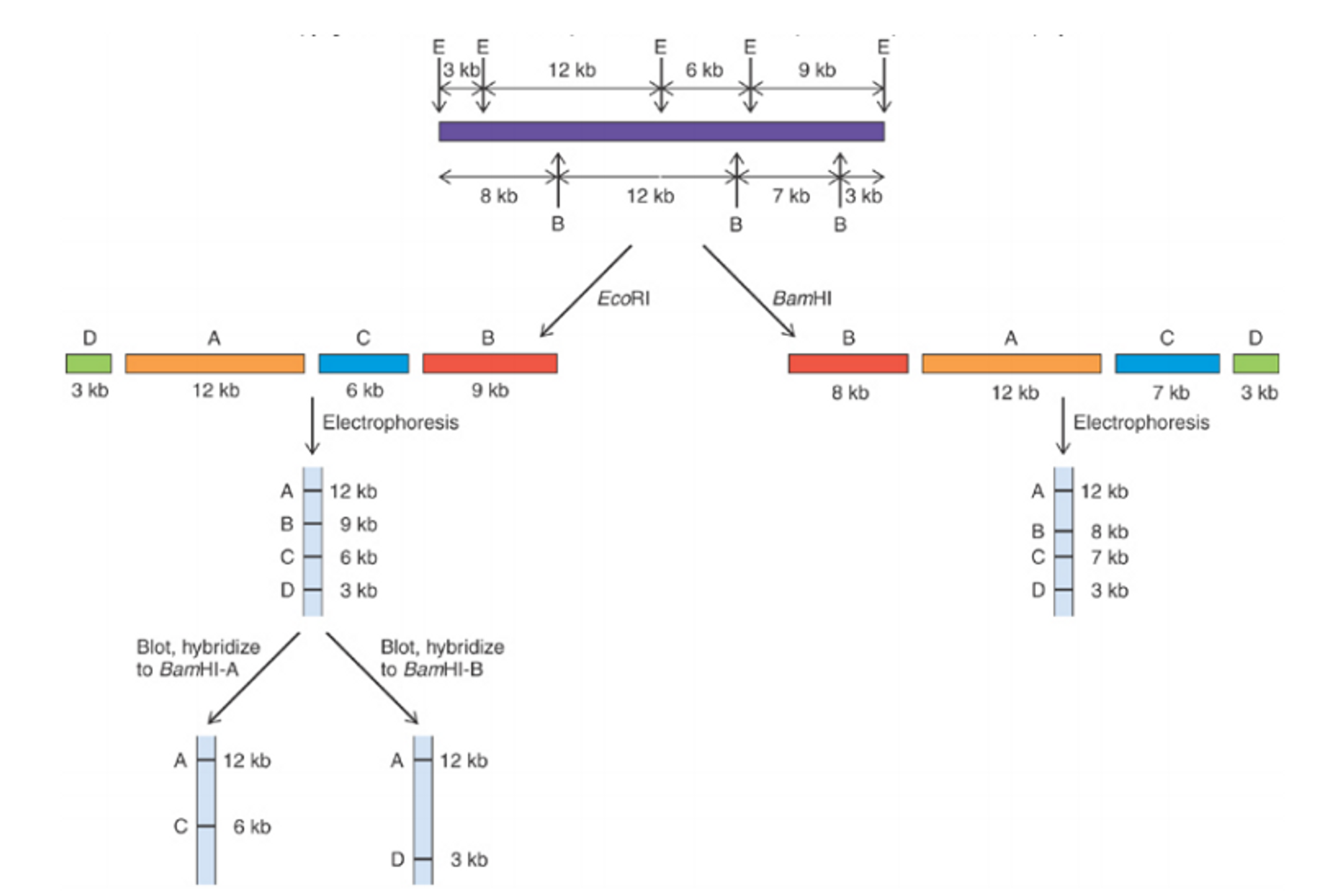
더 나아가 southern blot과 restriction mapping을 동시에 수행할 수도 있음. 위 그림에 나타나 있는 것처럼 특정 서열의 EcoR1, BamH1 절단 자리가 다양함. 이 때 EcoR1, BamH1을 각각 사용해서 잘라준 결과가 위 그림의 가운데에 나타나 있음.
한편 일단 EcoR1으로 잘라준 후 이 녀석들과 BamH1-A or B or C or D와 hybridize(혼성화)를 시켜주고 나서 이를 다시 southern blot하고 관찰할 수도 있음. (예를 들어 EcoR1으로 잘라준 후 BamH1-A와 hybridize시켜줄 시 맨 위 그림에서도 알 수 있듯 BamH1-A에 의해 잘리는 부분과 겹치는 부분이 EcoR1 절편에서의 A, C에 해당하므로 이들에 해당하는 band가 관찰됨) 그럼 왜 이런 과정을 거치는 것일까. 이는 제한효소 절단부위의 위치를 정확히 모르는 상황에서 유용할 수 있음. 즉, 이 방법을 이용하면 특정 DNA 서열 안에서 제한효소 절단부위의 상대적 순서를 유추할 수 있음.
다음으로 site-directed mutagenesis에 대해 알아보자. cloning을 한 gene을 대상으로 특정 부위의 염기서열을 우리가 원하는 염기서열로 바꿔주고자 할 때 본 방법이 사용될 수 있음.

위 그림에서처럼 원래는 A-T 서열이었는데 이 부분을 T-A로 바꿔주고자 한다면, 일부러 그 부위에 붙는 primer를 짤 때 우리가 바꿔주고자 하는 염기와 상보적인 염기를 그 자리에 위치시킴. (위 그림에서처럼 A-A, T-T로 위치시킴) 그럴 시 그 부분에서는 수소결합이 일어나지 않겠지만 나머지 부분에서 수소결합이 일어나므로 primer는 어느 정도 붙을 수 있음. 이후 이를 PCR시키고, 이 때 다시 생성되는 원래 cloned DNA는 Dpn1이라는 효소(이 녀석은 methylation이 일어난 DNA만 분해시켜줌)로 분해시켜주게 되면 결과적으로 우리가 원하는 mutagenesis가 일어난 cloned DNA만 얻을 수 있음.
다음 포스트에서는 trasncript(전사체, 즉 RNA)를 분석하기 위해 사용 가능한 실험방법들에 대해 알아보도록 하자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 5.6 : run-on transcription, reporter assay (리포터 어세이) (0) | 2023.06.27 |
|---|---|
| [분자생물학] 5.5 : nothern blot(노던 블롯), S1 mapping, primer extension, run-off transcr (0) | 2023.06.27 |
| [분자생물학] 5.3 : hybridization (혼성화) - 서던 블롯, DNA 지문분석, in situ 혼성, 웨스턴 블롯 (0) | 2023.06.25 |
| [분자생물학] 5.2 : radioactive, nonradioactive tracer (0) | 2023.06.24 |
| [분자생물학] 5.1 : DNA 전기영동, SDS-PAGE, chromatography(크로마토크래피) (0) | 2023.06.24 |