이번 포스트에서는 DNA와 단백질간의 상호작용을 살펴보고자 할 때 사용가능한 실험방법들에 대해 알아보자.
nitrocellulose filter binding assay

이 방식에서는 위 그림과 같이 DNA는 통과 가능하지만 protein은 통과 불가능한 filter를 사용함.
이 때 우리가 내용물의 양을 어느 정도 알고 있는 labeled DNA와 protein의 혼합물을 이 filter를 통해 부어줬을 때 만약 DNA와 protein이 서로 결합하여 상호작용한다면 protein과 붙은 DNA는 filter를 통과하지 못하고 남아있을 것임. 따라서 filter를 통과한 DNA의 양과 filter를 통과하지 못한 DNA의 양을 측정하면 DNA와 특정 protein간의 affinity(결합력) 정도를 확인 가능함.
gel mobility shift
gel mobility shift 방식은 EMSA(electrophoresis mobility shift assay)로도 불림.

EMSA의 실험원리가 위 그림에 나타나 있음. 우선 이 경우에도 DNA는 labeling함. 한편 여러 protein들과 함께 DNA를 섞어준 후 electrophoresis로 분리해 주었을 때 만약 넣어준 protein들과 DNA가 상호작용하지 않는다면 위 그림 맨 아래와 같은 band만 나올 것임.
그러나 만약 DNA 일부가 protein과 결합한다면 bare DNA band(DNA만 존재하는 band)와 함께 이동된 band도 관찰될 것임. (protein이 붙어 더 무거워졌으므로 당연한 현상) 그리고 만약 DNA가 여러 단백질들과 결합한다면 결합한 만큼 더 무거워져서 더 많이 이동될 것임. 이 이동의 정도를 보고 DNA, protein 간 결합여부, 결합세기를 파악할 수 있음.
footprinting
footprinting에는 매우 많은 종류가 있음. 예를 들자면 DNase footprinting, Dimethylsulfate(DMS) footprinting, Hydroxyl radical footprinting 등이 있음. 이 중에서 DNase footprinting, Dimethylsulfate(DMS) footprinting에 대해 조금 더 자세히 알아보자.
1) DNase footprinting

이 방식은 기본적으로 DNA의 일부에 protein이 붙은 후 DNase를 처리해주면 DNA와 protein이 붙은 부분 이외의 나머지 부분은 잘려나갈 것이라는 생각을 바탕으로 고안된 실험기법임. (이 때 모조리 다 절단되는 것을 막기 위해 DNase는 mild하게 처리해줌)
보면 위 그림과 같이 protein이 DNA의 특정 위치에 붙게 되면 그 위치에 해당하는 부분만 DNA 절단이 일어나지 않아서 protein 제거 이후 DNA 조각들을 electrophoresis로 나열했을 때 길이 추이에 불연속적인 부분이 생기게 됨. 그 결과가 위 그림 오른쪽에 나타나 있음.
보면 1, 2, 3, 4로 갈수록 protein의 양을 증가시켜준 것인데, protein 양이 늘어날수록 가운데에 band가 나타나지 않는 부분이 점점 더 선명해지는 것을 알 수 있음. 심지어 이 부분을 sequencing하기만 하면 protein이 DNA의 어떤 부위, 어떤 서열에 결합하는지까지 알 수 있음.
2) DMS footprinting

이 때는 DMS를 사용하게 되는데, DMS를 처리해 줄 시 특정 잔기에 methylation(메틸화)이 일어나게 됨. 이후 메틸 잔기가 붙은 site만 잘라주게 되면 앞서와 동일한 원리로 protein과 DNA가 결합한 부분을 제외한 나머지 부분에 해당하는 길이에서 electrophoresis band가 나타나게 됨. 그 결과가 위 그림 오른쪽에 나타나 있음.
참고로 이 때 1, 4의 경우 protein을 아예 안 넣어준 것이고, 2, 3의 경우 protein을 넣어준 것임. 이 때 1, 4와 2, 3에 대해 위 그림에서 꺽쇠로 표현된 부위에서 차이가 발생함을 알 수 있음. 이 때 한 가지 눈여겨볼 점이 있는데, 나머지 부분의 경우 2, 3 부위에서 band가 더 연하게 나오거나 아예 안나왔다면, *로 표시된 부분의 경우 오히려 1, 4에서보다 2, 3에서 band가 더 두껍게 나온다는 것을 확인 가능함. 이를 통해 protein에 의해 특정 부위에서의 DMS 활성은 오히려 더 증가될 수도 있음을 알 수 있음.
ChIP (chromatin immunoprecipitation)
이 방법은 protein이 DNA 안의 특정 gene과 실제로 결합하는지를 다시 한 번 확인할 때 주로 사용하게 됨.
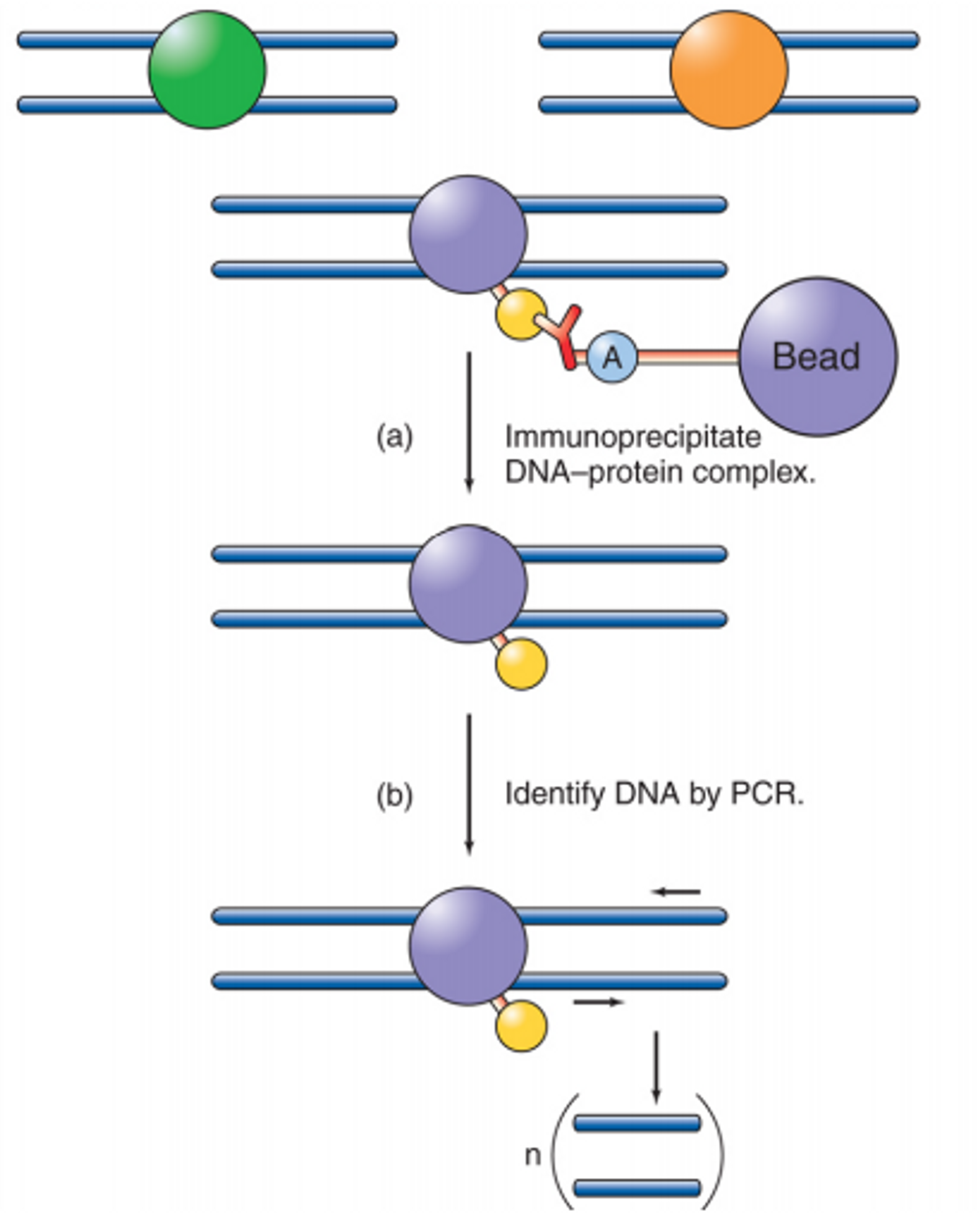
위 그림에 ChIP의 대략적인 과정이 묘사되어 있음. 보면 DNA와 붙은 특정 protein이 구체적으로 어디 붙었는지를 알고자 할 때 그 특정 protein(위 그림에서는 보라색)에 붙는 항체를 사용함. 이 때 이 항체의 Fc(항체의 줄기) 부분에는 아주 무거운 bead가 붙어있음. 이 때문에 결국 특정 protein-DNA 복합체는 매우 무거워져서 immunoprecipitate되게 됨. 이후 분리된 복합체 내의 DNA를 PCR을 통해 증폭시켜서 어떤 서열에 protein이 붙었는지를 확인할 수 있음.
다음 포스트에서는 단백질 단백질 상호작용에 대해 알아보기 위해 사용 가능한 방법들에 대해 알아보자.