이번 포스트부터는 polyadenylation이 어떻게 일어나는지와 관련된 구체적인 모델에 대해 알아보자.
polyadenylation은 구체적으로 어떻게 일어나는 것일까.

위 그림에 그 과정이 간략히 나타나 있음. 보면 AAUAAA와 GU/U element 사이가 poly A site로 인식되어 cleavage되게 됨. 이 cleavage 과정에서는 RNA polymerase II의 CTD가 매우 중요하게 작용함. 이와 함께 CPSF라는 단백질이 AAUAAA site를 인지하고, 이것 또한 cleavage에 있어 매우 중요하게 작용함.
한편 일단 cleavage가 일어나고 나면 그 이후부터는 CTD가 작용하지 않음. 대신 PAP라고 하는 녀석이 들어오고, 이 녀석이 초기에 대략 8~10개의 A를 3' 말단에 붙여주게 됨. 이후 10개 정도의 A가 합성되고 나면 PAB II라는 녀석이 추가로 들어와 이미 붙어있는 A에 이어서 poly A를 길게 연장시키게 됨.
이제 이와 관련된 실험결과를 살펴보자.

우선 이 실험에서는 CTD가 cleavage에 필수적인지를 알아보기 위해 L3 pre-mRNA를 사용했고 L3 band와 cleavage가 되어서 나타나는 5', 3' band의 여부를 통해 cleavage가 일어났는지 여부를 판단함.
위 그림 오른쪽 위 결과의 경우 IIA와 IIO 각각의 양을 점점 늘려줬을 때 cleavage에 의해 5', 3' band가 진해짐을 확인 가능함. 이를 통해 IIA form으로 존재하던, IIO form으로 존재하던 이들 RNA polymerase II들은 cleavage를 촉진시켜준다는 것을 알 수 있음.
조금 더 구체적으로 pol II 내부의 CTD가 cleavage에 필수적인지를 확인하기 위해 수행한 실험결과가 위 그림 오른쪽 아래에 나타나 있음. 보면 lane 4, 5, 6(인산화가 되어있지 않은 CTD 양을 점차적으로 늘려줌)과 lane 8, 9, 10(인산화가 되어있는 CTD양을 점차적으로 늘려줌)을 비교해 볼 시 인산화가 되어 있는 CTD가 그렇지 않은 CTD에 비해 cleavage를 더 잘 도와준다는 것을 알 수 있음. (5', 3' band가 더 진해짐)
참고로 그 옆쪽에 나타나 있는 (b) 결과를 보면 IIB를 아무리 증가시켜봐도 cleavage에 의한 band가 나타나지 않는다는 것을 알 수 있음. (이는 IIB가 CTD를 가지고 있지 않기 때문임)
이후 여러 추가실험을 통해 밝혀진 precleavage complex(절단이 일어나기 전에 형성되는 단백질 complex)의 모식도는 아래와 같음.

보면 CPSF(cleavage and polyadenylation specificity factor)가 AAUAAA를 인지하여 결합하고 있고, PAP(poly A polymerase)도 결합하고 있으며, 그 밖에 각종 element들도 결합해 결국 precleavage complex를 만들게 됨.
이제 cleavage가 일어나고 난 이후, polyadenylation이 initiation되는 과정과 관련된 실험 결과에 대해 알아보자.

이 때 넣고 빼준 각각의 factor들에 대해 살펴보자. DE-100, DE-600은 각각 DEAE를 통해 분리되었으며, sodium chloride 100, 600mmol을 넣었을 때 각각 elution된 정체모를 단백질들을 의미함. (그 당시에 정체는 몰랐지만 뭔가 polyadenylation과 관련되어 있을 것이라고는 알고 있었음. 지금은 DE-100이 poly A polymerase, DE-600이 specificity factor[CPSF]라는 것을 알고 있음)
그리고 AAUAAA 서열은 정상적인 polyadenylation signal, AAUCAA 서열은 제대로 기능하지 못하는 polyadenylation signal임.
실험 결과를 살펴보면 DE-100, DE-600, AAUAAA(정상)가 다 있는 경우에만 제대로 polyadenylation이 일어나 polyadenylated RNA band가 관찰됨.

다음으로 위 실험 결과를 살펴보자.
우선 왼쪽의 경우에는 AAUAAA(정상) signal이 포함된 RNA를 사용함. 우선 lane 1, 2, 3, 4를 살펴보자. 이 경우 WT의 mRNA를 넣었음. 보면 시간 경과에 따라 처음에는 A가 붙지 않은 녀석이 많다가 점차 polyadenylated된 mRNA들이 많아지는 것을 알 수 있음. (이 결과가 일종의 control)
다음으로 lane 5, 6, 7, 8을 살펴보자. 이 경우 애초에 처음 넣어줄 때부터 mRNA 3' 말단에 A를 어느 정도(대략 40개) 붙여준 상태로 동일한 실험을 반복함. 그랬더니 시간이 지나도 추가적인 polyadenylation이 앞서에 비해 잘 일어나지 않는다는 것을 확인할 수 있음.
다음으로 오른쪽 실험 결과를 살펴보자. 이 경우 애초에 AAGAAA signal을 사용했기 때문에 lane 13, 14, 15, 16을 보면 시간이 지나도 polyadenylation이 일어나지 않는다는 것을 알 수 있음. 그런데 lane 16, 18, 19, 20을 보면 처음부터 어느 정도의 A를 붙여준 상태에서 동일한 실험을 진행시켜줄 시 시간 경과에 따라 polyadenylation이 잘 일어난다는 것을 알 수 있음.
이를 통해 초반에 AAUAAA signal dependent하게 짧은 A만큼을 합성하는 step이 존재하고, 이후 어느 정도 길이(40 이하)의 A가 붙게 되면 그때부터는 AAUAAA signal independent하게 추가적으로 A를 합성하는 두 단계의 반응이 따로 존재할 것임을 알 수 있음.
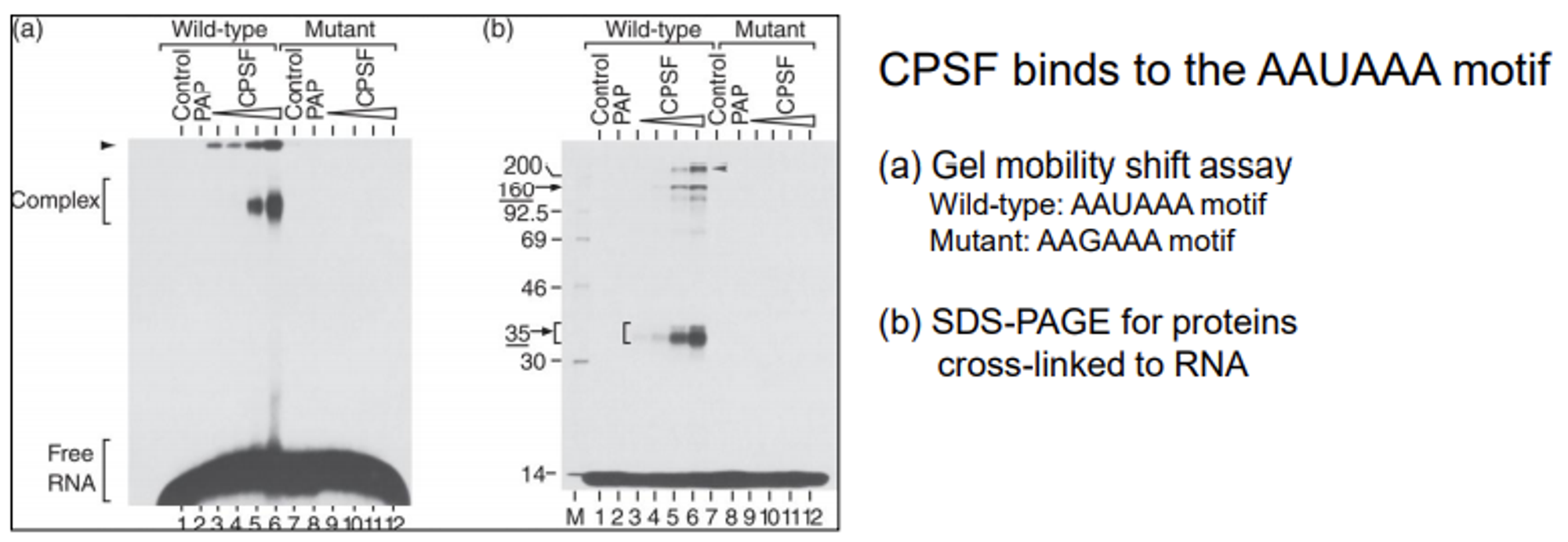
한편 위 실험 결과는 CPSF와 관련된 결과인데, 우선 (a)는 substrate에 해당하는 RNA들과 함께 각종 단백질들을 넣어주고 gel mobility shift assay를 수행한 결과임. 보면 CPSF에 의해 shift가 일어나고 있음. 이를 통해 CPSF가 polyadenylation을 위해 RNA에 가장 먼저 결합하는 단백질 중 하나라는 것을 알 수 있음.
다음으로 (b)의 경우 RNA, protein이 결합된 sample에 UV를 쬐어서 cross-linking을 시켜준 후 SDS-PAGE를 내려본 결과임. 이 때 160, 35kDa 부근에 signal이 나타남을 확인 가능하고, 이들 polypeptide가 모여 CPSF를 이루게 됨.
다음 포스트에서는 poly A가 elongation(신장)되는 과정에 대해 살펴보자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 15.4 : polyadenylation의 메커니즘 - 4 (0) | 2023.12.21 |
|---|---|
| [분자생물학] 15.4 : polyadenylation의 메커니즘 - 3 (0) | 2023.12.21 |
| [분자생물학] 15.4 : polyadenylation의 메커니즘 - 1 (0) | 2023.12.20 |
| [분자생물학] 15.3 : poly A의 기능 (0) | 2023.12.20 |
| [분자생물학] 15.2 : 5'-Cap의 기능 (0) | 2023.12.19 |