이번 포스트에서는 nucleosome-free zone에 대해 알아보도록 하자.
nucleosome positioning이 가능하므로, 실제로 어떤 부위에는 nucleosome이 매우 dense하게 존재해서 DNA를 감고 있는 반면, nucleosome이 거의 없는 nucleosome-free zone도 존재할 수 있음.
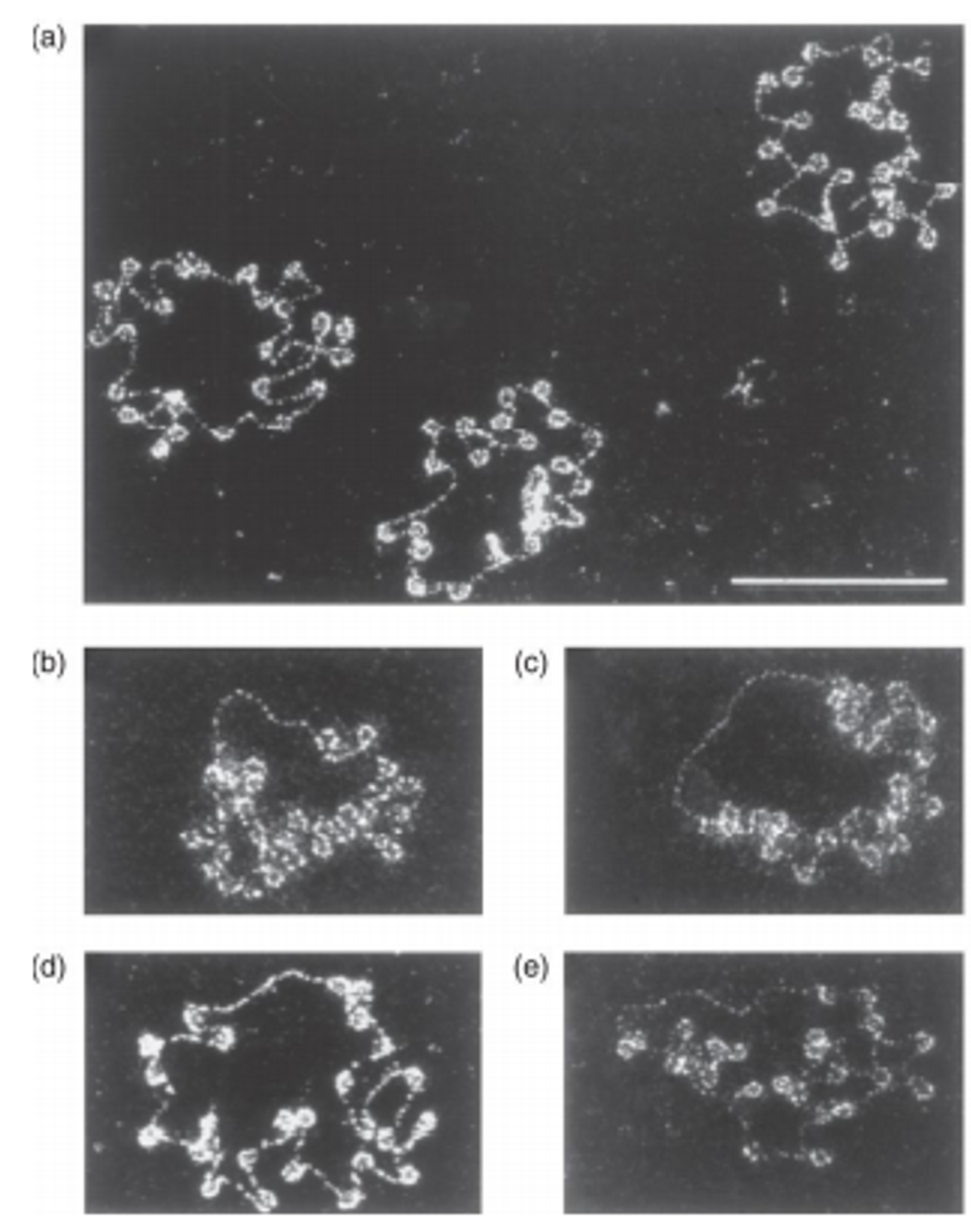
실제로 위 그림을 보면 (a)의 경우 매우 균등하게 nucleosome이 분포하고 있지만 (b), (c)와 같은 것들을 보면 한 쪽에 nucleosome이 치우쳐져 있고, 나머지는 nucleosome-free zone으로 존재한다는 것을 알 수 있음. (즉, (a)와 같은 경우 뿐만 아니라 (b), (c)와 같은 경우도 존재함)

이와 관련된 흥미로운 실험에 대해 알아보자. 우선 위 그림과 같은 circular DNA가 있으며, 이 때 우리가 ORI 근방 부분을 nucleosome-free zone으로 추측하고 있다고 하자. 이 때 이 부분이 정확히 nucleosome-free zone인지 여부를 확인하기 위해 각종 제한효소로 이 circular DNA를 잘라봄. (circular한 DNA이므로 어디가 어딘지를 알기 위해서는 제한 효소로 잘라볼 필요가 있음)
우선 BamH1으로 잘라봄. BamH1으로 자를 시 거의 정 가운데에 우리가 예측하는 nucleosome-free zone이 위치할 것이므로 위 그림 (b) 가운데와 같은 결과가 나올 것으로 예측됨.

위 그림은 실제 결과인데, 보면 정말 예측한 것과 유사한 모양이 나타났음을 확인 가능함.
한편 BgI1으로 자르게 될 경우 앞서 그림 (b)와 같이 맨 끝 부분에 nucleosome이 존재하지 않는 형태가 예상됨.

위 그림은 실제 결과인데, 이 경우에도 정말 예측한 것과 유사한 모양이 나타났음을 확인 가능함.

한편 위 그림에 나와있는 것처럼 EcoR1을 이용해 DNA를 잘라 (b)에 나타난 것과 같은 조각을 만든 후 DNase를 처리해주는 실험을 수행할 수도 있음. 이 때 DNase는 histone이 감고 있는 부분은 잘 못자르는 반면 nucleosome-free zone은 아주 잘 자를 것임.
특히 이 부분은 DNase에 대한 hypersensitivity가 나타나는데(bulk DNA에 비해 10배 더 sensitive할 시 그냥 DNase sensitive라 하고 bulk DNA에 비해 100배 더 sensitive할 경우 DNase hyper-sensitive라 함.
일반적으로 chromatin 중에 active하게 전사되는 gene 부위는 DNase sensitive하며, 이 gene들을 조절하는 control region들은 DNase hyper-sensitive함) 이 hypersensitivity에 의해 정말로 두 조각의 DNA fragment가 나타나는지 여부를 확인해봄.

그랬더니 실제로 위 그림과 같이 cut된 67 조각에 해당하는 band가 나타남. (다만 작은 조각들의 경우에는 band가 뚜렷하게 나타나지 않았는데, 이는 아마 너무 짧은 DNA 조각들의 경우 대부분 degradation되어버려서 나타난 결과일 것으로 생각됨)
방금 전 사용한 아이디어를 조금 더 정교하게 다듬으면 구체적으로 DNase-hypersensitive region이 어디인지를 특정할 수도 있음.

기본적인 idea는 위 그림과 같음. 우선 (a)와 같이 모든 부분이 다 nucleosome으로 이루어져 있는 경우 DNase를 처리해 줘도 잘려나간 부위가 없을 것임. 이후 protein을 제거해주고 특정 제한효소를 처리한 뒤 상보적으로 이 서열에 붙어 표지 가능한 probe를 붙여주게 되면 결과적으로 제한효소 절단부위 사이의 전체 길이에 해당하는 DNA band가 관찰될 것임.
다음으로 (b)와 같이 중간에 한 부분이 nucleosome-free zone(active한 gene의 control region)인 경우 DNA를 처리하게 되면 2개의 조각으로 나누어지게 됨. 이후 앞서와 동일한 과정을 수행하면, 이번에는 probe가 달린 조각이 앞서보다 짧을 것이고, 이 짧은 조각에 해당하는 1개의 band만 관찰될 것임. 위 그림에서의 조각 길이는 6kb임. 결과적으로 제한효소 절단부위로부터 6kb 떨어진 위치에 nucleosome-free zone이 있다는 것을 알 수 있음.
이제 이 방법을 이용해 수행한 실제 실험 결과를 살펴보자.

이 실험은 특별히 human globin gene에 대한 control region의 hyper-sensitivity를 확인하고 그 위치를 정확히 파악하기 위해 수행됨. (참고로 (a), (b)는 human globin gene이 활발히 발현되는 HEL의 핵을, (d)는 human globin gene이 거의 발현되지 않는 J6의 핵을 사용함)
우선 (a)부터 살펴보자. (a)에서는 DNase와 함께 1.4kb Bam Eco probe를 사용함. 그 결과 4번 band와 3a, 3b band를 관찰할 수 있었음. 이를 통해 4, 3a, 3b 위치가 nucleosome-free zone임을 확인함. (참고로 4, 3a, 3b가 다 나타나는 이유는 어떤 경우 4 부분이 끊어지지만 다른 경우 여기가 끊어지지 않은 채로 3a, 혹은 3b가 끊어졌기 때문임)
한편 4, 3a, 3b 이외의 다른 nucleosome-free zone이 또 존재하는지를 확인하기 위해 (b)에서는 3.3kb Eco RI probe를 사용함. 그 결과 1, 2번 band도 확인할 수 있었고, 결과적으로 (e) 그림에 나오는 것과 같이 1, 2, 3a, 3b, 4의 nucleosome-free zone의 위치를 특정해냄.
(a)와 (b)는 앞서서도 말했듯 human globin gene이 활발하게 발현하는 세포의 핵에서 유래한 sample을 이용함. 반면 (d)처럼 human globin gene이 활발하게 발현하지 않는 세포의 핵에서 유래한 sample을 이용할 경우 cut이 관측되지 않음. (즉, 특정 부위에서의 hypersensitivity가 관측되지 않음) 즉, 이 경우 gene transcription control region이 nucleosome으로부터 free하지 않고, 그 결과 gene의 activation이 잘 일어나지 않는다고 추측할 수 있음.
다음 포스트에서는 histone의 acetylation(아세틸화)에 대해 살펴보도록 하자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 13.6 : chromatin remodeling (0) | 2023.12.18 |
|---|---|
| [분자생물학] 13.5 : histone의 acetylation(아세틸화) (0) | 2023.12.18 |
| [분자생물학] 13.3 : chromatin 구조와 유전자 발현의 관계 (0) | 2023.12.18 |
| [분자생물학] 13.2 : chromatin의 응축화 - 2 (0) | 2023.12.18 |
| [분자생물학] 13.2 : chromatin의 응축화 - 1 (0) | 2023.12.18 |