이번 포스트에서는 지난 포스트에 이어서, 보다 더 거대 규모에서 이루어지는 chromatin의 응축화에 대해 알아보도록 하자.
30nm fiber
nucleosome들끼리 모여 형성하는 것으로 보이는 그 다음 단계의 구조가 바로 30nm fiber임. (형성되는 fiber의 지름이 30nm여서 이런 이름이 붙음)
사실 아직도 in vivo 환경에서 30nm fiber가 관찰된 적은 없음. 다만 in vitro에서는 쉽게 관측 가능한데, 30nm fiber를 in vitro에서 관찰한 실험 결과는 아래와 같음.

이 실험은 앞서 nucleosome을 관찰한 실험과 거의 동일하나, (a)~(g)로 갈수록 점차 salt 농도를 높여봤음. 그랬더니 위 그림에서 나타난 것처럼 점차 어떤 구조가 추가적으로 생김. (g)에 나타난 fiber가 바로 30nm fiber임. 이 30nm fiber 구조는 앞서 형성된 nucleosome 구조만 존재할 때에 비해서 DNA를 대략 6~7배 더 응축시켜주는 효과가 있음.
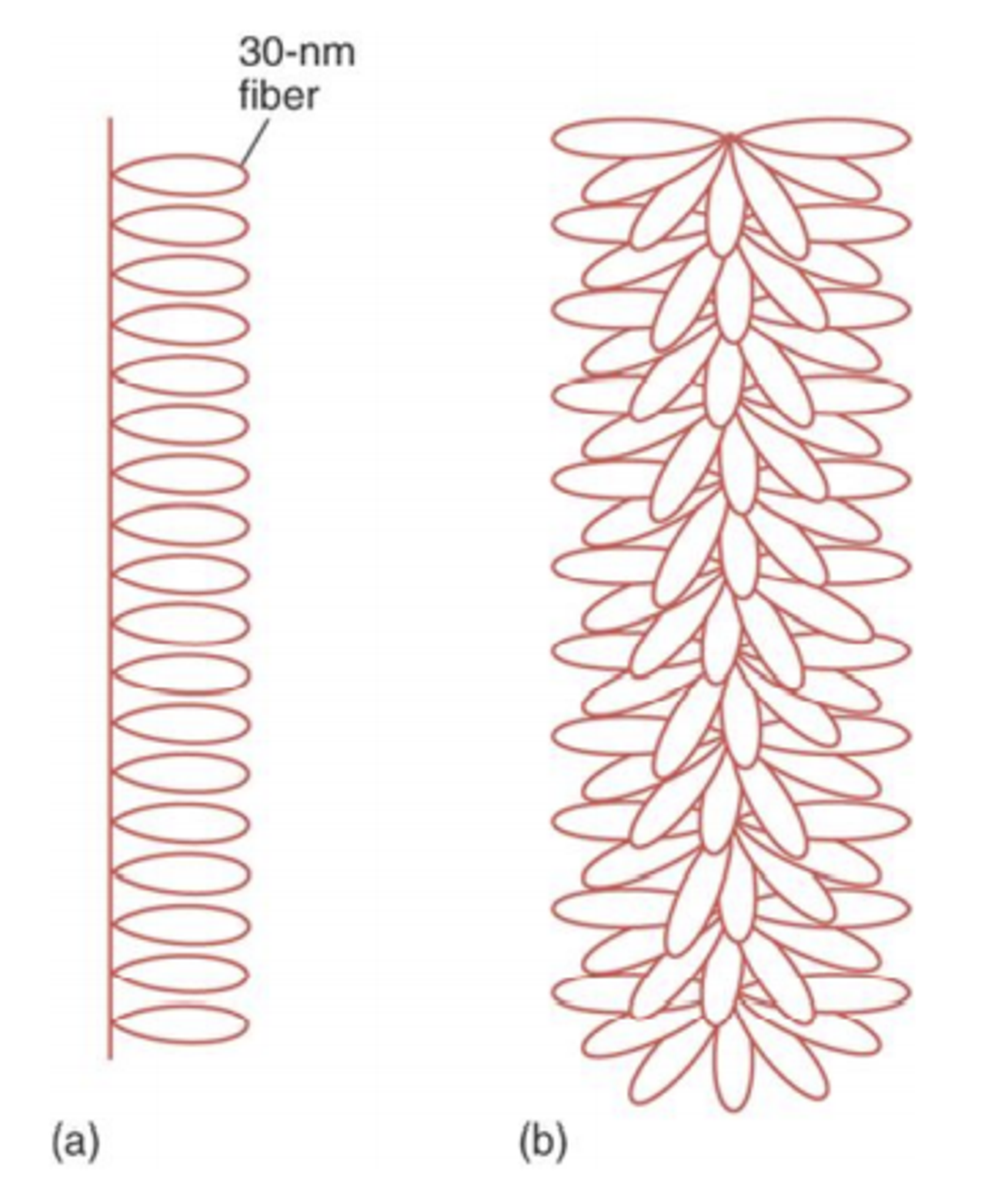
30nm fiber의 구조와 관련된 두 가지 model이 있는데, 그 중 첫 번째 model은 solenoid model임.

참고로 solenoid는 아래와 같은 모양을 의미함.

물론 어떤 관측, 혹은 실험 결과는 이 solenoid model을 지지함. 그런데 solenoid model을 지지하지 않는 결과도 관측됨. 이에 대해 조금 더 알아보자.

위 그림에 나타나 있는 것과 같은 4개의 nucleosome으로 이루어진 구조를 x-ray crystallography로 관찰해봄. (물론 이 때 사용한 x-ray crystallography의 resolution은 9옹스트롬정도로, 보통 한 2~3옹스트롬 정도의 resolution을 가지고 있어야 atom의 정확한 position을 특정할 수 있다고는 함. 그러나 9옹스트롬 정도의 resolution만으로도 어느 정도 정확한 position 확인이 가능함)
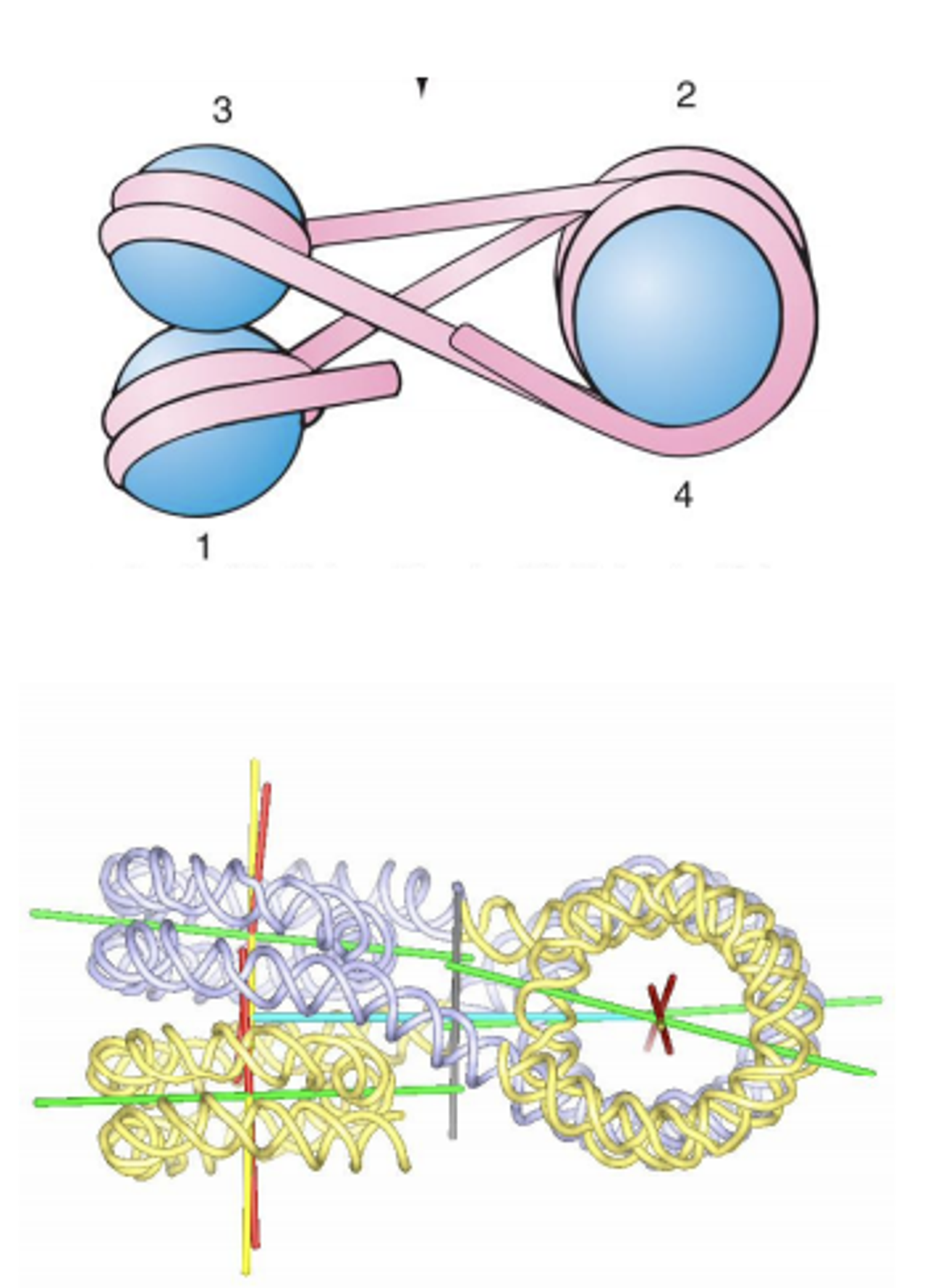
그 결과 위와 같은 구조가 특정됨. 그런데 보면 1, 2, 3, 4 nucleosome의 위치가 지그재그형으로 solenoid model과 맞지 않음.
따라서 이 결과를 바탕으로 아래와 같은 two-start helix model이 새롭게 제안됨.
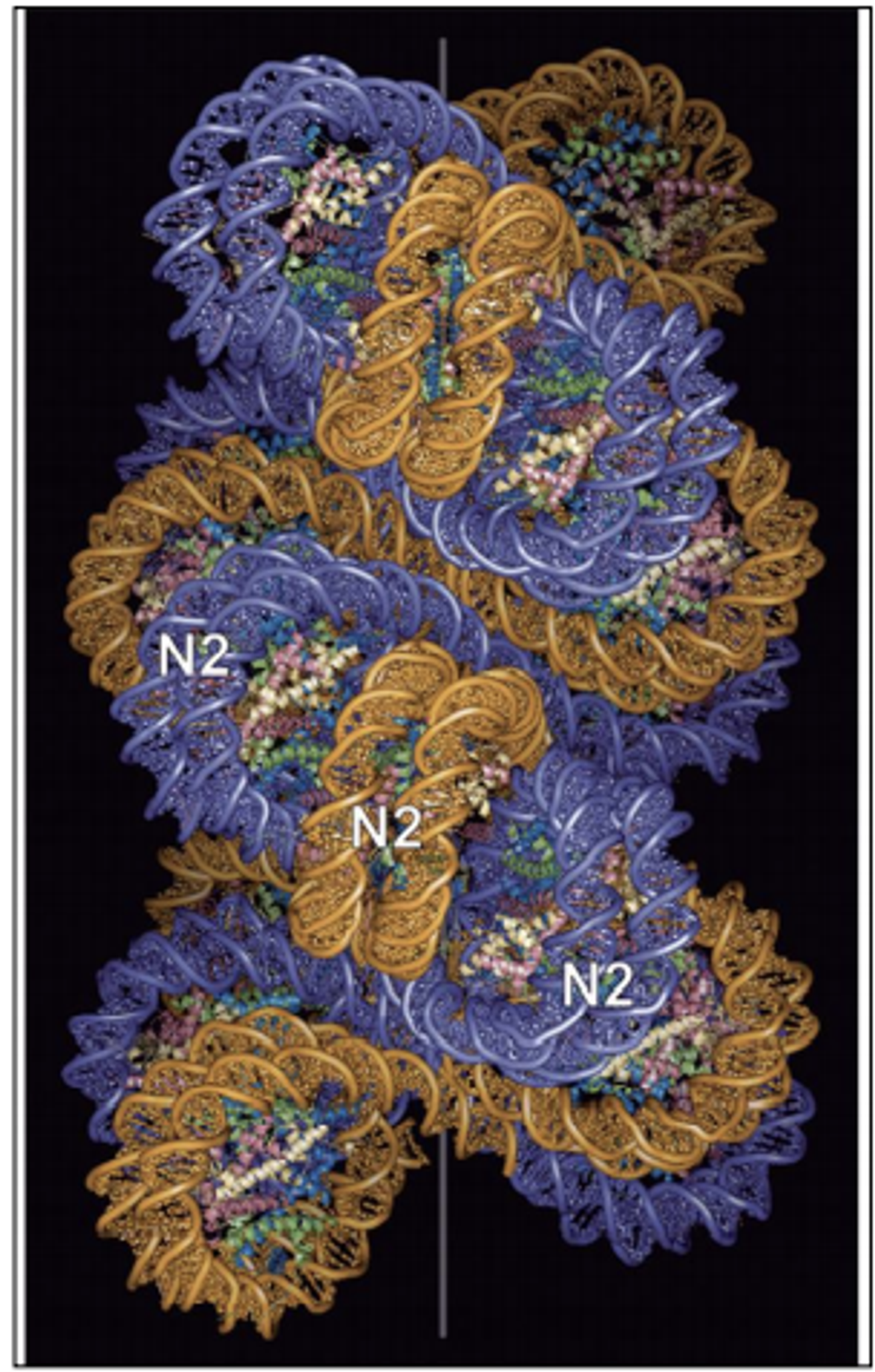
보면 이름에서도 알 수 있듯 두 개의 helix가 쌍으로 start point부터 복잡하게 감겨올라가는 구조임. 위 구조는 딱 보기에도 앞서 봤던 solenoid model보다 훨씬 단단하다는 것을 알 수 있음. (solenoid를 풀 때보다 two-start helix를 풀 때 더 큰 힘이 필요함)
solenoid model과 two-start helix model은 둘 다 실험적인 간접증거들을 바탕으로 만들어진 model들임. 따라서 이 둘 중 어느 model이 진짜 30nm fiber의 구조인지를 확인하기 위해 single-molecule force spectroscopy를 수행함.
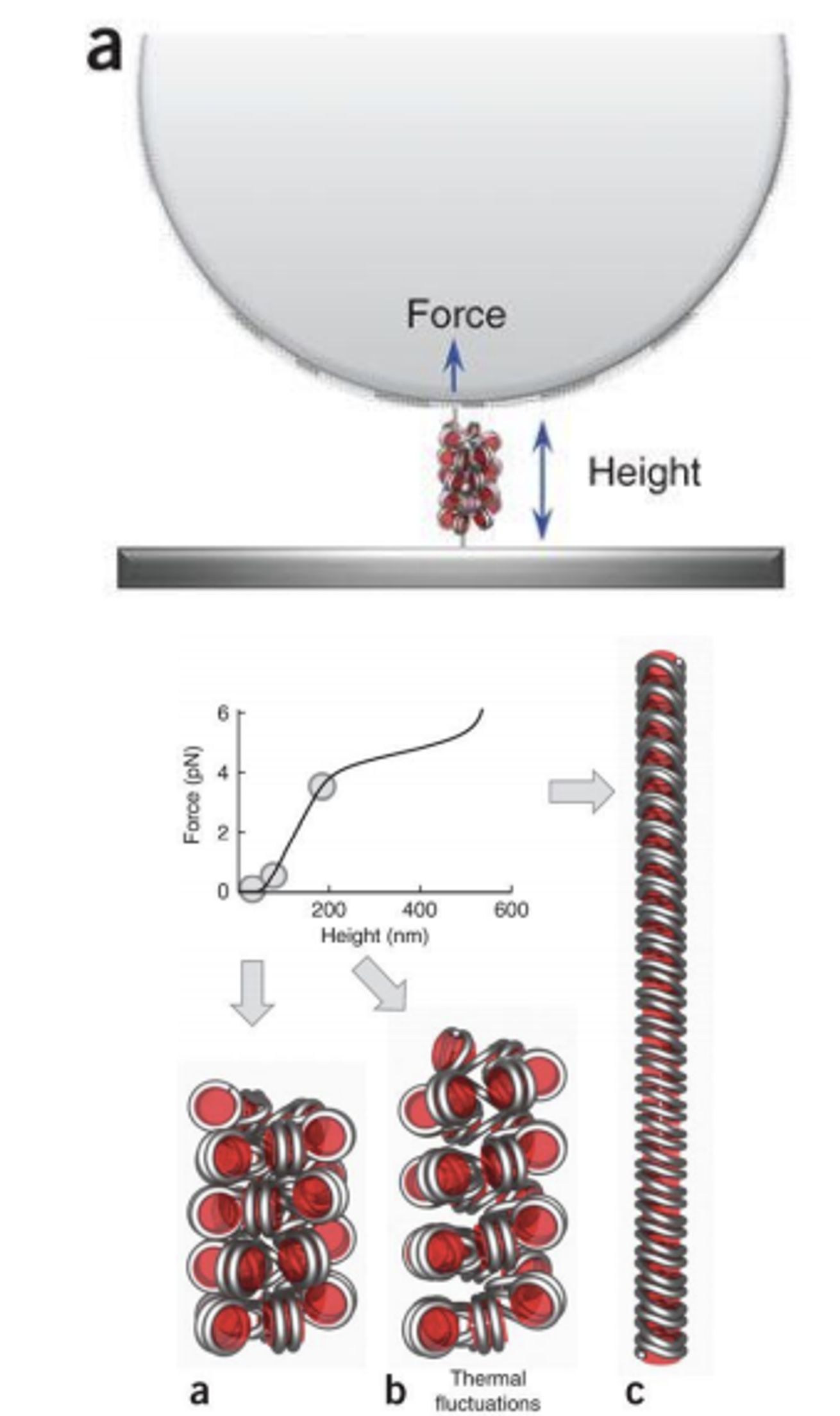
위 그림에 single molecule force spectroscopy의 대략적인 모식도와 실제 실험 결과가 나타나 있음.
이 실험은 기본적으로 1개의 DNA('single molecule'을 대상으로 force를 측정하는 장비를 사용했으므로)를 얻은 후 DNA의 한 쪽은 고정된 곳에, 나머지 한 쪽은 bead에 붙여둔 후 bead를 (magnetic field를 이용해서) 우리가 원하는 대로 조금씩 움직여가며 가해지는 힘, 그 때의 DNA 길이 등을 측정하는 식으로 수행됨.
그 결과 solenoid model과 two-start helix model에 해당하는 (특정 길이까지 늘릴 때 필요한) 힘이 다 측정됨.
한편 결과를 조금 더 면밀히 이해하기 위해 알아야 하는 개념이 있는데, 바로 nucleosome repeat length(NRL)라는 것임. NRL은 한 nucleosome의 시작 부위에 붙는 DNA부터 다음 nucleosome의 시작 부위에 붙는 DNA까지의 길이를 의미함. in vivo상에서의 관측 결과 NRL은 165~212bp정도의 범로 관찰됨.
이 때 NRL이 대략 197bp 근방(상대적으로 큰 NRL)인 경우에는 더 잘 늘어나는 경향이 나타나며 이런 경우가 solenoid model의 형태를 가지는 경우임. 한편 NRL이 대략 167bp 근방인 경우에는 덜 늘어나는 경향이 나타나며 이런 경우가 two-start helix model의 형태를 가지는 경우임.
뒤에서 다시 살펴보겠지만 NRL이 크면 클수록(DNA가 한 nucleosome당 더 많이 감겨있으므로) DNA가 더 compact해서 inactive함. 결론적으로 대부분의 inactive한 chromatin 부위는 solenoid shape으로 이루어져 있고, 일부 active한 30nm fiber는 two-start double helix 모양으로 이루어져 있음.
지금까지 배운 30nm fiber는 interphase nucleus의 대부분에서의 chromatin 구조에 해당할 것으로 생각됨.
higher order chromatin folding
세포가 mitotic phase로 돌입하게 되면 chromatin이 더 접혀 chromosome을 이루게 됨. 이 과정에서 higher order chromatin folding이 추가적으로 일어남. 이 때 folding에 의해 만들어지는 형태를 radial loop라고 함. (즉, chromosome에서 radial loop가 관찰될 것으로 생각됨)
radial loop은 위 그림과 같이 중간에 특정한 scaffold가 있고 scaffold 주변에 30nm fiber가 감겨있는 구조임. (물론 radial loop도 아직 확실치 않은 하나의 model에 불과함)
radial loop와 같은 형태를 생각하게 된 이유는 아래와 같은 실험의 결과 때문임.
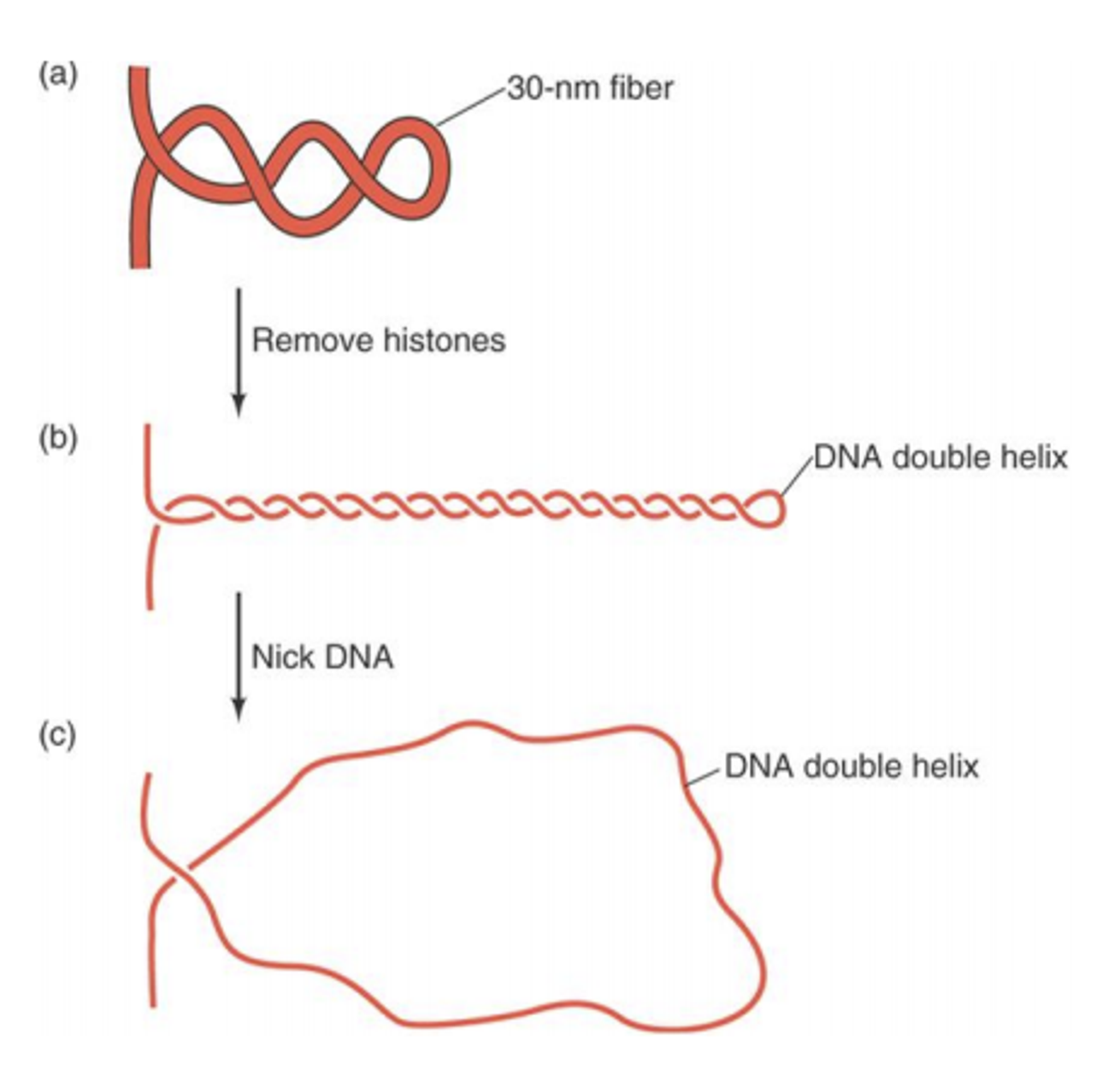
하나하나 살펴보자. 우선 30nm fiber가 형성되어 있는 chromatin에서 histone을 제거해줌. 그랬더니 (b)와 같은 supercoil이 형성됨. (진핵생물에서 supercoil이 형성되었다는 말은 DNA의 양 끝이 (b)그림 왼쪽과 같이 어딘가에 고정되어 있다는 뜻임) 이후 DNA에 nick을 만들어주게 되면 supercoil이 형성되어 있던 부분이 풀리면서 (c)와 같은 거대한 고리를 만들게 됨.
결국 이렇게 형성된 큰 고리에 의해 침강계수 S가 달라지게 되고, 원심분리를 통해 실제로 이런 loop가 형성되었는지, 크기는 어느정도인지 등을 측정할 수 있게 됨. 이 때 이런 loop가 측정되었다는 것은, 결국 진핵생물에서도 circular like한 DNA 구조가 존재해 supercoil이 일어날 수 있다는 것을 의미하고, 결과적으로 scaffold에 의해 DNA의 양 끝이 잡혀 이런 일이 가능하지 않을까 예상해볼 수 있음.
즉 이 실험 결과를 통해 무언가 30nm fiber를 잡아주는 중심부 구조(scaffold)가 있음을 확인할 수 있었음.
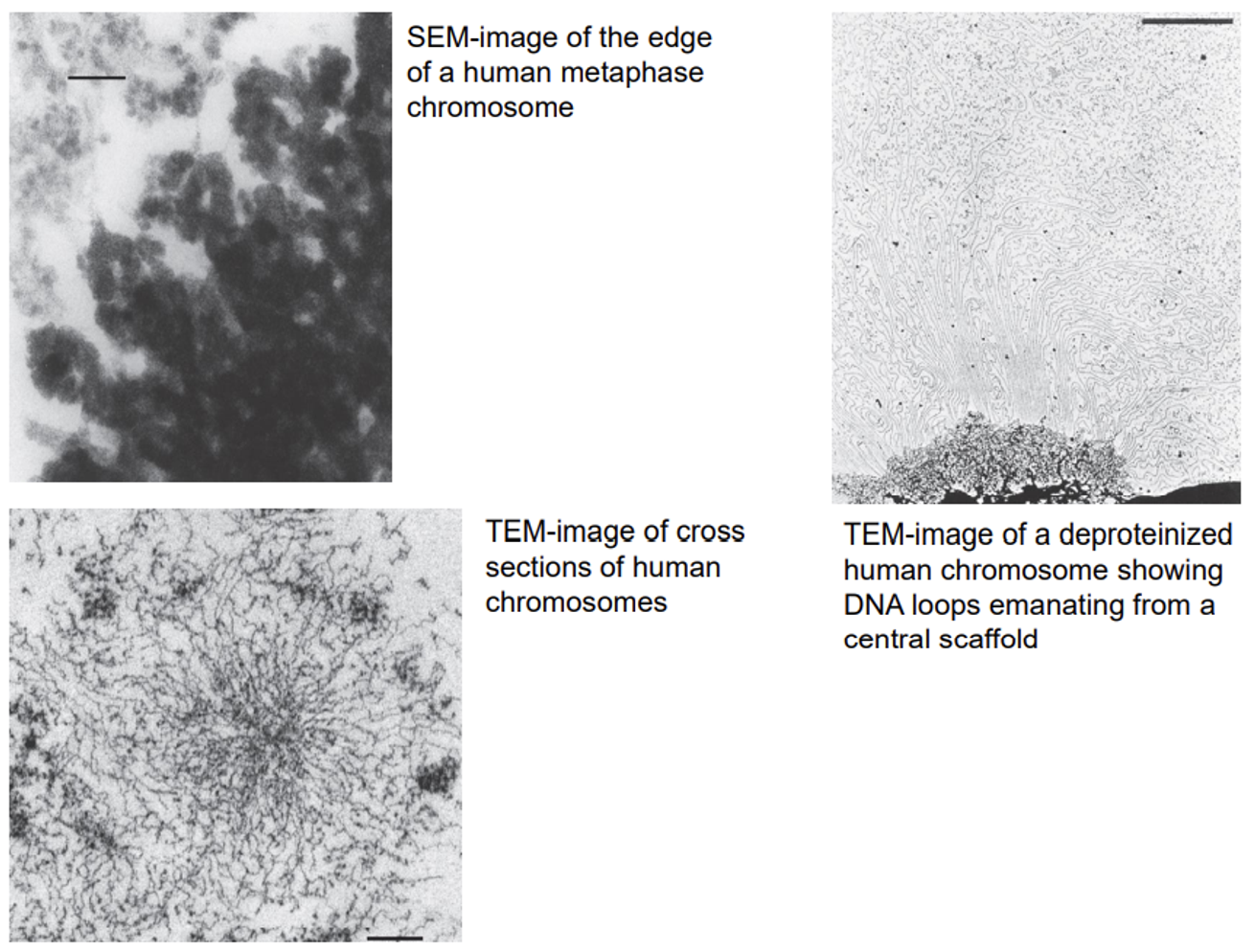
위 그림은 전자현미경을 이용해서 실제로 chromosome이 형성될 때의 구조를 관찰한 것임. 보면 가운데의 scaffold를 중심으로 해서 가쪽으로 loop 형태의 30nm fiber가 튀어나와있음을 확인 가능함. (참고로 위 그림 왼쪽 위가 human metaphase에서의 SEM image, 왼쪽 아래가 TEM image, 오른쪽 위가 histone을 제거한 후 찍은 TEM image임)
다음 포스트에서는 chromatin의 구조가 유전자의 활성과 어떤 연관이 있는지에 대해 알아보도록 하자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 13.4 : nucleosome-free zone (0) | 2023.12.18 |
|---|---|
| [분자생물학] 13.3 : chromatin 구조와 유전자 발현의 관계 (0) | 2023.12.18 |
| [분자생물학] 13.2 : chromatin의 응축화 - 1 (0) | 2023.12.18 |
| [분자생물학] 13.1 : chromatin의 기본 구조 및 histone (0) | 2023.12.18 |
| [분자생물학] 12.7 : 진핵생물에서의 전사인자 조절 (0) | 2023.12.18 |