이번 포스트부터는 chromatin이 어떤 식으로 응축되는지에 대해 단계별로 자세히 알아보도록 하자.
chromatin을 이루는 데 있어 가장 기본이 되는 unit이 바로 nucleosome임.
한 세포 내에 포함되어 있는 DNA를 직선상에 쭉 펴면 2m 길이가 된다고 함. 그런데 이 긴 DNA를 10um 남짓의 작은 핵 안에 packing시키기 위해서는 아주 정교한 compaction이 필요함. (특히 이 compaction 과정에서 DNA가 꼬이면 안되기 때문에 packing order는 매우 정교하게 구성되어 있음)
이러한 packing을 위해 행해지는 folding의 첫 번째 step이 바로 nucleosome을 형성하는 것임. nucleosome은 histone의 core를 가지고 있고, 이 core를 중심으로 DNA가 꼬여있는 모양새임.
nucleosome이라는 구조는 처음에 x-ray diffraction이라는 방법에 의해 관측됨. 이 방법을 이용하면 x-ray를 쪼였을 때 sample이 가지는 특정한 repeat이 있는 경우 이 repeat에 해당하는 x-ray diffraction pattern을 얻을 수 있게 되고, 결과적으로 repeat의 size를 알 수 있게 됨.
이 때 sample로 intact한 nuclei를 사용했고, 그 결과 nuclei 안에 계속 반복되는 특정한 repeat(혹은 unit)이 있음을 확인했으며, 그 크기가 대략 100옹스트롬 정도였음을 측정함. 실제로 추후에 nucleosome 크기를 측정해봤더니 대략 100옹스트롬 정도로 측정됨.
한편 이후에 chemical을 이용해 nucleosome을 이루고 있는 histone 단백질들을 서로 cross-linking시켜봄. (서로 가까이 있는 녀석들이 cross-linking될 것이고, 결국 서로 가까이 있었다는 것은 서로 interaction을 하고 있었다는 것을 의미함) 그랬더니 H3-H4, H2A-H2B가 서로 붙어있음을 확인함.
한편 solution 상태에의 sample에 H3과 H4를 넣어봤더니 tetramer인 (H3-H4)2 형태가 형성됨을 확인함. 그리고 이런 기본적인 물성 실험의 연장선상에서 연구를 수행한 결과 앞서 말한 것처럼 DNA와 대략 동일한 mass의 histone 단백질이 존재하고 있다는 사실도 알아냄.
이러한 실험 결과 1개의 histone octamer에 대략 200bp정도의 DNA가 어떤 식으로든 같이 존재하고 있다는 사실을 알아냄. (실제로는 감겨서 존재하고 있음)
H1 and chromatin
그렇다면 시각적으로 nucleosome을 처음 관찰한 실험은 어떻게 수행되었을까. 이 때는 chromatin에 protease의 일종인 trypsin과 함께 high salt buffer를 처리해줌. 이런 처리를 해주게 되면 상대적으로 DNA와의 상호작용이 적고 trypsin에 의해 잘 잘려나가는 H1을 제거할 수 있음. 이후 H1이 제거된 chromatin 구조를 전자현미경으로 관찰해봄.

그랬더니 위 그림과 같은 결과를 얻음. 이 때 마치 beads-on-a-string과 같은 구조를 관찰할 수 있었고, 이 때의 bead를 nucleosome이라 이름붙임.
nucleosome structure
이후 과학자들은 구체적인 nucleosome의 구조가 궁금해서 x-ray crystallography를 이용해 구조생물학적 연구를 수행해봄.

위 그림에 나온 구조는 DNA, H1을 제외한 octamer 그 자체만을 대상으로 수행한 x-ray crystallography 결과임. 보면 (H3-H4)_2가 중심에 core처럼 위치해있고, 가쪽에 H2A-H2B dimer가 달라붙어 있는 형태임.
사실 이 구조만으로도 DNA가 어떤 식으로 감길 것인지에 대해 어느 정도 예측할 수 있는데, octamer의 표면 charge가 어떻게 분포되어있는지, groove가 어떻게 형성되어 있는지 등을 감안하면 아래와 같이 DNA가 감길 것이라 예측할 수 있음.

따라서 이 때부터 대략 octamer 당 DNA가 2바퀴정도 감길 것이라는 예측을 하고 있었음.
한편 구조생물학자들은 실제로 histone에 DNA가 달려있는 형태 그 자체를 보기 위해 노력함. 이 때 앞서서도 언급했듯 x-ray crystallography 시 가장 큰 난관이 crystal을 만드는 단계이며, 이 때 crystal을 이루는 녀석들이 다 동일해서 일정하게 packing되어야 함.
따라서 각각의 element들의 heterogeneity를 최소화하기 위해 nuclease를 extensive하게 처리해 대부분의 DNA들은 다 잘라주고 최소한 DNA만 남겨서 crystal을 만들고 x-ray crystallography를 수행함. 그 결과 아래와 같은 구조를 얻음.
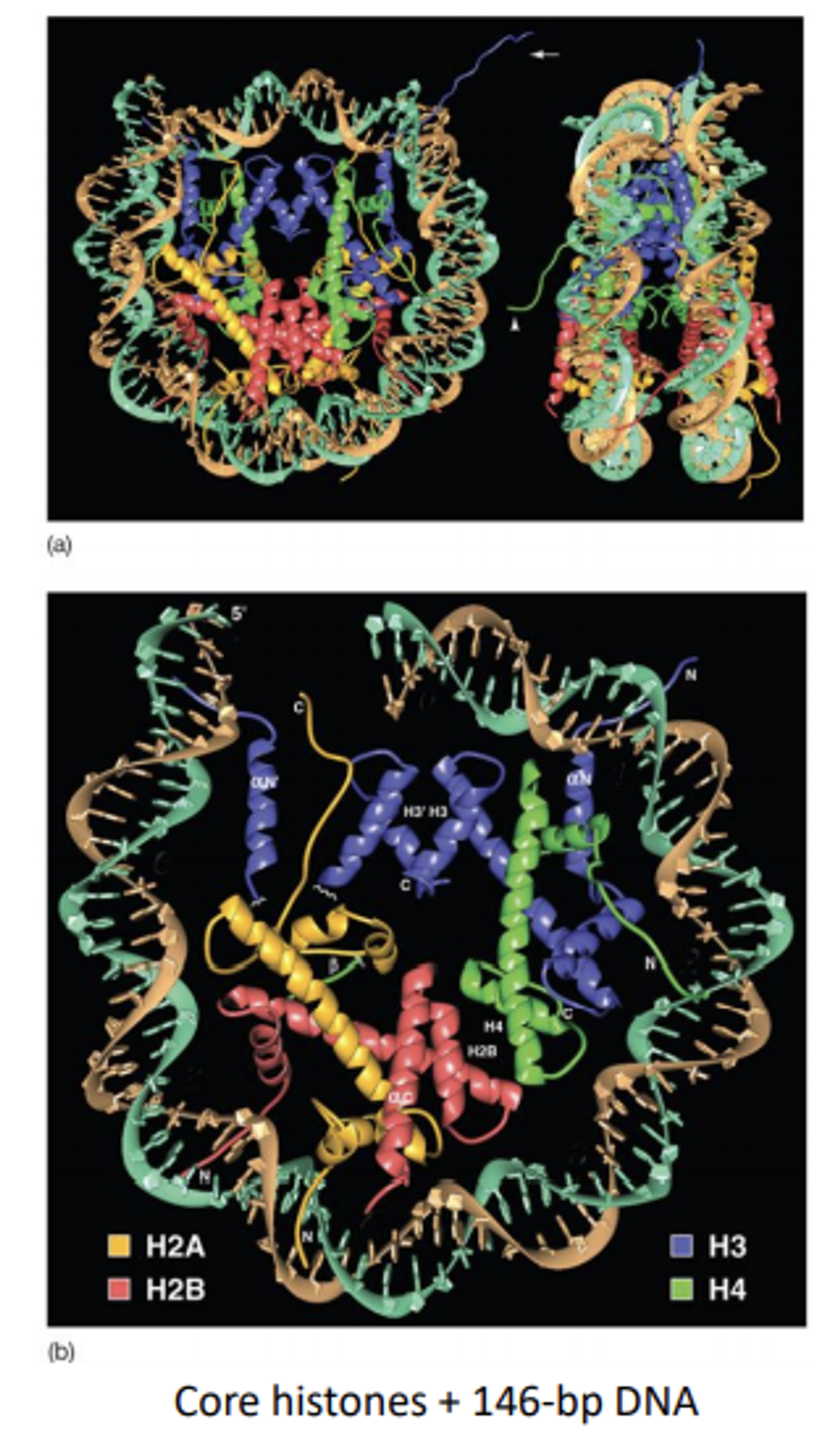
이 때는 거의 대부분의 DNA들을 다 자른 채로 구조 분석을 수행하였으므로 당연히 실제와는 다소 다른 모습이 측정되겠지만, 일단 위 data 하에서는 histone 단백질에 대략 DNA가 1.65번 감긴다는 것을 알아냄.
한편 단백질 fold를 더 자세히 관찰하기 위해 별도의 데이터 가공을 통해 아래와 같은 입체구조를 얻음.
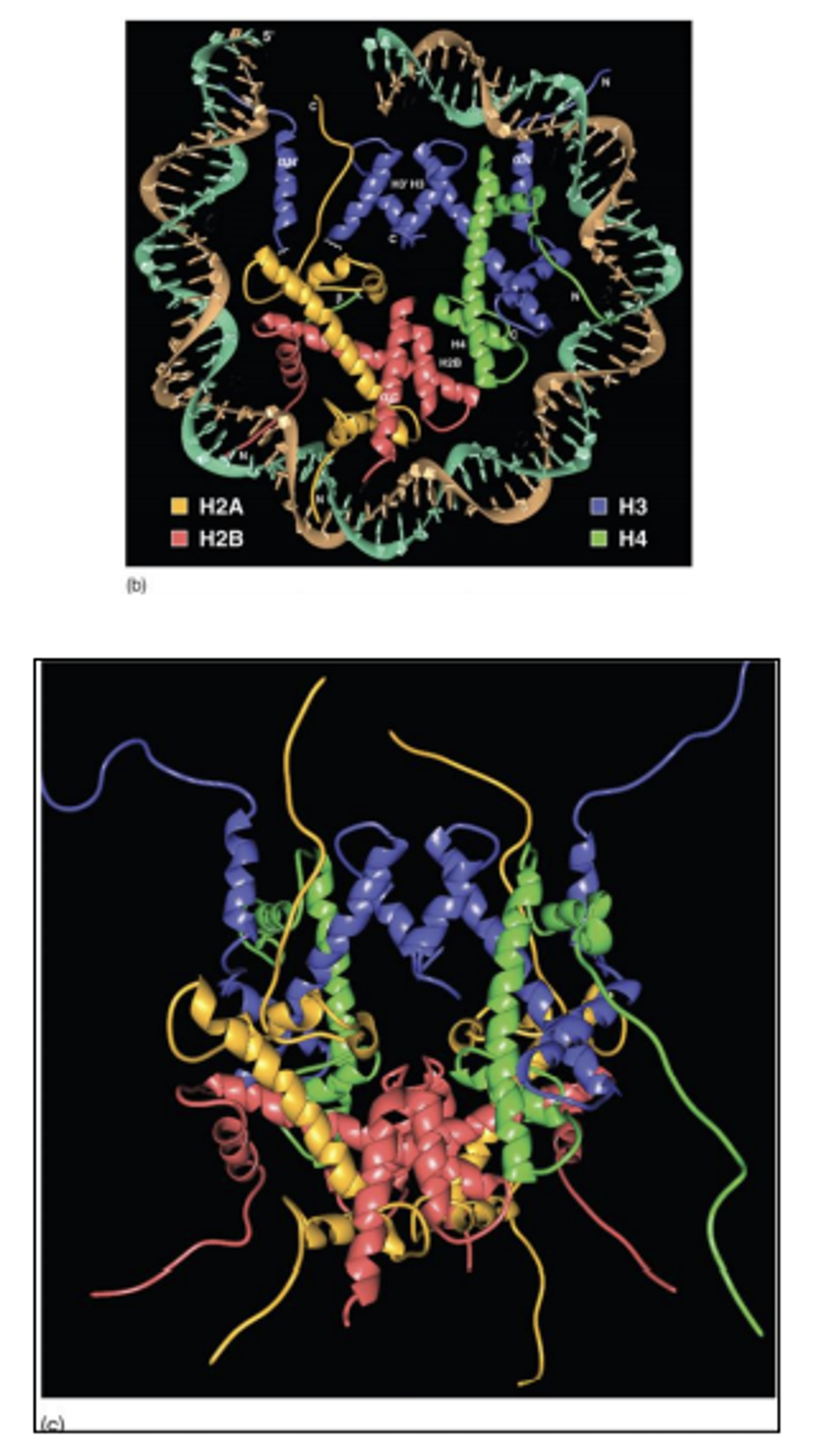
그 결과 core histone들이 공통적으로 histone fold를 가지고 있다는 것을 알아냄. (histone fold는 3개의 alpha-helix들이 loop에 의해 연결된 구조이며, 위 그림 (b)에서 가장 왼쪽의 보라색 alpha-helix부터 차례대로 3개의 alpha-helix들이 이루는 N자형 구조가 바로 histone fold임)
이 밖에 또 공통적으로 core histone에 extended tail이 관측됨. 이 extended tail은 전체 core histone mass의 28%를 차지할 정도로 property가 크다는 사실도 알아냄. (tail이 위 그림 (c)에 방사상으로 표현되어 있음) 이 tail은 위 그림에서도 보이듯이 unstructured되어 있음. 그렇기에 안쪽에 존재하는 rigid한 structure(DNA와 다양하게 interaction하기 상대적으로 어려움)들에 비해 tail domain들은 더 flexible해서 DNA와의 interaction을 많이 매개하고 있음.
이 뿐만 아니라 이 unstructured domain에 enzyme들이 쉽게 접근해서 tail들을 modification시킬 수 있음. (modification 결과 전사에 대한 전반적인 regulation이 일어남) 이 tail 부분은 사실 최근에 여러 질병들과의 관련성 등이 밝혀지면서 매우 주목받고 있는 연구대상 중 하나임. (이후 등장할 histone code라는 개념과 이 tail이 밀접하게 관련되어 있음)
한편 과학자들은 core histone이 DNA를 얼마나 잘 접고 있는지를 알아내기 위해 DNA chromatin에 protein을 다 제거해봄. (즉, deproteinize시켜봄)
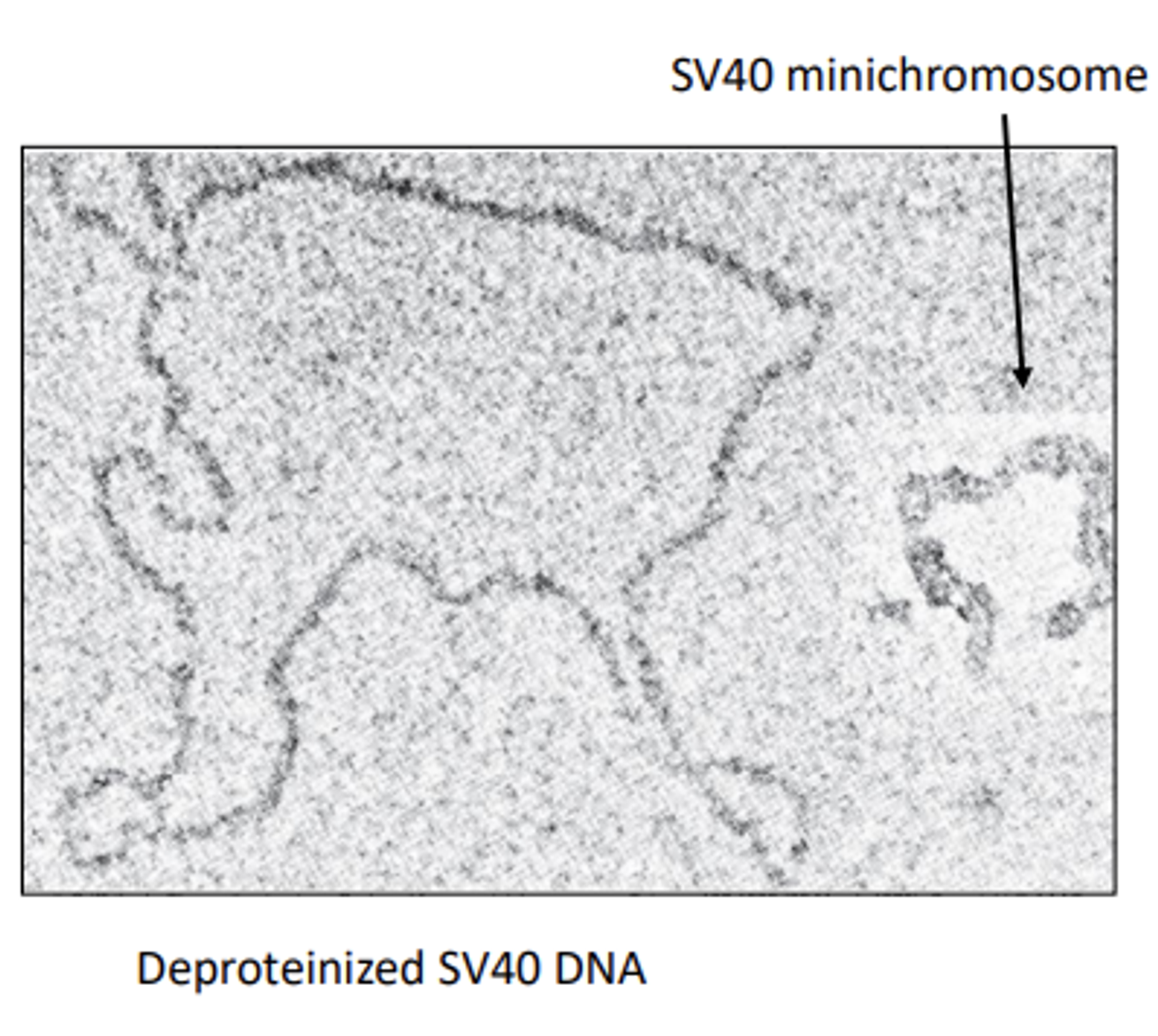
그 결과가 위 그림에 나타나 있음. 보면 core histone이 없어질 시 DNA가 엄청난 길이로 풀리는 것을 확인 가능함. 결과적으로 이 실험을 통해 core histone에 의한 winding 만으로도 DNA를 6~7배 compaction시키는 효과가 있다는 것을 알아냄.
물론 이 정도 compaction만으로는 2m 길이의 DNA를 10um 크기의 핵 내에 넣기에 불충분함.
따라서 다음 포스트에서는 DNA들을 어떻게 추가적으로 compaction시킬 수 있을지에 대한 내용들을 살펴볼 것임.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 13.3 : chromatin 구조와 유전자 발현의 관계 (0) | 2023.12.18 |
|---|---|
| [분자생물학] 13.2 : chromatin의 응축화 - 2 (0) | 2023.12.18 |
| [분자생물학] 13.1 : chromatin의 기본 구조 및 histone (0) | 2023.12.18 |
| [분자생물학] 12.7 : 진핵생물에서의 전사인자 조절 (0) | 2023.12.18 |
| [분자생물학] 12.6 : insulator (0) | 2023.12.18 |