이번 포스트에서는 TFIIB의 구조와 기능에 대해 알아보자.
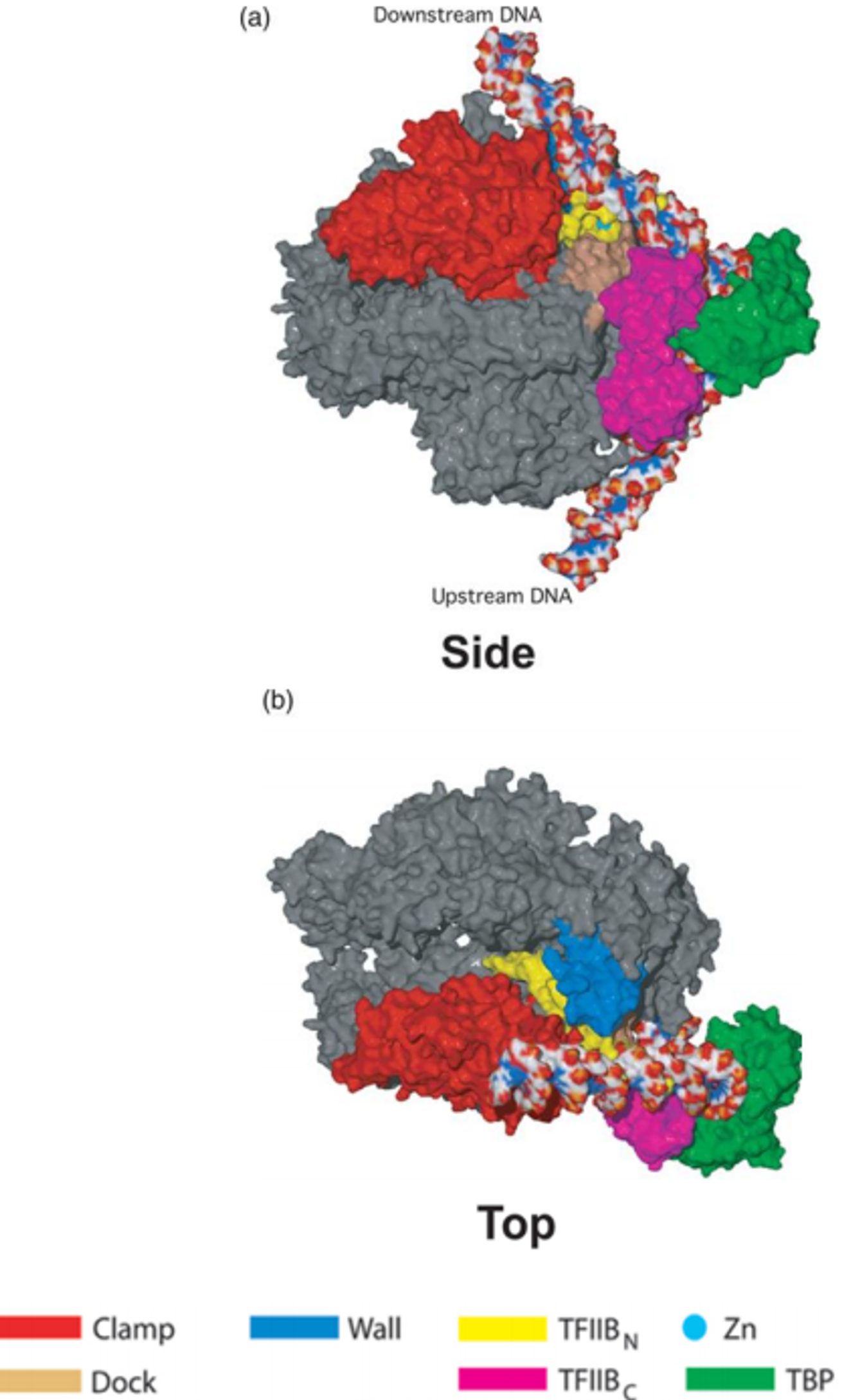
위 그림에서 회색, 붉은색으로 표시된 부분이 RNA polymerase II이고 초록색으로 표시된 부분이 TBP임. 이 때 TBP와 RNA polymerase II 사이에 끼어들어가 있는 듯한 모양새를 하고 있는 보라색+노란색의 구조가 TFIIB임. 조금 더 구체적으로 분홍색은 TFIIB 중 C-terminal이 포함된 부분(TFIIB_C)이고 노란색은 TFIIB중 N-terminal이 포함된 부분(TFIIB_N)임.
이 중 TFIIB_C 부분은 위 그림에도 나와있는 것처럼 TBP와 interaction하고 TFIIB_N 부분은 RNA polymerase II와 interaction함. 즉, TFIIB는 TATA box(조금 더 정확히는 TATA box와 붙은 TBP)로부터 일정한 거리만큼 떨어진 곳에서 RNA polymerase II의 전사가 시작될 수 있도록 위치를 조정해주는 역할을 수행함. 결국 이전 chapter에서 봤던 것처럼 TATA box에 의해 transcription initiation site가 결정되는 이유는 TFIIB가 작용하기 때문임.
한편 TFIIB는 딱 5'→3' 방향으로 전사가 일어날 수 있도록 도와주는 역할도 함. 사실 그냥 TATA box로부터 특정 거리 떨어진 지점을 고르는 방식이었다면 TATA box보다 downstream 뿐만 아니라 TATA box보다 upstream에서도 전사가 개시될 수 있어야 . 그러나 TFIIB의 모양 자체가 비대칭적이어서 그 모양대로 TBP, RNA polymerase II가 연결되게 되면 무조건 TATA box보다 downstream 부분에서 RNA polymerase II에 의한 전사가 시작되게 됨.
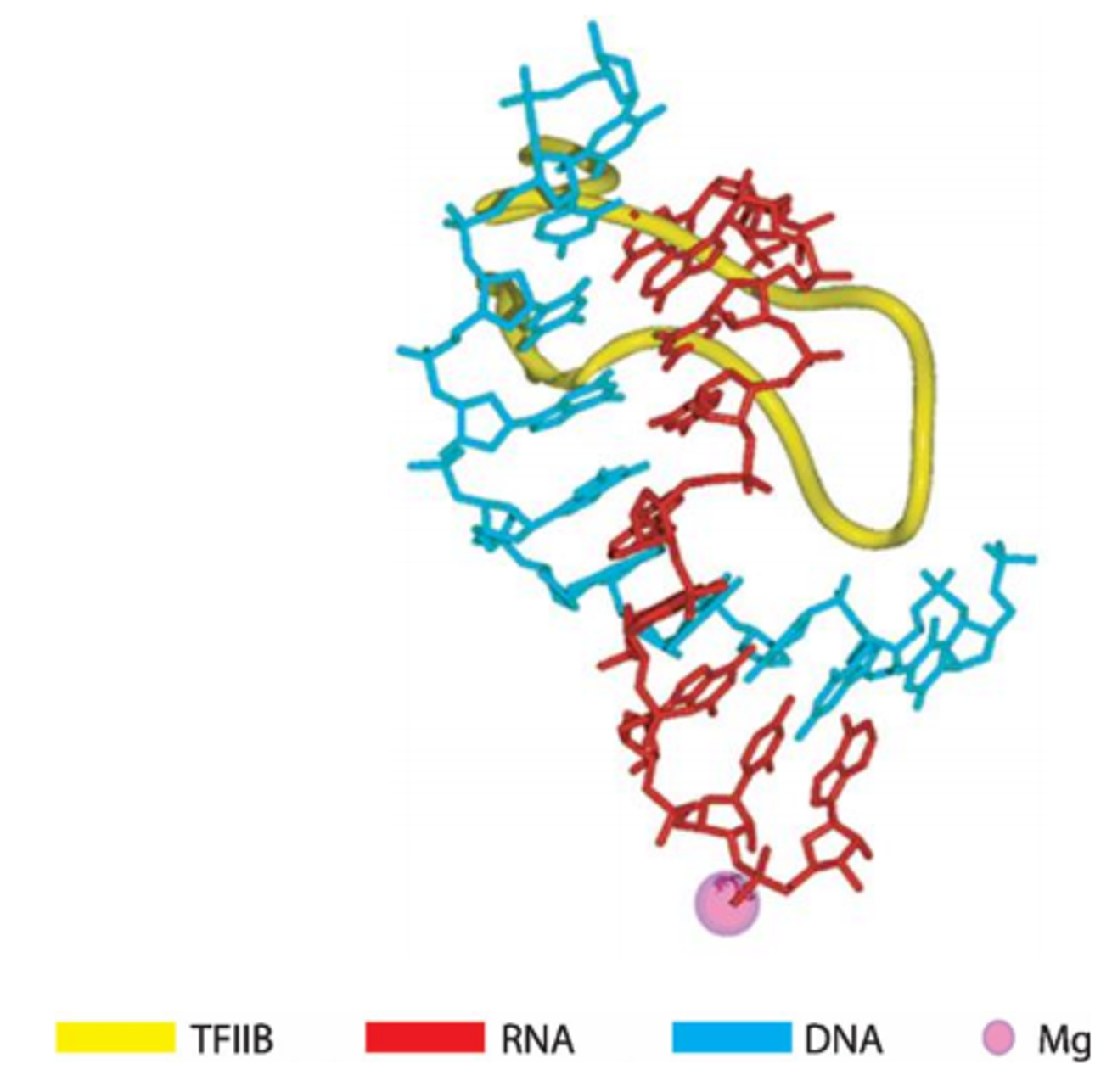
TFIIB가 가지고 있는 domain의 구조에 대해 조금 더 자세히 알아보자. 위 그림에서 분홍색 공으로 표시된 부분이 Mg2+가 존재하는 catalytic site이고, 이 부분을 포함한 위쪽 영역에(그림상에 나타나있지는 않지만) RNA polymerase가 위치하고 있음.
한편 TFIIB가 노란색 finger-like loop 형태로 표현되어 있는데, 이 때 TFIIB의 N-terminal이 RNA polymerase가 있는 구간까지 깊숙이 침투해있음을 알 수 있음. TFIIB loop의 N-terminal 끝 부분은 DNA와 상호작용해서 catalytic site의 위치를 더 잘 positioning해 줄 수 있게 되는데, human의 경우 TFIIB의 loop 끝부분에 + charge를 가지는 lysine으로 이루어진 2개의 lysine residue가 있어서 - charge를 띄는 DNA와 tight하게 interaction할 수 있게 됨. (즉, TFIIB가 catalytic site의 위치를 대략적으로, 하지만 견고하게 지정해줌)
참고로 yeast의 경우 TFIIB의 loop 끝부분이 aspartic acid와 같이 - charge를 띄는 acidic한 녀석들로 바뀌어 있음. 이 경우 DNA와의 interaction이 잘 일어나지 않게 됨. 그렇기에 yeast에서는 initiation site가 한 군데로 딱 정해져 있지 않음.
이 때문에 사람의 경우 TATA box보다 정확히 25~30bp 이후의 지점에서 전사가 개시되지만 yeast의 경우 TATA box로부더 40~120bp 사이의 지점에서 매우 variation이 큰 전사 개시가 나타나게 되는 것임.
한편 구체적으로 TFIIB와 RNA polymerase II가 상호작용하는 부위가 어디인지에 대해 알아보기 위해 아래와 같은 실험을 수행함.
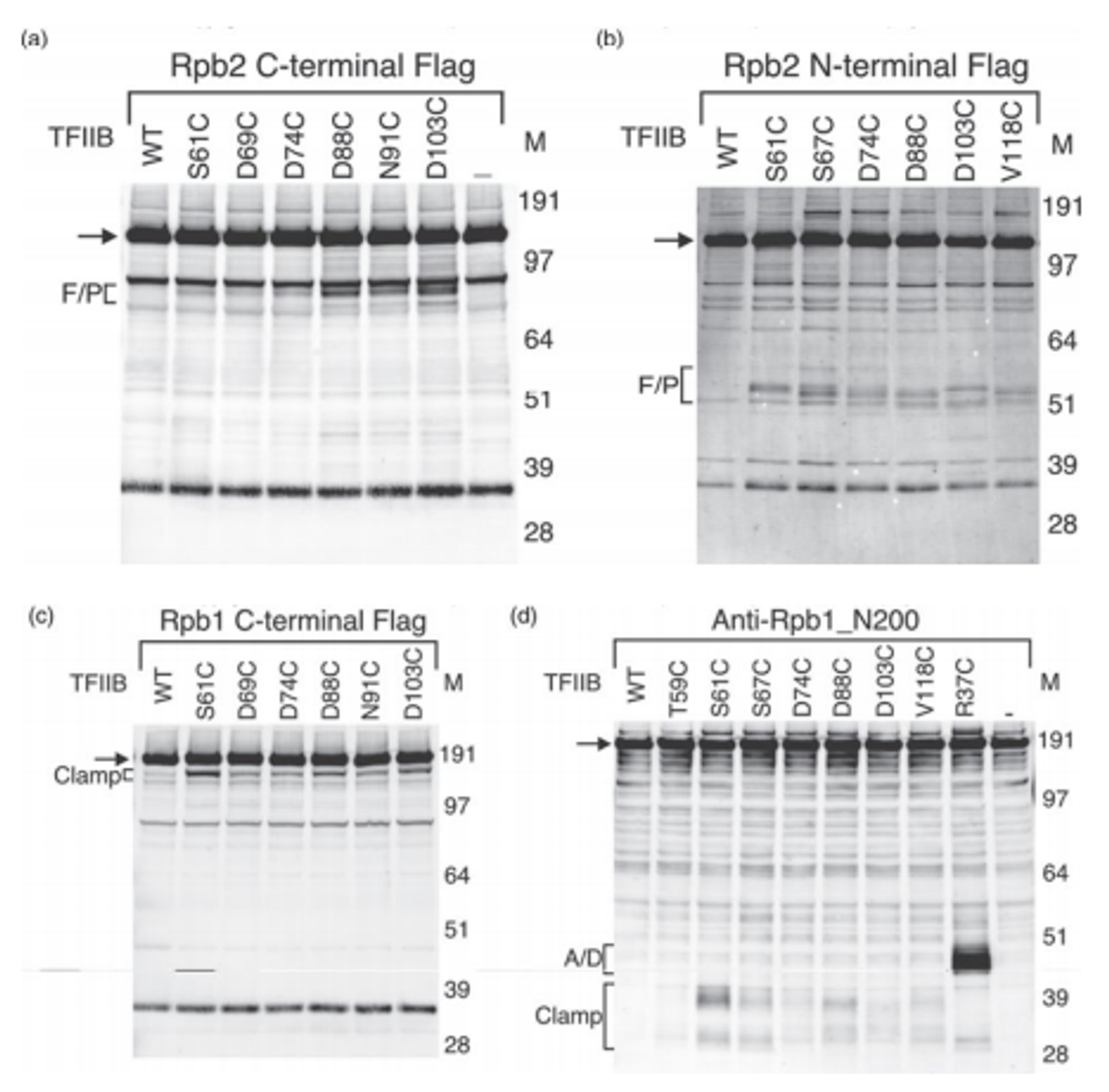
이 실험의 목적은 기본적으로 TFIIB와 Rpb2(RNA polymerase II의 subunit 중 하나)의 interface를 확인하는 것임. 이를 위해 TFIIB의 특정 amino acid를 site directed mutagenesis를 이용해 C(cysteine)로 바꿔줌. 한 예로 위 data에 등장하는 S61C는 61번째 serine을 cysteine으로 바꿔준 것을 알 수 있음.
그런데 치환된 C는 끝부분에 SH group을 가지고 있어서 매우 reactive함. 한편 이 reactive한 SH에 붙을 수 있는 물질인 Fe-BABE를 넣어주게 되는데, 그러면 C로 치환된 TFIIB residue에는 모두 Fe-BABE가 붙어있을 것임. 그런데 이 Fe-BABE는 자신의 주변부에 있는 15옹스트롬 범위 내에 hydroxyl radical을 형성시킬 수 있음.
그 결과 Fe-BABE의 주변부에 위치한 protein들이 다 잘림. 한편 TFIIB와 RNA polymerase II는 서로 같이 넣어줄 시 상호작용을 할 것이고, 그 과정에서 TFIIB의 binding residue와 특별히 가까워지는 RNA polymerase II 부위가 존재할 것임. 이 부위는 Fe-BABE에 의해 형성된 radical에 의해 잘릴 만큼 충분히 가까울 것이고, 결과적으로 protein cleavage가 일어날 것임. 이를 이용해서 TFIIB와 붙는 RNA polymerase II의 site가 어디인지를 알아낼 수 있음.
이제 본격적으로 결과를 살펴보자. 우선 gel 상에 나타나는 신호는 다 어떤 식으로든 Rpb-2, 혹은 Rpb-1을 표지한 것들임. 크게 두 가지 방법이 사용되었는데, Rpb에 C terminal flag를 달아주는 방식와 N terminal flag를 달아주는 방식들이 사용됨. 그리고 단백질을 분리해주는 실험이기 때문에 당연히 SDS-PAGE에 이은 western blotting을 수행함.
기본적인 idea는 wildtype TFIIB 혹은 아예 TFIIB를 넣어주지 않았을 때의 Rpb signal과 비교해서 site directed mutagenesis가 일어난 TFIIB를 넣어줬을 때 새로 생기는 band가 어디인지를 확인하는 것임. 위 그림상에 F/P, A/D, Clamp로 표시되어 있는 부분이 모두 새로 나타난 band 부분임.
한 예로 (b) data에서는 가운데 부분에 새로운 band가 나타남. 그러면 reference position과 비교해서 새로 잘려 생성된 대략적인 단백질 크기를 유추할 수 있음. 예를 들어 그 크기가 50 amino acid 정도였다고 치면 대략 N terminal로부터 50 amino acid 떨어진 부분에 TFIIB residue와 상호작용하는 부분이 있었다는 것을 알 수 있음.

이 결과들을 바탕으로 알아낸 RNA polymerase II 상의 TFIIB와의 binding site는 위 그림과 같음. 이 때 파란색으로 표시된 부분은 더 강하게 상호작용하는 부분, 하늘색으로 표시된 부분은 상대적으로 덜 강하게 상호작용하는 부분을 나타내며, (e)는 TFIIB의 finger 부분과 상호작용하는 부분, (f)는 TFIIB의 finger 뒤쪽 linker 부분과 상호작용하는 부분을 나타내주고 있음. (e, f에 대한 각각의 정보는 어느 부분을 C로 mutagenesis시켜줬는지를 알고 있으므로 당연히 얻을 수 있음)
다음 포스트에서는 TFIIH의 구조와 기능에 대해 알아보자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 11.4 : TFIIH의 구조와 기능 - 2 (0) | 2023.12.17 |
|---|---|
| [분자생물학] 11.4 : TFIIH의 구조와 기능 - 1 (0) | 2023.12.17 |
| [분자생물학] 11.2 : TFIID의 구조와 기능 - 2 (0) | 2023.12.17 |
| [분자생물학] 11.2 : TFIID의 구조와 기능 - 1 (0) | 2023.12.16 |
| [분자생물학] 11.1 : 진핵생물의 보편전사인자(general transcription factor) (0) | 2023.12.16 |