이번 포스트에서는 핵산의 구성요소 중 하나인 염기(base)에 대해 자세히 알아보자.

base들은 위와 같이 pyrimidine, 혹은 purine 중 하나로 나누어짐. 이들은 nitrogen이 포함된 heteroaromatic molecule들이며 거의 planar한 구조를 가지고 있음.
그리고 이들의 경우 250-270nm 상에서 UV light를 잘 absorption하는 성질이 있으므로 이를 이용할 시 DNA, RNA등의 양을 정량적으로 측정하는 것이 가능함.

purine, pyrimidine에 포함되는 base들은 위와 같음. 이 중 thymine은 DNA에서만 발견되고, uracil은 RNA에서만 발견됨.
base들은 일반적으로 pH 7에서 neutral molecule임. 그럼에도 이들은 내부에 N, O를 포함하고 있으므로 H-bond donor와 acceptor로 잘 기능할 수 있음.
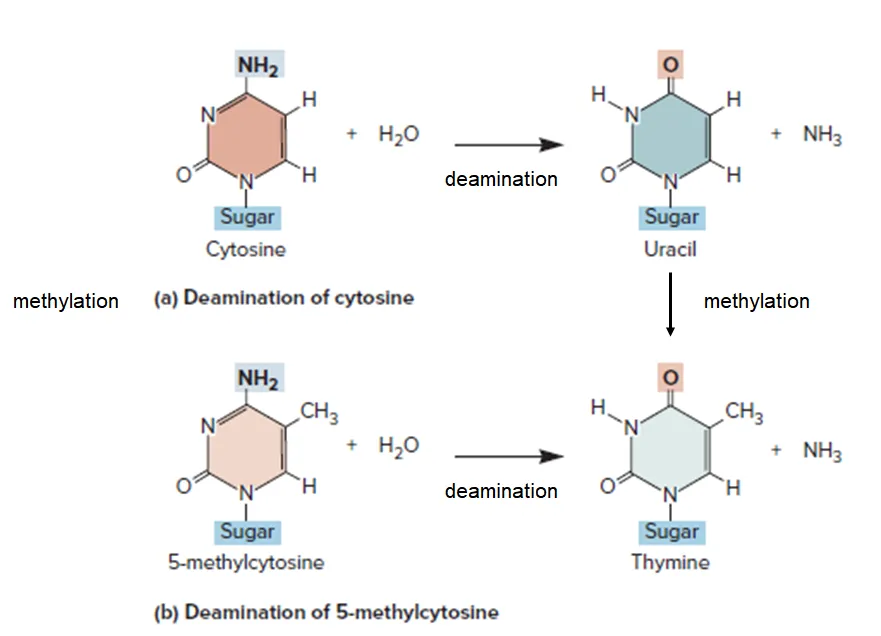
위 그림은 pyrimidine base의 특징들을 나타내주고 있음.
보면 cytosine이 deamination될 시 4번 위치에 있는 amine group이 떨어져나가며 uracil이 됨. 한편 uracil이 다시 methylation되면 thymine으로 변하게 됨.
한편 cytosine의 5번 base 위치가 methylation될 수 있으며 그 결과 5-methycytosine이 만들어짐. (이 과정은 DNA methyltransferase에 의해 촉진됨) 이 methylation은 transcription의 on/off를 결정할 수 있는 상당히 중요한 표지 중 하나임. 그런데 5'-methylcytosine이 deamination될 시 thymine이 될 수 있음.
이로부터 왜 RNA에 U가 있고 DNA에 T가 있는지에 대한 실마리를 얻을 수 있음.

위 그림에는 nucleotide, 그 중에서도 pyrimidine base의 생합성 과정이 나타나 있음.
보면 UMP, 즉 uridine base가 먼저 만들어진 후 이로부터 C와 T가 다 만들어질 수 있음. 따라서 RNA의 입장에서는 energy를 조금 덜 쓰고도 만들어질 수 있는 U를 이용하는 것이 더 유리했을 것임.
그런데 DNA에도 만약 U가 있다면, 이 경우 U가 그냥 원래부터 있던 정보인지, 아니면 C가 deamination된 결과 바뀐 것인지를 판단하기가 어려움. 따라서 정보의 제대로 된 보존과 관리를 위해서 DNA는 U(더 정확히는 dU)를 유전정보로 사용하지 않음. 그러다 보니 만약 DNA mutation에 의해 C가 dU로 바뀌면 이 dU를 제거하는 기작이 우리 몸 안에 존재하고 있음.

위 그림 B에 이 과정이 나타나 있음. 보면 우선 이상한 base를 제거해서 abasic site를 만들게 되고, 이 부분의 인지 결과로 phosphodiester bond가 끊어진 후 DNA repair 과정이 일어남. 이런 과정을 일컬어 base excision repair라 함.
이런 repair를 유발하는 녀석은 dU 뿐 아니라 G가 oxidation되어 만들어진 8oxoG 등의 abnormal base들임.

한편 base와 관련해 중요한 개념 중 하나인 tautomerization(토토머화)에 대해 알아보자. 위 그림을 보면 U 내부의 N, O 중 어디에 H가 붙냐에 따라 lactam, lactim, double lactim 구조가 형성될 수 있음. (이는 isomer와는 다름) 이러한 형태간의 interconversion을 Lactam-lactim tautomerism이라 부름.
한편 이 밖에 또 다른 대표적인 tautomerism으로 keto-enol tautomerism이 있음.
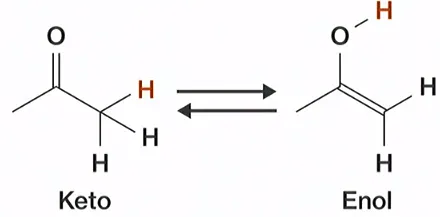
보면 위와 같이 keto에서의 C에 붙은 H가 O로 이동해 enol을 만드는 과정, 혹은 반대의 과정도 tautomerization임.

위 그림에는 앞서 말했던 base들이 absorption pattern이 나타나 있음. 보면 실제로 260nm 근방에서 absorption이 일어난다는 것을 알 수 있음. 이러한 absorption은 π→π*로의 transition 과정에서 일어나는 것임.
한편 absorption과 관련해서 Beer-Lambart law인 A=εbc를 이용하면 dsDNA, ssDNA, ssRNA 각각에서의 c를 알 수 있음. (A는 측정값으로부터 logI/I0 식을 통해 계산되어 나오고 b도 알며, 이 때 ε(빛을 흡수할 수 있는 능력)도 알고 있다고 가정)
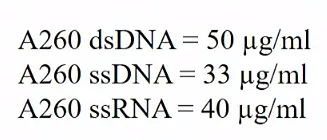
위 그림은 참고로 dsDNA, ssDNA, ssRNA의 c를 나타내주고 있음.
한편, 단백질 중 tryptophan, tyrosine의 경우에도 280nm 부근에 대해 maximum absorption을 보임.

따라서 이제 A260/A280의 비율을 계산해 볼 시 purity를 계산하는 것이 가능함. 참고로 pure한 DNA, RNA의 경우 이 값이 2가 나오고 pure한 protein의 경우 약 0.6이 나옴.
다음 포스트에서는 핵산을 이루는 당인 pentose sugar에 대해 알아보자.
'전공자를 위한 생물학 > 생화학' 카테고리의 다른 글
| [생화학] 7.4 : 인산기(phosphate group) (0) | 2024.11.08 |
|---|---|
| [생화학] 7.3 : 오탄당(pentose sugar) (0) | 2024.11.08 |
| [생화학] 7.1 : 핵산(nucleic acid) (0) | 2024.11.08 |
| [생화학] 6.6 : 렉틴(Lectin) (0) | 2024.11.08 |
| [생화학] 6.5 : 당지질(glycolipid) (0) | 2024.11.08 |