이번 포스트에서는 유전자 재조합 기술에 대해 알아보자.
인간의 whole genome 정보를 알게 된 것은 2019년 기준으로 대략 20년 전에 해당함. 실제로 전세계적으로 수행한 human genome project와 회사 celera의 노력으로 15년만에 human genome을 sequencing할 수 있었음.
재조합 유전자(recombinant DNA)는 자연계에서는 함께 존재하지 않는, 서로 다른 생물학적 근원으로부터 유래된 DNA분자들을 인위적으로 연결해 만든 유전자를 의미함. 이런 재조합 유전자를 이용하면 새로운 protein을 만들어낼 수 있음. (이 말인즉슨, 새로운 생명현상을 유발시킬 수 있음)
이 때 재조합 유전자의 copy를 clone이라 부르며, 이 copy를 만들어내는 과정을 cloning이라 함.
그렇다면 재조합 유전자는 어떻게 제작할 수 있을까. 유전자 재조합 기술은 크게 3가지 단계를 거쳐 진행되게 됨.
cleavage by restriction enzyme
첫 번째 단계는 제한효소(restriction enzyme)를 활용하여 특이적인 DNA 조각을 생산하는 것임. 이 때 제한효소는 원래는 bacteria가 가지고 있는 효소이며, bacteria는 외부 virus 등이 침략할 시 restriction enzyme을 이용해 viral DNA를 cut해주게 됨.
제한효소는 DNA의 특정한 인식 염기서열(restriction site)을 인지하여 결합하고 잘라내게 됨. 만약 우리가 cloning하고자 하는 서열 내에 restriction site가 없을 경우 PCR을 통해 restriction site를 인위적으로 넣어주고 난 뒤 잘라주게 됨.
restriction site는 일반적으로 짝수개의 염기로 구성(4, 6, 8개가 대부분)되어 있음. 이 때 당연히 길이가 가장 긴 8개짜리 restriction site가 가장 특이적이겠지만, 이 서열을 합성해서 넣을 때 드는 비용도 가장 비싸다는 것이 단점임. 따라서 일반적으로는 6bp 길이의 restriction site를 사용해주게 됨.

restriction enzyme의 종류에 따라, 위와 같이 만들어지는 cleavage 산물의 모양새가 달라질 수 있음. 이들을 크게 나눌 시 cohesive end(sticky end)가 만들어지는 경우와 blunt end가 만들어지는 경우로 나눌 수 있음. 이 때 cohesive end는 제한효소가 특정 부위를 대각선으로 cut해서 튀어나온 형태의 end가 만들어지는 것이고 blunt end는 제한효소가 특정 부위를 일직선으로 cut해서 뭉툭한 end가 만들어지는 것임. 그런데 이 때 cohesive end는 DNA ligase에 의해 잘렸던 조각이 붙을 때 잘렸던 조각들끼리 상보적으로 다시 붙을 수 있는 반면 blunt end는 DNA ligase에 의해 붙을 때 random하게 다른 녀석과 붙어버릴 수 있음. 따라서 blunt end를 만들어내는 restriction enzyme은 생명공학 실험에서는 잘 사용되지 않음.
한편, 위 그림을 자세히 보면 restriction enzyme이 인지하는 염기서열이 대부분의 경우 가운데를 중심으로 서로 상보적임을 알 수 있음. (이런 관계를 만족하기에 대부분의 경우 restriction site의 bp 갯수가 짝수개인 것)
그리고 restriction site의 bp 갯수가 4개인 경우, 이 site가 너무나 많이 존재할 수 있고 그러다 보니 우리가 원하는 부위 이외의 나머지 부분도 restriction enzyme에 의해 잘릴 수 있어서 cloning을 할 때는 잘 사용되지 않는 편임. 대신 4개 길이의 site를 자르는 restriction enzyme은 DNA를 random하게 자를 시 많이 사용됨. (DNA를 random하게 자르는 방식을 사용하는 실험 기법의 대표적인 예가 바로 celera에서 sequencing을 위해 사용했던 whole genome shotgun sequencing method임)
참고로 유전자 재조합을 할 시 서로 다른 종류의 제한효소를 써서 다른 종류의 cohesive end가 만들어진 경우에도 cohesive end 중 일부(예를 들어 4개정도)가 서로 상보적이라면 이들 end를 서로 ligase로 붙일 수 있음. (다만 이렇게 붙어서 형성된 product는 완전히 다른 restriction site를 가질 것이므로 다시 앞서 사용되었던 제한효소로 자르지는 못할 것임) 이런 식으로 여러 종류의 제한효소를 같이 사용하는 경우에는 restriction enzyme의 작동 pH 조건이 비슷한 녀석들을 사용하는 것이 좋을 것임.
DNA ligation
그 다음에는 만들어진 조각들을 vector와 연결시키는 단계가 이어지게 됨. (이 단계를 거치지 않는다면 우리가 만들어낸 조각들은 host cell의 defense mechanism에 의해 파괴되어 버릴 것임)

위 그림에 ligation step의 대략적인 모식도가 나타나 있으므로 참고할 것.
이 때 vector란 cloning되기를 바라는 DNA 조각이 host cell 내에서 복제될 수 있도록 도와주는 carrior DNA임. (vector는 우리가 사용하고자 하는 host cell system에 따라 각기 다르게 맞춰서 사용하게 됨)

위 그림에는 host cell이 bacteria일 때 vector로 가장 대표적으로 사용되는 plasmid의 구조가 나타나 있음. 보면 이 녀석을 vector로 사용하기 위해서 당연히 origin of replication(ori)이 있어야 하며, 다양한 제한효소자리가 모여있는 multiple cloning site(MCS)가 존재해서 실제로 실험할 때는 우리가 사용하고 싶은 restriction enzyme을 골라 실험할 수 있게끔 setting되어있음.
그 밖에 vector는 특정한 선택적 유전자 marker를 지녀서 vector를 가진 host를 vector를 가지지 못한 host로부터 구분할 수 있게끔 구성되어 있음.
이를 위해 흔히 삽입되는 서열의 종류를 2가지로 나눌 수 있음. 첫 번째는 항생제 내성 gene이 vector에 포함되어 있는 경우임. 그럴 경우 특정 항생제 환경에서 host cell을 배양할 시 vector가 포함되어 있어 항생제 내성이 있는 경우만 살아남게 될 것임. (특히 이런 경우 host cell 입장에서는 vector 내부에 생존에 필수적인 유전자가 들어있으므로 vector를 변형하거나 제거하지 않고 온전히 잘 보존하고 복제시킬 것임)
두 번째는 특정 nutrient 결핍 시에도 잘 살게끔 해주는 gene이 vector에 포함되어 있는 경우임. 이 경우 host cell을 미리 특정 nutrient 결핍 환경 하에서 못 살아남도록 만든 다음에 vector를 transformation하게 됨. 그럴 시 특정 영양분 미포함 배지 하에서 vector가 포함된 host cell들만 살아남게 될 것임.
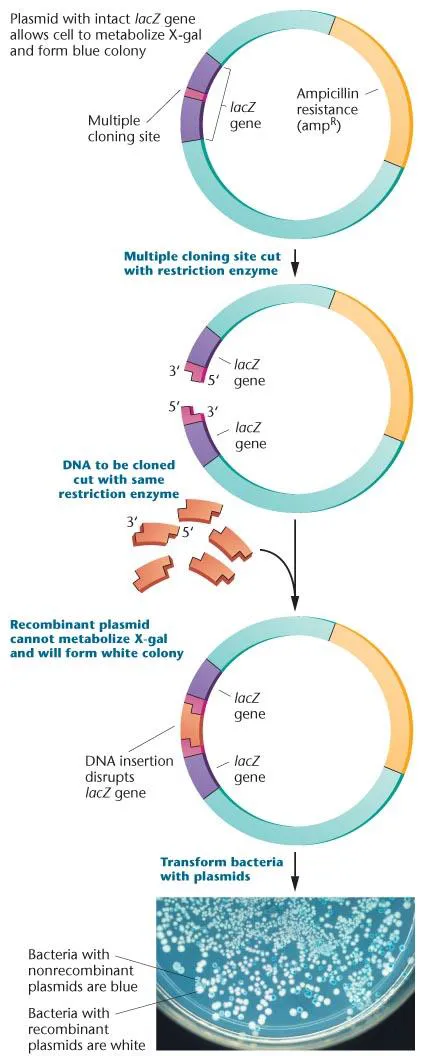
한편, transformation된 vector가 self-assembly된 vector인지 아니면 정말로 우리가 원하는 DNA가 삽입된 vector인지를 확인하기 위해 대표적으로 사용가능한 방법이 바로 위 그림에 나타나 있는 blue-white selection임. 이를 위해 위 그림에도 나타나 있는 것처럼 lacZ gene이 vector 내에 포함되어 있으며, 딱 lacZ gene 가운데에 MCS가 위치하고 있음. 따라서 DNA가 vector 내에 삽입될 시 lacZ gene이 벌어져서 제기능을 못하게 됨.
일반적으로 lacZ gene이 정상적으로 발현될 시 X-gal 배지 하에서 푸른색으로 보이게 되고(이 경우 non-recombinant vector), lacZ gene이 정상적으로 발현되지 않을 시 X-gal 배지 하에서 흰색으로 보이게 됨. (이 경우 recombinant vector) 따라서 이러한 system 하에서 흰색 colony만을 선별할 시 우리가 원하는 recombinant DNA를 포함하고 있는 host cell만을 선별해낼 수 있음.
transformation
이 단계에서는 만들어진 재조합유전자를 host cell에 transfer시켜서 다량의 copy가 host cell 안에서 만들어지게끔 해줌. (즉, host cell이 특정 DNA를 복제해주는 역할을 함)
일반적으로 E. coli에 transformation을 시키는 과정을 예로 들어 생각해보자. 일단 이 때 competent cell을 사용하는데, 이 때 competent cell이란 물리, 화학적으로 DNA 도입능력이 강화된 cell을 의미함. 그리고 이러한 competent cell에 transformation을 시키고자 할 때는 보통 CaCl2를 처리한 환경에 노출시키는데, 그럴 시 Ca2+가 cell membrane의 - charge를 상쇄시키는 역할을 해서 - charge인 DNA가 쉽게 접근할 수 있게 됨.
이후 competent cell을 재조합된 plasmid와 섞고 얼음에 30분 보관함. (그럴 시 competent cell 막 가까이에 재조합 plasmid가 위치하게 될 것) 그 다음에 42도에서 heat shock을 가해줌. 이 때 주로 37도 정도 되는 사람 대장에서 서식하는 대장균에게 42도는 매우 harsh한 condition이므로 cell membrane이 찢어지며 plasmid가 cempetent cell 내부로 들어가게 됨. 이후 1시간동안 항생제의 일종인 암피실린이 없는 환경하에서 키움. 이 때 일반적으로 대장균이 한번 분열하는데 40분이 걸리지만, 그냥 stress상황인 것을 고려해 넉넉히 1시간을 준 것임. 그 다음에 다시금 암피실린 배지에서 배양하며 selection을 거치게 됨.
다음 포스트에서는 DNA library에 대해 알아보자.
'전공자를 위한 생물학 > 유전학' 카테고리의 다른 글
| [유전학] 10.3 : DNA sequencing (0) | 2024.11.02 |
|---|---|
| [유전학] 10.2 : DNA library, PCR (0) | 2024.11.02 |
| [유전학] 9.3 : 헌팅턴병, 정신분열증, 자폐증 (0) | 2024.11.01 |
| [유전학] 9.2 : 행동 우선 접근법과 유전자 우선 접근법 (0) | 2024.11.01 |
| [유전학] 9.1 : 행동유전학(behavioral genetics)의 기초 - 2 (0) | 2024.11.01 |