이번 포스트부터는 번역의 종결(termination) 과정에 대해 알아보도록 하자.
우리는 익히 termination을 유발하는 3개의 codon을 일반생물학에서 배워서 알고 있음. 3개의 종결 코돈(stop codon)이 그것인데, amber codon으로 불리는 UAG, ochre codon으로 불리는 UAA, opal codon으로 불리는 UGA가 여기에 해당함.
이들의 존재에 대해 처음 알게된 것은 이들을 넣어줬을 때 발생하는 nonsense mutation과 관련된 study를 통해서임. (nonsense mutation은 아예 단백질이 짧게 만들어져서 기능하지 못하는 경우를 말하고, missense mutation은 중간에 하나의 amino acid가 완전히 잘못 들어가는 돌연변이를 말함)

위 그림에 소개되어 있는 논문에서는 nonsense mutation이 유발된 bacteriophage를 E. coli에 감염시킨 뒤, 다시금 E. coli에 mutation을 시켜서 원래라면 nonsense mutation에 의해 죽어야 할 개체를 살리는, revertant mutant를 찾아 분석하는 연구 flow를 바탕으로 stop codon의 존재에 대해 밝힘.

방금 글로 설명한 flow가 위 그림에도 나와있음. 보면 우선 gene의 특정 gene 부위를 delete해서 두 gene 중 하나의 activity를 turn off시킨 후 amber mutation에 의해 nonsense mutation이 유발된 bacteriophage를 제작함. 일단 deletion에 의해서는 A gene의 발현이 turn-off 될 것이고 추가적으로 만들어준 amber mutation에 의해서는 A 뿐만 아니라 B gene의 발현도 turn-off됨. 이후 이 bacteriophage를 E. coli에 넣어 배양하게 되는데, 이 과정에서 잘 살아남는(amber suppressor strain) E. coli를 뽑아서 분석해봄.

그 결과 위 그림과 같이 E. coli 자체의 tRNA에 mutation이 일어나 원래라면 결합하지 못하는 stop codon에 결합할 수 있게 된 tRNA가 생기게 되었고(이 tRNA가 nonsense mutation의 suppressor로 작용), 그 결과 revertant mutant가 발생하게 되었다는 것을 알게 됨. 이를 통해 stop codon의 존재에 대해 알게 됨.
그렇다면 구체적으로 nonsense sequence에 대해서는 어떻게 알게 되었을까. 대표적으로 amber codon인 UAG 서열에 대해 알게 된 실험 과정에 대해 알아보자.

위 그림에 나타나 있는 실험은 기본적으로 wild-type에 nonsense mutation을 유발해 nonsense mutant를 만든 후(stop codon 포함) 다시금 nonsense mutation을 중화시켜(stop codon 중 하나를 다른 것으로 바꿔) revertant를 만든 후 이 revertant protein의 amino acid sequence를 분석하는 방식으로 수행됨.
구체적으로 이 때 사용한 protein은 alkaline phosphatase(AP)이며, 정상 단백질과 revertant 단백질 각각을 Trp(K, R과 같은 단백질의 앞쪽을 cut) 등을 처리해 fragment로 만든 후 2D gel chromatography한 결과가 위와 같음. 이 때 실선으로 나타낸 것이 정상 단백질, 점선으로 나타난 것이 revertant이며, 보면 revertant의 경우 2D 상에서 shift가 일어났음을 알 수 있음. 이 때 shift가 일어난 이런 revertant dot만 고른 뒤에 이들에 대해 N-terminal sequencing을 수행해 봄.
이후 정상 단백질의 amino acid sequence와 revertant의 amino acid sequence를 비교한 결과가 아래 표와 같음.

위 표 중에서 특히 peptide 1의 data들을 서로 비교해보자. 일단 normal한 경우에는 lys, try, gly, leu의 비율이 1:1:1:1로 나타남. 그런데 R3a의 경우에는 lys, gly, leu, ser이 1:1:1:1로 나타남. 이를 통해 try가 ser로 바뀌었다는 것을 알 수 있음. 유사하게 R4a의 경우에는 try가 glu로 바뀌었음을 알 수 있음.
이와 같이 normal과 revertant의 비교에서 달라진 position이 try 부위임을 확인하고, 이 부분에서 달라진 amino acid들 모두에 대한 codon을 나열해보면 결과적으로 아래와 같은 map을 그려볼 수 있게 됨.

그런데 이 때 normal에서의 amino acid 혹은 revertant의 amino acid를 coding하는 codon sequence에 다시금 mutation이 일어나게 되면 nonsense mutation을 유발하는 sequence로 다시 변할 수 있음. 따라서 결과적으로 위 그림과 같이 unknown nonsense codon을 중심으로 wild-type, revertant amino acid codon의 map이 그려짐. 이제 각각의 sequence들 중 중복되는 part를 바탕으로 원래 amber codon을 추측해볼 수 있음.
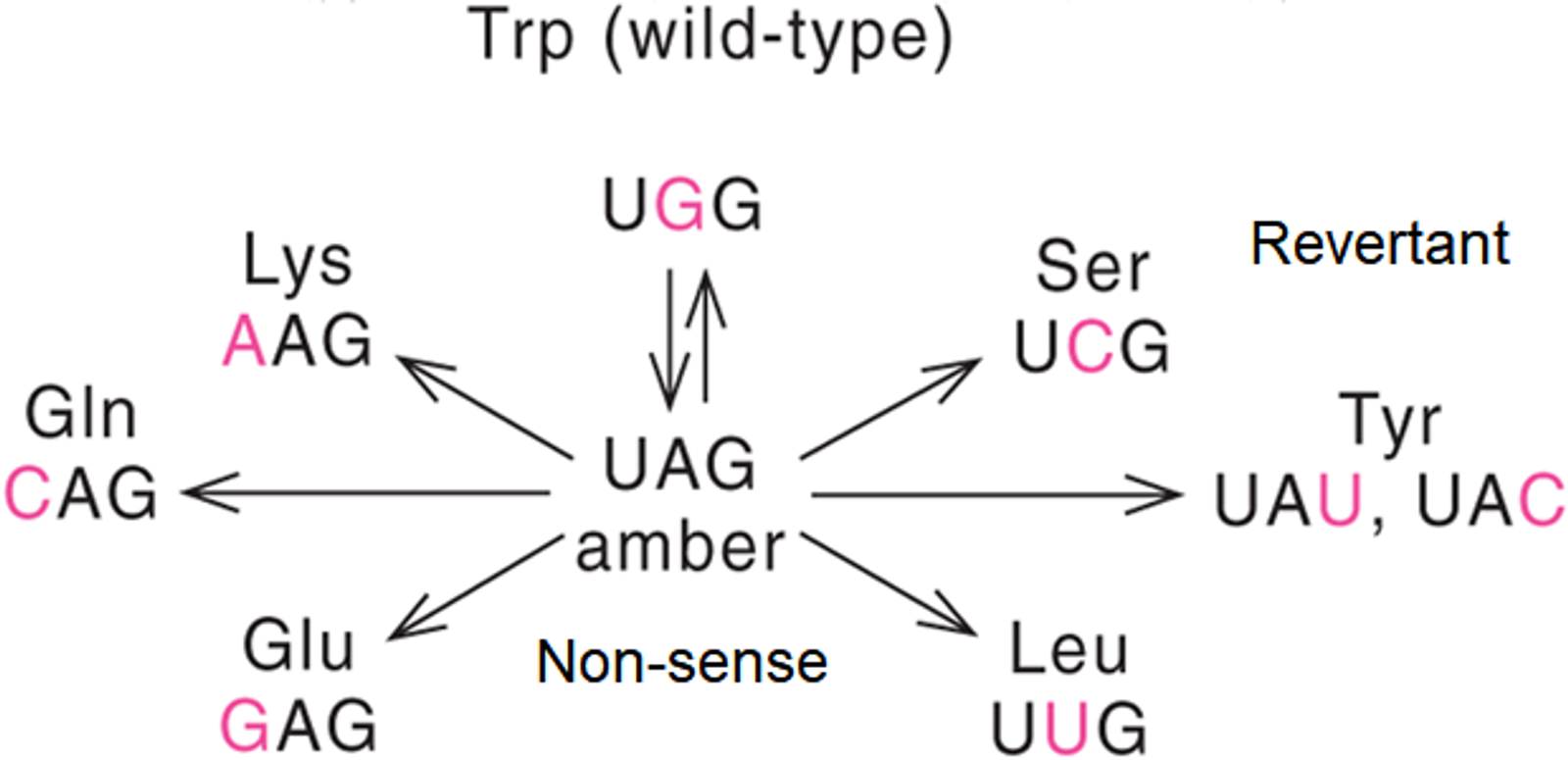
이렇게 해서 amber codon의 sequence를 알아냄. (더 정확히 표현하자면 amber mutant를 일으키는 codon이 UAG라는 것을 알아냄)
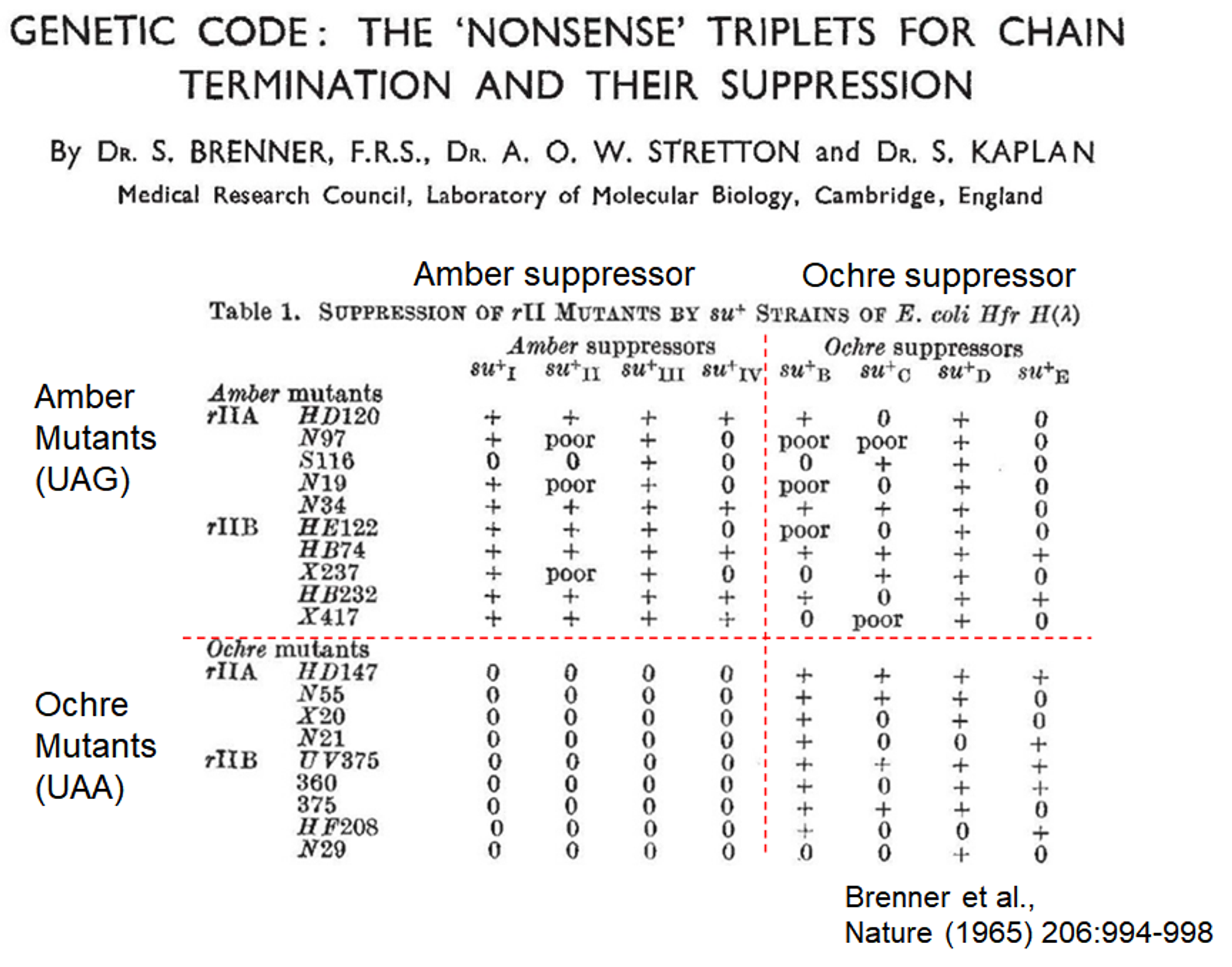
앞서 봤던 것처럼 amber mutant에서는 당연히 amber suppressor를 만들어줄 시 revertant가 생성됨. 그런데 amber suppressor를 만들어줘도 revertant가 형성되지 않는 mutant가 발견됨. 그 중 하나가 바로 Ochre mutant이며, 이 녀석의 경우 Ochre suppressor를 만들어줘야 비로소 revertant가 생성됨.
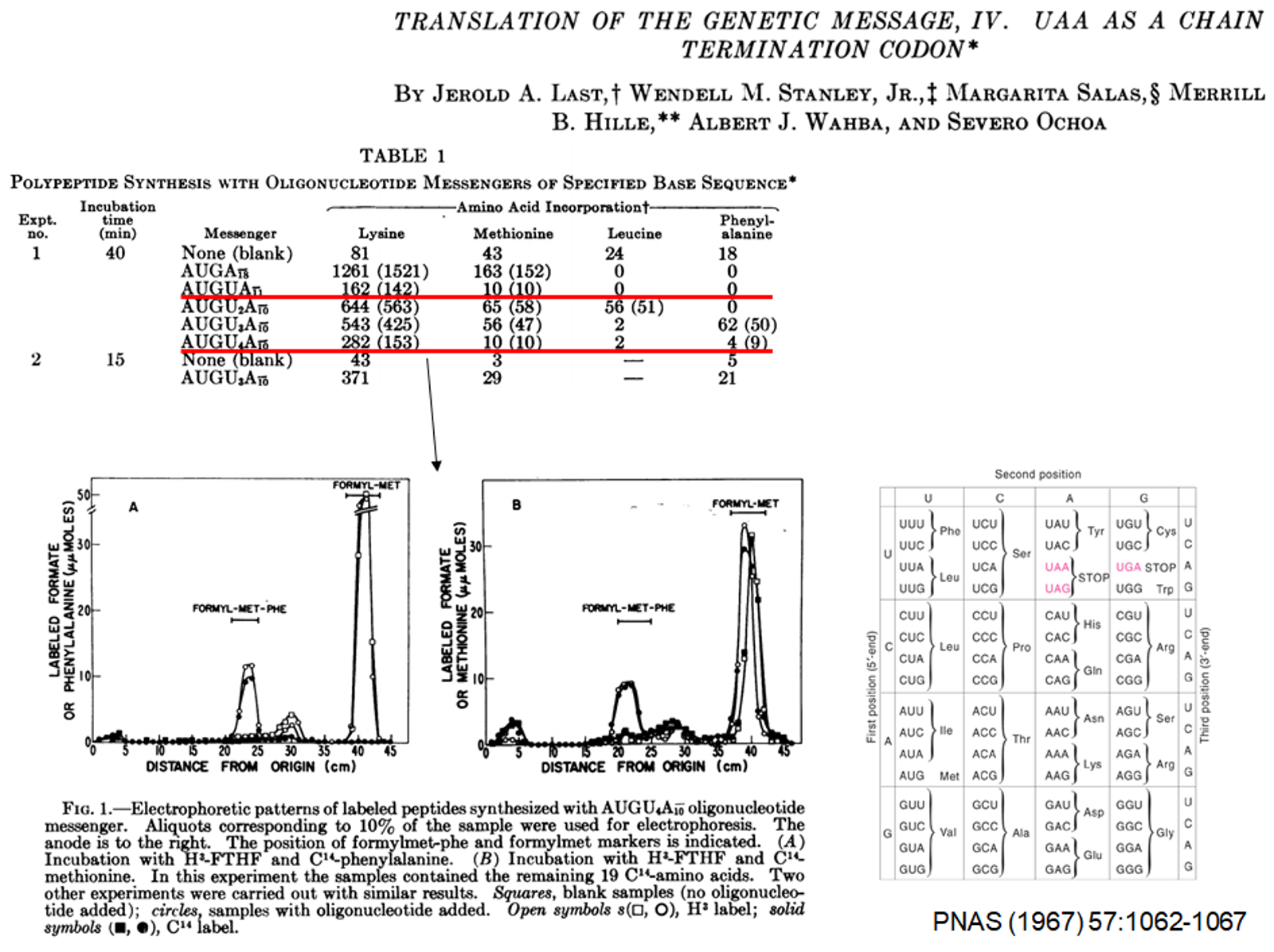
구체적으로 ochre codon의 sequence정보를 알아낸 실험이 위에 나타나 있음. 이 경우에는 protein sequence를 분석하는 방법이 아닌 다른 방법을 사용함.
이 경우에는 AUGAAAA...의 서열, AUGUAAAAAA....의 서열, AUGUUAAA...의 서열, AUGUUUAAA....의 서열, AUGUUUUAAAA...의 서열 등을 사용해 실제로 이들을 발현시킨 뒤 합성된 peptide를 이루는 amino acid 조성을 조사해봄. (참고로 AUG는 Met, AAA는 lys을 coding함) 그랬더니 특별히 AUGUAAAA..와 AUGUUUUAAA...에서만 나머지에 비해 턱없이 적은 amino acid 비율(특히 턱없이 적은 lys비율)이 관찰됨. 그런데 결국 이들을 AUG부터 3개씩 끊어보면 AUG/UAA/..., AUG/UUU/UAA/... 으로 이들 둘에 있어 겹치는 codon은 UAA라는 것을 알 수 있음.
이를 통해 결국 UAA가 다음 codon의 translation을 막아주는 효과를 나타낸다는 것을 알게 되었고, 결과적으로 UAA가 ochre codon이라는 것을 알게 됨.
이 밖에 Opal mutation의 경우에도 이들의 영향을 상쇄시킬 수 있는 opal suppressor의 발견을 바탕으로 normal과 revertant의 protein sequence 비교를 통해 opal codon이 UGA라는 것을 알아냄.
다음 포스트에서는 release factor(방출인자)에 대해 알아보도록 하자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 18.3 : 번역 종결(translation termination) - 3 (0) | 2024.08.10 |
|---|---|
| [분자생물학] 18.3 : 번역 종결(translation termination) - 2 (0) | 2024.08.10 |
| [분자생물학] 18.2 : 번역 신장 (translation elongation) - 3rd step (0) | 2024.08.09 |
| [분자생물학] 18.2 : 번역 신장 (translation elongation) - 2nd step (0) | 2024.08.09 |
| [분자생물학] 18.2 : 번역 신장 (translation elongation) - 1st step (0) | 2024.08.09 |