이번 포스트에서는 원핵 생물에서 일어나는 transcription elongation 과정에 대해 알아보자.
elongation이 일어날 때는 core polymerase가 중요한 역할을 하는데, 그 중에서도 beta, beta' subunit이 phosphodiester bond를 형성하는 데 있어 매우 중요한 역할을 함.
진핵생물이 가지고 있는 RNA polymerase에 비해 원핵생물이 가지고 있는 RNA polymerase는 종류도 많지 않고, subunit 하나하나를 purify한 다음 이들을 다시 in vitro 하에서 reconstitute하는 것도 가능함. 실제 예시를 하나 살펴보자.
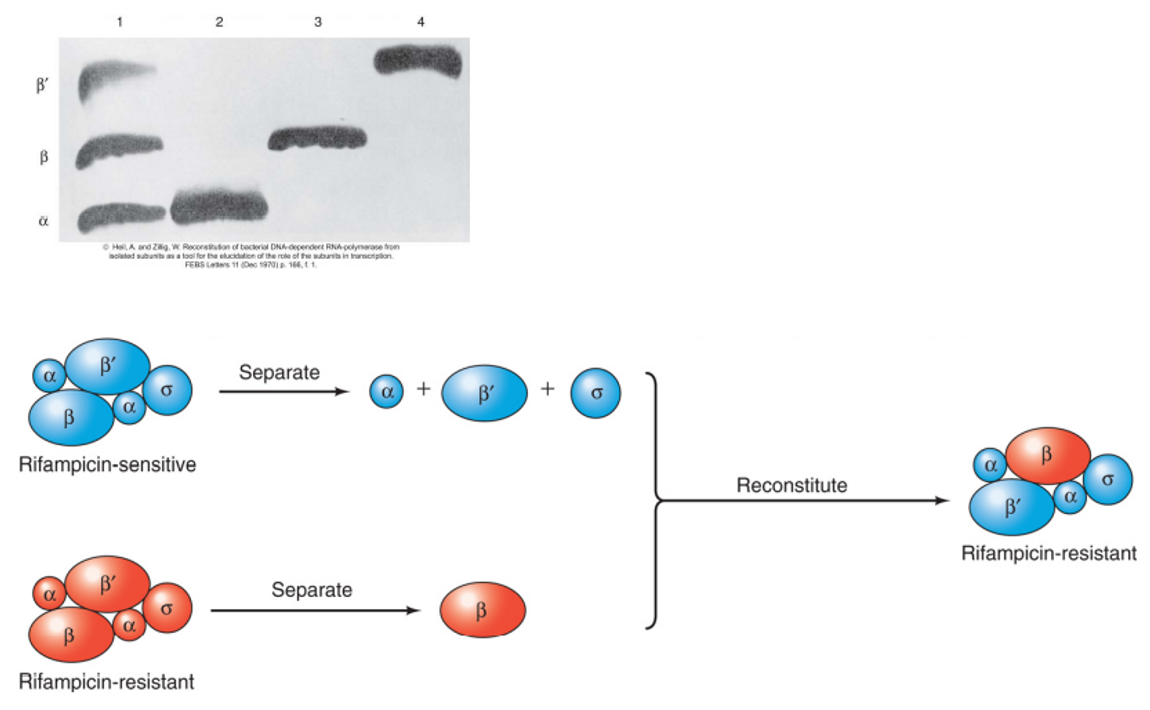
위 그림은 이전 포스트에서 살펴본 Rifampicin에 대해 저항성을 가지는 종이 사용하는 RNA polymerase 중에서 Rifampicin 저항성을 부여하는 부위가 어디인지를 찾기 위해 수행한 실험을 나타내고 있음.
이 때 Rifampicin-sensitive한 종에서 분리한 RNA polymerase로부터 alpha, beta', sigma subunit을 분리한 후 Rifampicin-resistant한 종에서 분리한 RNA polymerase로부터 \beta subunit을 분리하여 이들을 다시 reconstitute해 줄 경우 새로 생성된 RNA polymerase도 Rifampicin-resistant하다는 것을 알 수 있음. 따라서 이 결과를 통해 beta subunit이 Rifampicin에 대한 저항성을 부여한다는 것을 알 수 있음.
한편, 직전에 beta, beta' subunit이 phosphsdiester bond의 형성에 관여한다고 했었는데, 이러한 사실을 어떻게 알게 되었는지에 대해 알아보자.

위 그림에 대략적인 실험의 과정이 나타나 있음. 이 때 사용한 reagent가 위 그림 (a)에 나타나 있는데, 이 녀석은 일종의 ATP analog임. 이 녀석이 elongation에 사용되면, 이 녀석의 고유한 성질(amino group과 공유결합을 형성해서 cross-linking해줌)에 의해 이 녀석 주변에 접촉한 polymerase subunit들이 다 cross-linking을 통해 연결된 채로 RNA 사이에 끼어들어가있음. 한편 elongation 과정에서 사용한 NTP들은 모두 radioactive하게 만들어줬기 때문에 결국 위 그림 오른쪽과 같이 elongation 과정에서 어떤 subunit이 관여했는지를 알 수 있음. 그 결과 beta, beta' subunit이 elongation, 그 중에서도 phosphodiester bond formation 과정에서 역할을 한다는 것을 확인 가능함. (정리하자면 위 그림 오른쪽에서 gel을 통해 분리한 대상은 protein, 이 protein을 눈에 보이게 해준 것은 결합된 radioactive nucleotide, protein과 nucleotide 사이에 cross-link를 형성해 준 것은 ATP analog reagent임)
다음으로 elongation이 진행될 때 RNA-DNA 간에 생기는 hybrid가 있을텐데, 이 hybrid의 길이가 어느 정도인지를 알아보자.
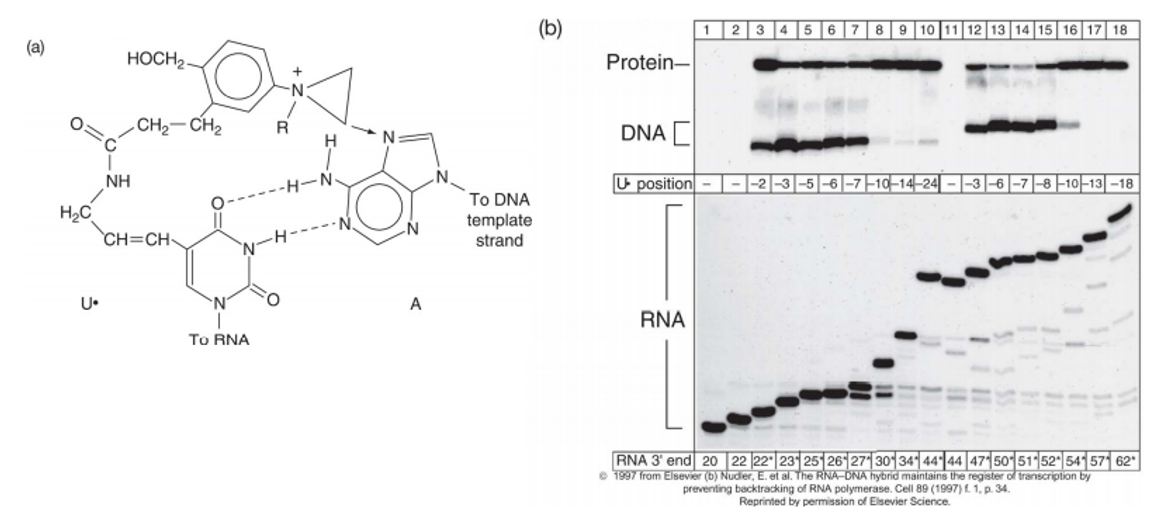
이를 알아보기 위해 수행한 실험이 위 그림에 나타나 있음. 이 때 실험 결과가 복잡하므로 위 그림 (b)의 10번까지만 이해해도 충분함. 이 때 사용한 reagent는 (a)에 나타난 것과 같은 U*이며, 이 녀석 또한 일종의 cross-linking agent임.
한편 (b)에서 U* position으로 표시된 부분은 RNA의 3' 끝을 -1로 두는 식으로 U*의 위치를 표현한 것임. 이 때 이 실험은 사실 sequence를 다 알고 있는 상태에서 진행한 것인데, 하나하나 nucleotide를 넣어가며 그때 U*에 의해 같이 붙잡혀오는 protein, DNA들이 있는지 여부를 본 것임. 보면 -2부터 -24 position에 걸쳐 protein이 붙잡혀오게 되며(RNA polymerase) -2부터 -10 가량에 걸쳐 DNA가 같이 붙잡혀옴. 이후에는 붙잡혀오지 않는데, 이는 아마도 합성이 계속 진행되면서 바깥쪽으로 5' 말단이 노출되었고, 이곳에 U*이 위치하게 되어서인 것으로 추정됨.
아무튼 이 결과를 통해 약 7bp 가량의 길이에 대해 RNA-DNA hybrid가 생성된다는 것을 확인할 수 있었음.
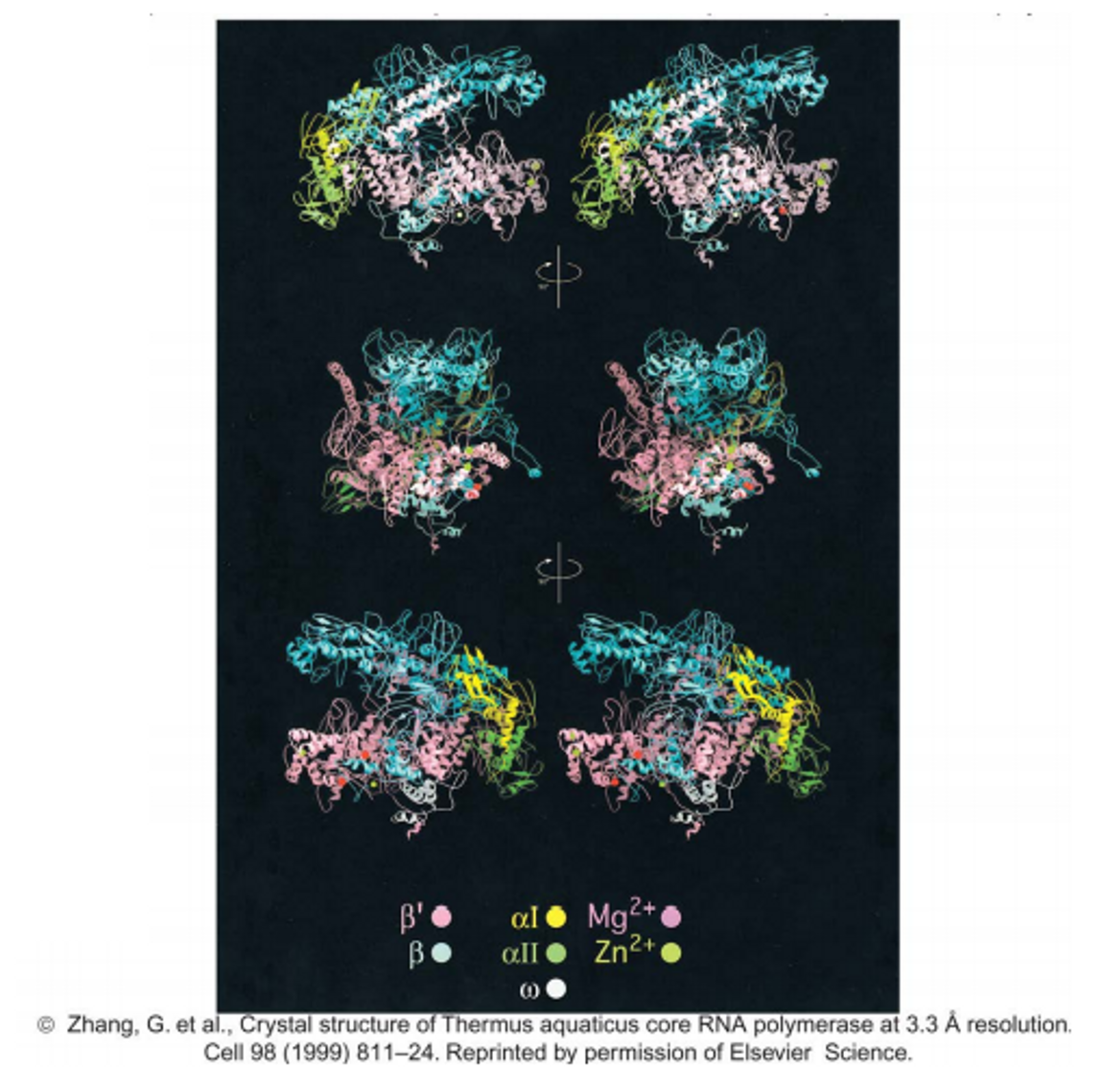
한편 X-ray crystallography를 이용해서 core polymerase의 구조를 실제로 파악할 수 있음. 이 때 core polymerase와 같이 작용하는 각종 ion들이 어디에 위치하는지(예를 들어 Mg2+는 Asp 쪽에 위치) 등도 알 수 있음.

한편 동일한 방법으로 아예 DNA와 결합한 상태의 holoenzyme 구조를 위와 같이 파악할 수도 있음. 이를 통해 각종 molecular biology tool로 간접적으로 확인했던 사실들을 실제로 확인할 수 있음. 더 나아가서 위 구조를 통해 Y253, F248, W256이 가지는 aromatic group이 promoter의 melting을 유발한다는 사실도 알 수 있으며, sigma factor 내부에 존재하는 K241, R237과 같은 basic amino acid들이 promoter와의 결합을 촉진한다는 사실도 알 수 있음.
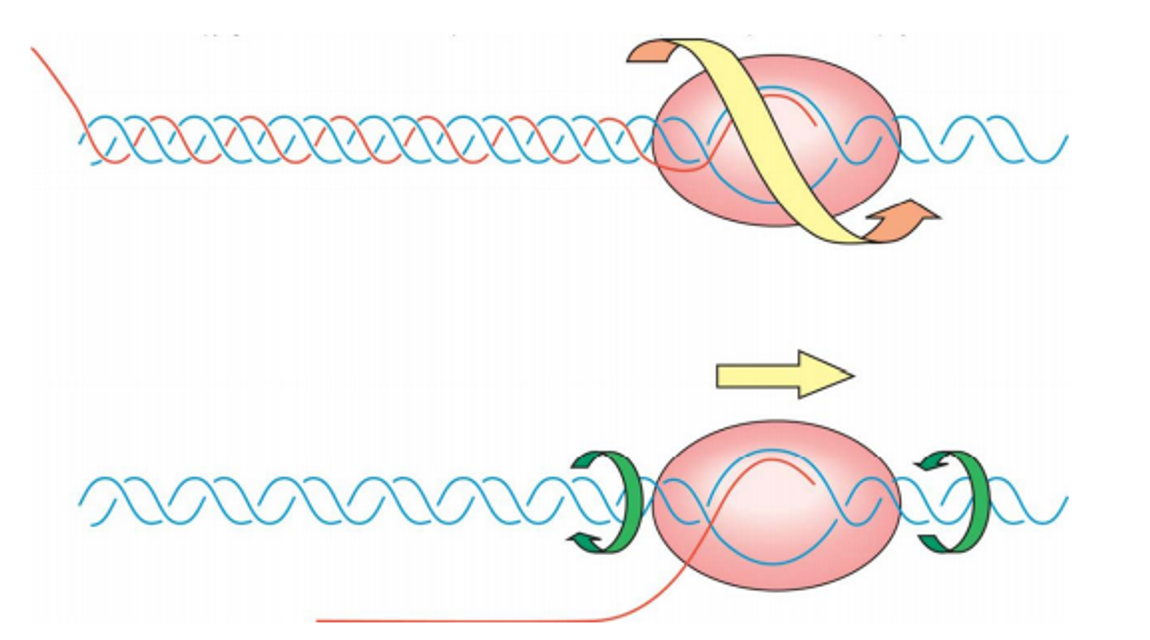
그런데 위 그림처럼 elongation 과정에서 DNA 가닥이 계속해서 일시적으로 풀리게 되므로 남아있는 upstream의 DNA 가닥들은 잘못하면 RNA와 꼬여 매우 이상한 구조를 형성할 수도 있음. 이를 방지해주는 역할을 하는 것이 topoisomerase이며, topoisomerase의 활성에 의해 위 그림 아래와 같이 과꼬임이 방지됨.
다음으로 RNA polymerase는 elongation 과정에서 가다 멈추고를 반복하거나, 심지어 뒤로 가기도 함. 이 때 가다 멈춰주는 것은 원핵생물의 경우 전사와 번역이 동시에 일어나므로 새로 생긴 RNA 가닥에 ribosome이 계속 붙어서 번역이 이루어질 수 있도록 하기 위해서이고, 뒤로 가는 것은 혹시 잘못 합성된 가닥이 있으면 이 가닥을 고쳐주는 proofreading(교정)을 수행하기 위해서임.
다음 포스트에서는 원핵생물에서의 전사 종결(transcription termination) 과정에 대해 알아보자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 7.1 : lac operon(젖당 오페론) - 1 (0) | 2023.08.30 |
|---|---|
| [분자생물학] 6.4 : 원핵생물의 transcription termination(전사 종결) (0) | 2023.08.30 |
| [분자생물학] 6.2 : 원핵생물의 transcription initiation(전사 개시) - 5 (0) | 2023.08.30 |
| [분자생물학] 6.2 : 원핵생물의 transcription initiation(전사 개시) - 4 (0) | 2023.08.30 |
| [분자생물학] 6.2 : 원핵생물의 transcription initiation(전사 개시) - 3 (0) | 2023.08.30 |