이번 포스트에서는 분자생물학의 핵심 실험 방법 중 하나인 PCR에 대해 알아보도록 하자.
PCR은 DNA의 특정 부분을 증폭하거나, cloning 이후 DNA 조각의 양을 정량하는 데 많이 사용됨. 이 기술은 Kary Mullis가 1980년대에 처음 개발하였으며, 그는 이 업적으로 이후에 노벨상을 수상함.
처음 PCR이 개발되었을 때는 온도를 변화시키는 매 단마다 계속 DNA polymerase를 넣어줬었음. 그러나 열수구에 사는 고세균으로부터 heat-stable polymerase를 발견함에 따라 이를 이용해서 PCR을 완전히 자동화하는 데 성공함.
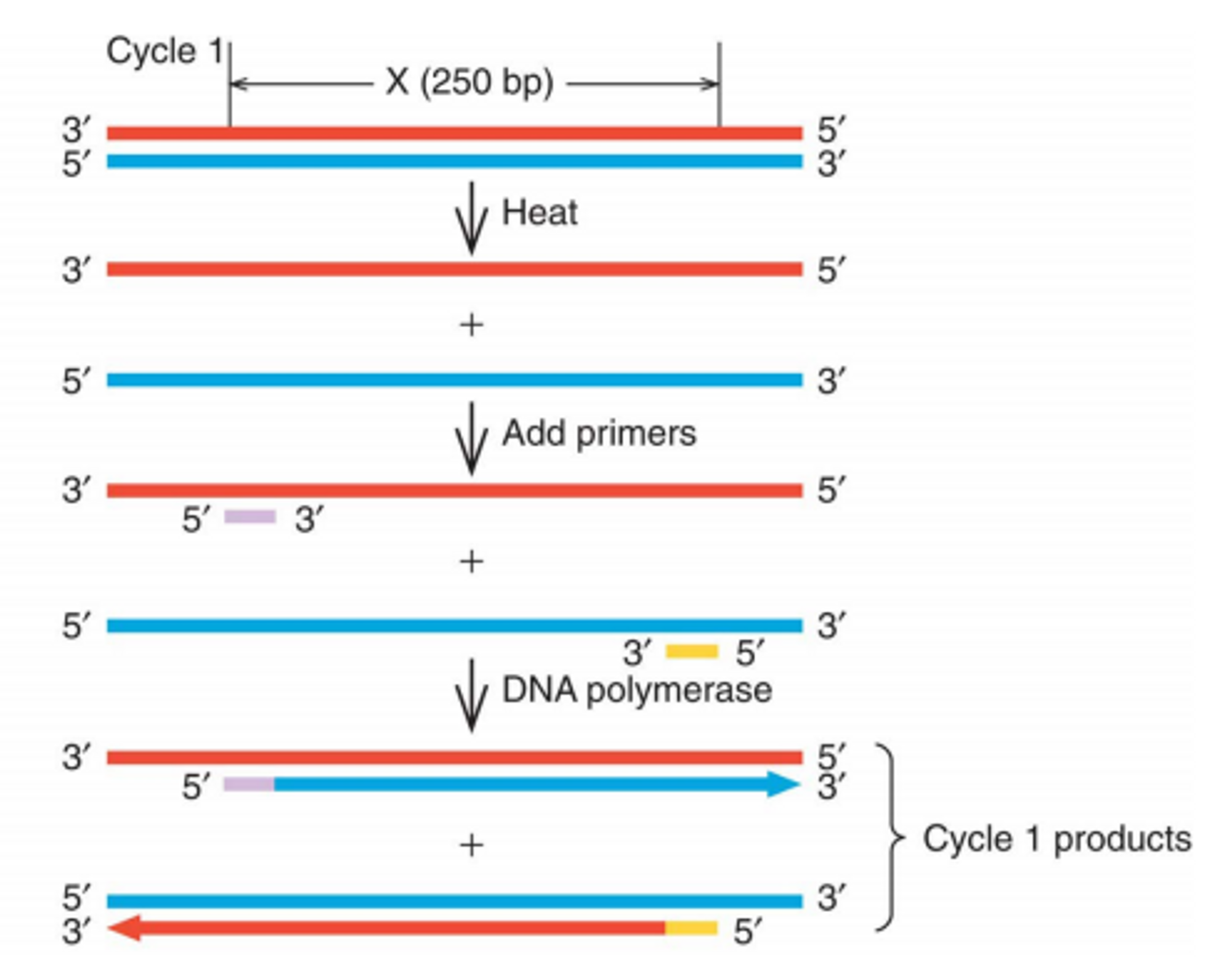
위 그림과 같이 98도의 고온(DNA 이중가닥 분리), 65도의 저온(primer의 결합), 72도의 중온(DNA polymerase에 의한 합성)을 거치게 되면 한 cycle의 PCR이 이루어지게 됨. 실제로는 대략 15~40 cycle정도가 일반적으로 수행됨.
한편, 하나의 mRNA에서부터 cDNA clone을 만들어낼 때에는 reverse trancriptase PCR(RT-PCR)이 사용됨. RT-PCR과 standard PCR의 차이는 크게 아래와 같음.
1) RT-PCR은 mRNA에서부터 시작하지만, PCR은 dsDNA(이중가닥 DNA)에서부터 시작함.
2) RT-PCR은 mRNA를 DNA로 변하는 것으로부터 반응이 시작됨.
그렇지만 일단 위에서 나열된 것과 같은 과정 이외에 standard PCR 과정과 동일한 메커니즘으로 cDNA가 증폭됨.

위 그림과 같이 RT-PCR이 진행됨. 이 때 주목해야 할 것은 처음에 넣어주는 primer에 mRNA와 상보적으로 결합하는 빨간 부분도 있는 반면 비상보적인 노란 부분도 있다는 점임. 이 때 노란 부분은 후에 제한효소를 이용해 잘라주어 sticky end를 만들어주고 cloning을 시행하기 위해서 같이 넣어주는 것임. (초록색 부분도 마찬가지. 노랑 초록이 서로 다른 제한효소에 의해 절단되므로 directional cloning이 가능해짐) 나머지 과정은 지금까지 설명했던 과정과 대동소이하므로 그림을 보면 모두 납득할 수 있을 것임.
그렇다면 우리가 가진 mRNA의 양을 정량적으로 비교하기 위해서는 어떤 방법을 사용해야 할까. 일반적으로 처음 사용한 mRNA 양이 많으면 결과적으로 증폭된 cDNA의 양도 많을 것이므로 PCR 후 gel electrophoresis(gel 전기영동)를 수행한 뒤 band의 두께를 비교하여 mRNA의 양을 비교할 수도 있기는 함. 그러나 이는 정확하지 않고, 보통 band의 유무로 mRNA의 유무를 비교하는 정도의 분석에 그치는 경우가 많음.
이럴 때 사용할 수 있는 방법이 바로 real-time PCR임.

위 그림과 같이 우리가 증폭하고자 하는 서열의 가운데에 붙는 reporter probe를 넣어줌. 이 때 reporter probe에는 F(fluorescent)와 Q(quenching)가 존재함. 이 때 F와 Q가 근처에 있으므로 F는 Q에 의해 발광을 하지 못함. 그러나 중합반응이 일어나게 되면 reporter probe도 분해되고, 그 결과 F가 Q로부터 자유로워지게 되어 형광이 방출됨. 이 형광 방출량을 PCR 전체 과정에 걸쳐서 분석하게 되면 증가하는 DNA의 양을 정량적으로 분석할 수 있음.
다음 포스트에서는 실제로 단백질의 형태로 특정 유전자를 발현시키고자 할 때 사용할 수 있는 전략들에 대해 알아보도록 하자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 5.1 : DNA 전기영동, SDS-PAGE, chromatography(크로마토크래피) (0) | 2023.06.24 |
|---|---|
| [분자생물학] 4.3 : expression vector (발현 벡터) - baculovirus, Ti plasmid (1) | 2023.06.24 |
| [분자생물학] 4.1 : cloning (클로닝) - genomic library, cDNA cloning, RACE (0) | 2023.06.24 |
| [분자생물학] 4.1 : cloning (클로닝) - vector (벡터) (3) | 2023.06.24 |
| [분자생물학] 4.1 : cloning (클로닝) - restriction enzyme (제한효소) (0) | 2023.06.24 |