이번 포스트에서는 genomic library에 대해 알아보고, 뒤이어 cDNA cloning에 대해 알아보도록 하자.
특정 species의 genome 전체를 포함하는 것이 바로 genomic library임. 사람의 경우 유전체 전체의 크기가 3 X 10^9 bp정도이므로, 한 phage 당 20kbp를 담을 수 있다는 가정 하에 대략 1.5 X 10^5개의 clone을 만들면 genomic library를 만들 수 있음. 일단 이렇게 genomic library를 만들어놓으면, 우리가 관심 있는 gene이 어디 있는지를 search하는데 활용할 수 있음. (찾은 이후에는 그 gene을 이용한 연구를 수행할 것임)
그렇다면 우리가 원하는 gene이 어디 있는지를 어떻게 찾을 수 있을까.
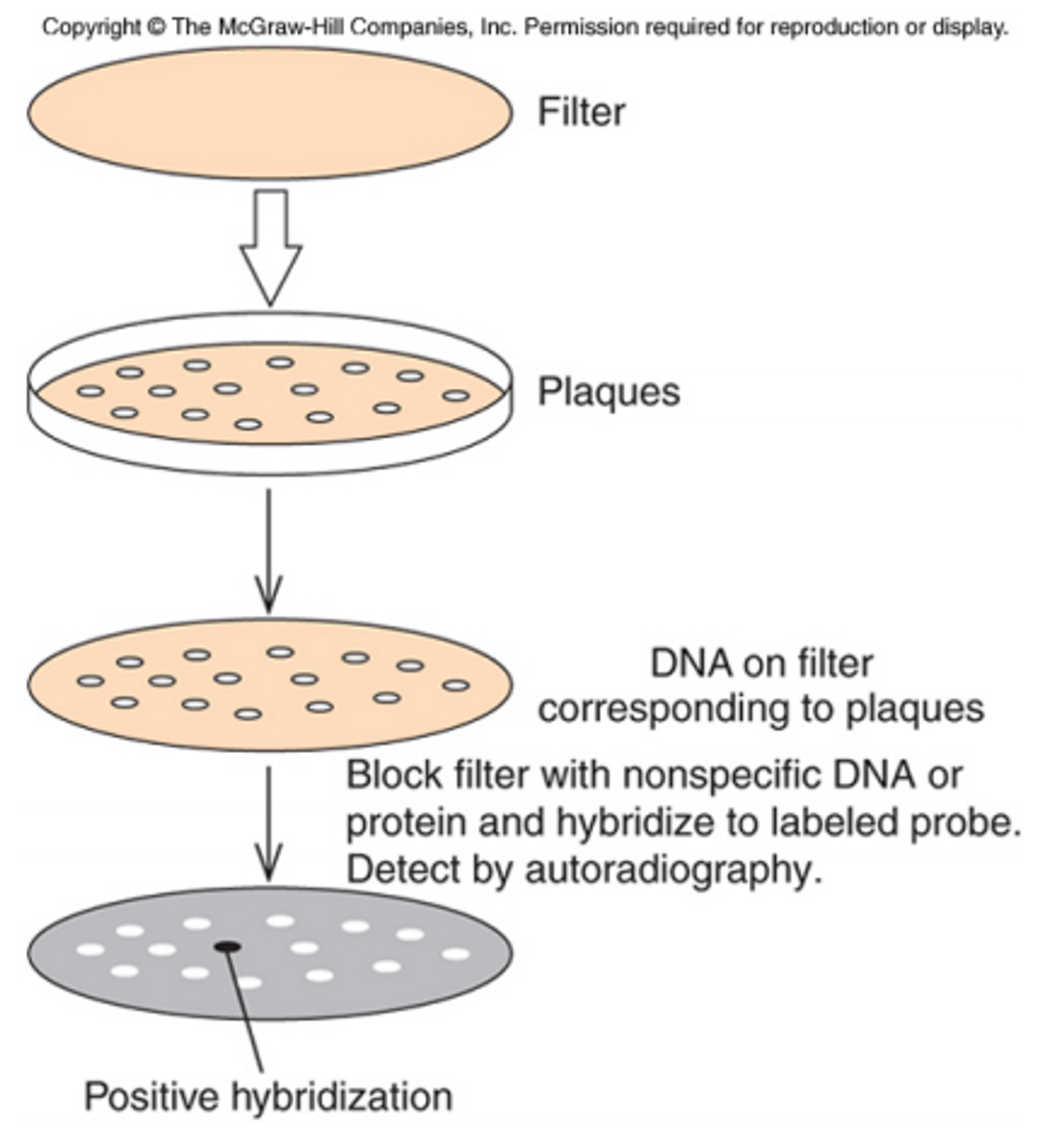
위 그림에서 나타나 있는 plaque hybridization을 통해 이것이 가능함.
genomic library를 구한 후 위 그림의 plate에 키우게 됨. 그러면 plaque가 생김. (당연히 이 과정은 엄청나게 큰 plate에서의 배양을 통해 이루어져야 할 것임) 그러면 이후 이 plaque를 종이필터로 옮기게 됨. 이후 내가 원하는 gene이 plaque 중 어디에 있을지를 찾기 위해 probe를 사용함. (probe는 다양한 형태로 사용할 수 있는데, 가장 흔한 probe는 우리가 관심 있는 gene의 특정 서열을 알고 있을 때 그 특정 서열을 labeling한 것임) 이 probe를 종이 필터 위에 뿌려주게 되면 autoradiography를 통해 우리가 관심을 가지고 있는 gene이 어느 plaque에 있는지를 확인할 수 있음.
probe를 이용해서 특정 clone을 identify하는 방법에는 크게 두 가지가 있음. 하나는 polynucleotide(oligonucleotide) 형태의 probe를 사용하는 것이고, 나머지 하나는 antibody를 사용하는 것임(이 경우 우리가 관심 있게 보고 있는 gene의 유전자 산물을 알고 있을 때 그에 상보적으로 결합하는 antibody를 제작해서 probe로 사용하는 방식임).
그러면 polynucleotide probe에 대해 조금 더 알아보자. 가장 많이 쓰이는 방식은 앞서 살펴본 것처럼 우리가 관심 있는 gene의 서열 일부를 알 때 그 서열과 결합할 수 있는 labeled probe를 만드는 것임. 이 때 주의해야 할 점은, 온도, solvent 농도, 염 농도와 같은 것들을 적절히 조절해서 너무 잘 붙게도 하지 말고, 너무 잘 안 붙게도 하지 말아야 한다는 것임. 딱 적당한 정도로 붙어야 우리가 원하는 gene과 probe 간의 specific한 결합만 일어날 것이기 때문.
이 밖에 protein을 이루는 amino acid 서열의 일부를 알고 있을 때, 이를 역산해서 nucleotide sequence를 유추해 낸 후 이를 바탕으로 probe를 만들어내는, protein-based polynucleotide probe 제작 방식도 있음. 그런데, 이 때 대부분의 amino acid를 가리키는 triplet들은 하나 이상이며, 어떤 경우에는 6개의 triplet code가 결국 동일한 하나의 amino acid를 indicate하는 경우도 존재함. (degenerate) 이 때문에 역산을 통해 예상하는 nucleotide probe가 여러 종류가 될 수밖에 없음. (가능한 모든 nucleotide를 만든 후 이들을 다 넣어주어야 함) 그러다 보니 최소한의 probe 개수만으로 특정 gene을 감지하기 위해 서열을 잘 선택하는 것이 중요함. 이에 대한 간단한 예시를 살펴보자.

위 그림과 같이 10개의 amino acid 서열을 안다고 해 보자. 오른쪽에는 각 amino acid를 만들게끔 지시하는 codon들의 종류가 나타나 있음. 이 때 저 10개의 서열 중 5개를 골라서 가장 적은 degeneracy(두 개 이상의 코돈이 하나의 아미노산을 지정하는 경우)를 가지는 17-mer probe를 만들고자 한다면 어떤 서열을 골라야 할까.
이에 대한 해결을 위해서는 우선 기본적으로 amino acid의 한자 약어 규칙에 대해 숙지하고 있어야 함.
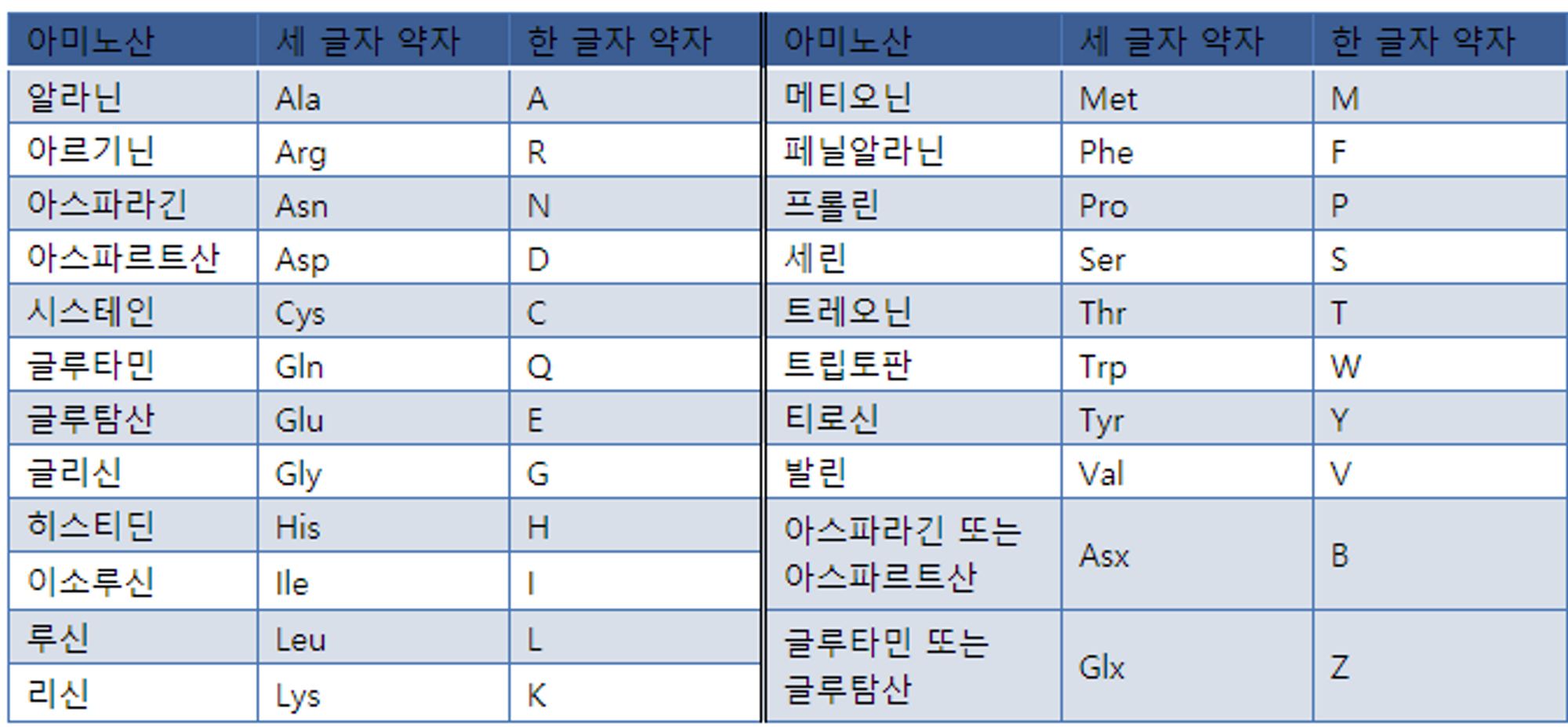
이를 참고해서 이제 각 amino acid의 degeneracy를 계산해보자. 그러면 R, L, M, E, W, I, C, P, M, L이 각각 6, 6, 1, 2, 1, 3, 2, 4, 1, 6의 degeneracy를 가진다는 것을 알 수 있음. 그러므로 M-E-W-I-C를 골랐을 때의 degeneracy가 12로 가장 적음. 한편 이 때 17-mer를 만들어야 하므로, 이후 이어지는 P에 해당하는 codon의 앞의 두 서열을 더 추가해주면 됨. 이 때 P에 matching되는 codon의 앞 두 서열은 CC로 동일하므로 이 경우에도 여전히 degeneracy는 12임. (만약 앞 두 서열이 일정하지 않은 경우에는 가능성 수 만큼을 곱해서 degeneracy를 구해주어야 함)
다음으로 cDNA cloning에 대해 알아보도록 하자.
cDNA는 complementary DNA, 혹은 copy DNA의 약자로, RNA를 다시 DNA로 역전사시킨 후에 이를 cloning하는 방식을 cDNA cloning이라 불러줌. cDNA library는 동일 개체라 하더라도 발생과정마다 다 다르며, 동일 개체에서 장기마다도 다 다름. 그렇다면 cDNA library는 어떻게 제작할 수 있을까.
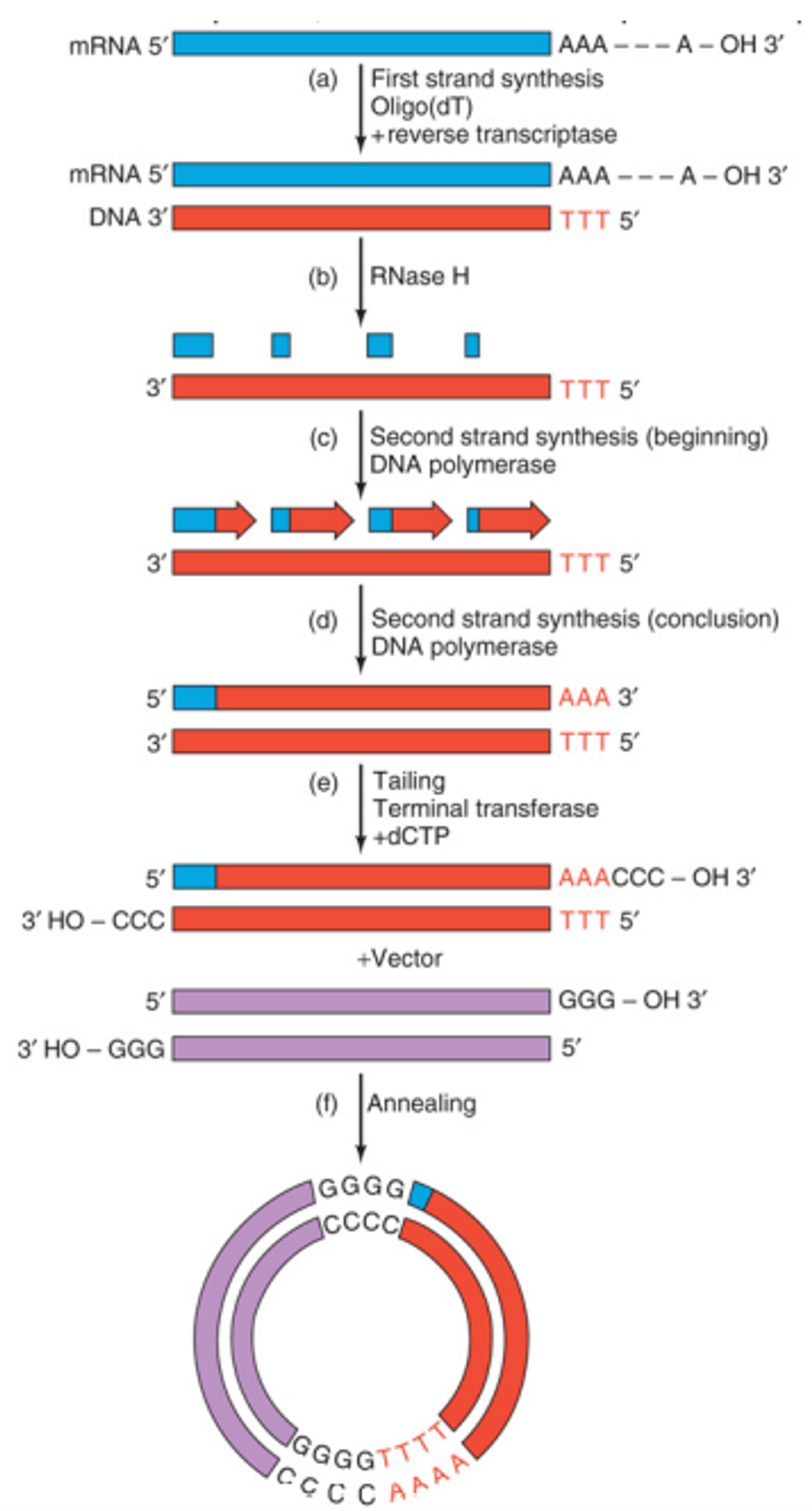
위 그림에 나타난 과정에 대해 자세히 살펴보자. 우선 우리가 원하는 mRNA를 추출함. (이 때 추출시에는 poly A tail을 targeting하는 것이 가장 일반적임) 이후 oligo dT primer를 넣어준 후 reverse transcriptase(역전사 효소)를 넣어주면 mRNA의 poly A tail 부분부터 5' end까지 reverse transcription(역전사)됨.
그러고 나서 RNase H라는 효소를 넣어주어서 원형으로 사용된 mRNA를 제거해줌. 이 때 완벽히 제거해주지는 않고 적당히 조각들이 남아있을 정도로만 RNase H를 처리해줘서 남은 mRNA 가닥들이 primer 역할을 할 수 있도록 해줌.
이후 DNA polymerase를 넣어줌. 이 때 넣어주는 DNA polymerase는 5'→3' exonuclease 활성을 가짐. 따라서 일단 primer부터 쭉 합성해나가다가 다음 조각의 primer를 만나게 되면 이 녀석들을 분해해가면서 새로운 가닥을 합성해나감. 이후 terminal transferase를 처리해서 재조합에 필요한 sticky end를 첨가해주면(이 경우에는 dCTP를 넣어주면) vector와의 annealing에 의해 cDNA cloning을 완료할 수 있음.
이제 이 과정에서 사용된 여러 효소들에 대해 조금 더 자세히 알아보자.
우선 reverse transcriptase(RT)는 RNA-dependent DNA polymerase임. 이 때 RT는 primer 없이는 DNA synthesis를 시작하지 못하며 그렇기에 일반적으로 oligo dT를 primer로 많이 사용함.
다음으로 Ribonuclease H(RNase H)는 RNA-DNA hybrid에서 RNA strand를 제거할 수 있음. 이 때 앞서서도 설명했듯 RNA를 완벽히 제거하지는 않게금 적당량을 조절해 넣어주는데, 남은 RNA 조각들은 nick translation에서의 primer로 작용함. 참고로 nick translation은 nick(홈)이 존재할 때의 DNA synthesis과정이며, 그림으로 나타내면 아래와 같음.

이 때 E.coli가 사용하는 DNA polymerase 1이 사용되며 이 녀석은 앞서 말한 것처럼 5'→3' exonuclease activity가 있으므로 nick의 뒤에 있는 부분은 새로 합성되고 nick의 앞에 있는 부분은 분해됨. 그 결과 위 그림과 같이 nick이 5'→3'으로 이동하는 듯한 양상으로 관찰됨.
다음으로, 생성된 cDNA가 full-length가 아닌 경우, 비어있는 부분의 조각을 채워넣는 방법에 해당하는 RACE(rapid amplification of cDNA ends)에 대해 알아보자.
실제로 missing piece(비어있는 부분)가 5' end 쪽에 위치하는 경우(이 경우가 제일 흔함) 이를 5' RACE, 3' end 쪽에 위치하는 경우 이를 3' RACE를 사용할 수 있음.
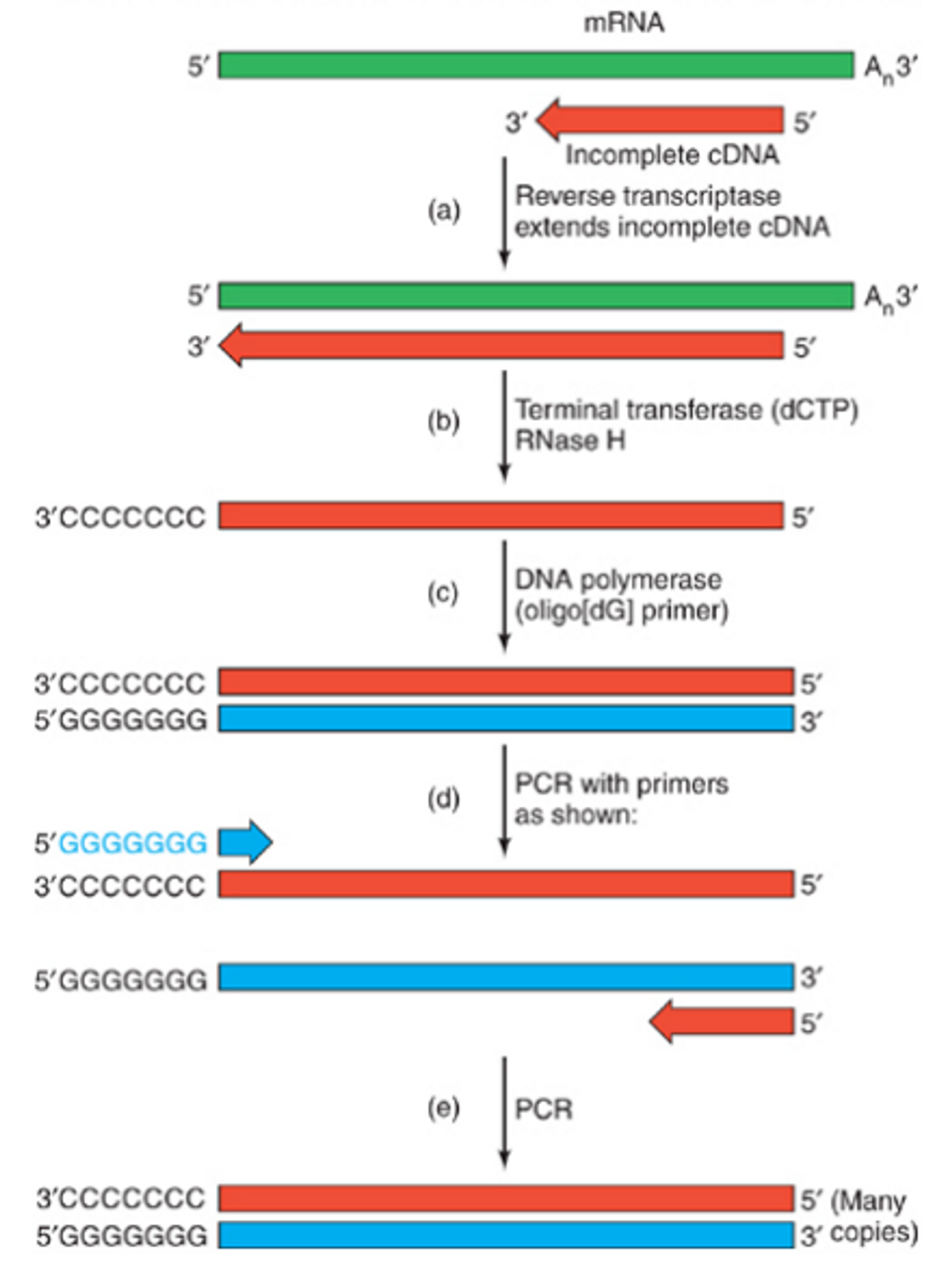
위 그림은 그 중에서도 5' RACE의 과정을 나타내주고 있음. (3' RACE의 방법도 거의 동일함)
위 그림의 맨 위쪽과 같이 mRNA의 5' 부분에 대해 missing piece가 존재하는 경우, 이 서열에 reverse transcriptase를 처리해주게 되면 기존에 존재하던 incomplete cDNA를 primer로 하여 missing piece가 이어서 합성됨. 이후 RNase H를 처리해서 주형으로 사용된 mRNA는 제거해주고, terminal transferase와 dCTP를 처리해서 3' end에 poly C를 달아줌. 한편 DNA polymerase와 함께 poly G primer를 넣어주게 되면 위 그림의 (c) 과정에서 나타나 있는 것처럼 상보적인 DNA 서열이 합성됨.
이후 우리가 합성하고자 하는 부분에 대한 primer와 함께 PCR을 진행해주면 우리가 원하는 cDNA part를 많이 얻어낼 수 있음.
다음 포스트에서는 cloning에 이어서, 분자생물학의 핵심 실험기법인 PCR(polymerase chain reaction)에 대해 알아보도록 하자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 4.3 : expression vector (발현 벡터) - baculovirus, Ti plasmid (1) | 2023.06.24 |
|---|---|
| [분자생물학] 4.2 : PCR, RT-PCR, real-time PCR (0) | 2023.06.24 |
| [분자생물학] 4.1 : cloning (클로닝) - vector (벡터) (3) | 2023.06.24 |
| [분자생물학] 4.1 : cloning (클로닝) - restriction enzyme (제한효소) (0) | 2023.06.24 |
| [분자생물학] 3 : 유전자의 기능과 특징 - 저장, 전사, 번역, 복제, 돌연변이 (0) | 2023.06.24 |