지난 포스트에서 유도한 식에 이어서, 이제 다음으로 우리가 직접 측정하기 어려운 [E], [ES}와 같은 term들을 어떻게 제거해서 Km을 계산하기 쉽게 묘사할 수 있을지에 대해 생각해보자.
일단 우리는 처음에 넣어준 enzyme의 총량, 즉 [ET]의 값은 알고 있음. 그리고 [ET]=[E]+[ES]라는 관계식이 성립한다는 것도 알고 있음. 이를 이용해 우선 측정하기 곤란한 term 중 하나인 [E] term을 Km 식에서 없애보자.
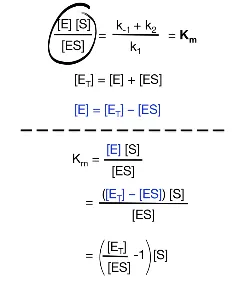
그러면 Km 식을 결과적으로 위 그림 맨 아래에 나와있는 것과 같이 다시 쓸 수 있음.
그 다음으로 이제 [ES] term을 제거해보자. 이를 위해 기질의 농도가 매우 높은 극단적인 상황을 생각해보자. 이 상황이라면 앞서 살펴봤던 것처럼 초기속도는 Vmax에 도달할 것이고, 이 시점에서의 [ES]는 [ET]와 똑같아질 것임. (E가 기질에 의해 full saturation되었을 것이므로) 따라서 결과적으로 Vmax를 아래와 같이 표현할 수 있음.
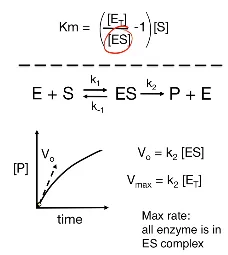
.
이 때 위 그림 맨 아래에 나타나 있는 V0, Vmax 식을 자세히 보면 k2가 공통적이라는 것을 알 수 있음. 한편 Vmax/V0를 계산해보면 k2가 약분되면서 결과적으로 ET/ES가 됨. 따라서 결과적으로 Km 식에서 ET/ES를 Vmax/V0로 대체해서 아래와 같이 다시 쓸 수 있음.
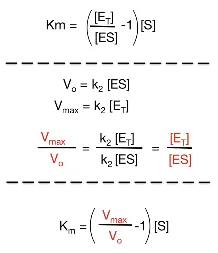
한편 이 식을 다시금 V0에 대해 정리해 주면 아래와 같이 V0를 [S]에 대한 함수로 정리할 수 있음.

이렇게 얻어진 equation을 Michaelis Menten equation이라고 부름.

결과적으로 이런 유도 과정을 통해서 위와 같은 Michaelis-Menten equation을 얻을 수 있었음. 이 때의 Km이 enzyme의 특성을 나타내주는 quantitative parameter임. 그리고 kcat은 turnover number이며 1초당 product로 얼마나 많이 변화되는지 정도를 의미함. 이 때 kcat은 아래와 같이 조합되어 Vmax를 정의할 수 있음.
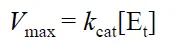
한편 Km은 구체적으로 Michaelis constant로 불리며 이 녀석은 equilibrium constant와 꽤나 비슷한 개념이라 생각해봐도 무방함. 이 constant는 아래와 같이 정의됨.
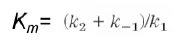
Km은 enzyme과 substrate간의 affinity 정보를 approximate해줄 수 있는 값임. 이 때 만약 k2 값이 굉장히 작으면(일반적으로 ES→E+P가 어려운 반응) 이 경우 k2는 무시 가능함. 그럴 시의 Km은 Kd가 되어버림. 이렇게 되면 말 그대로 Km은 affinity 그 자체를 의미하게 됨.
그리고 steady state에서 maximum velocity Vmax를 생각할 수 있음
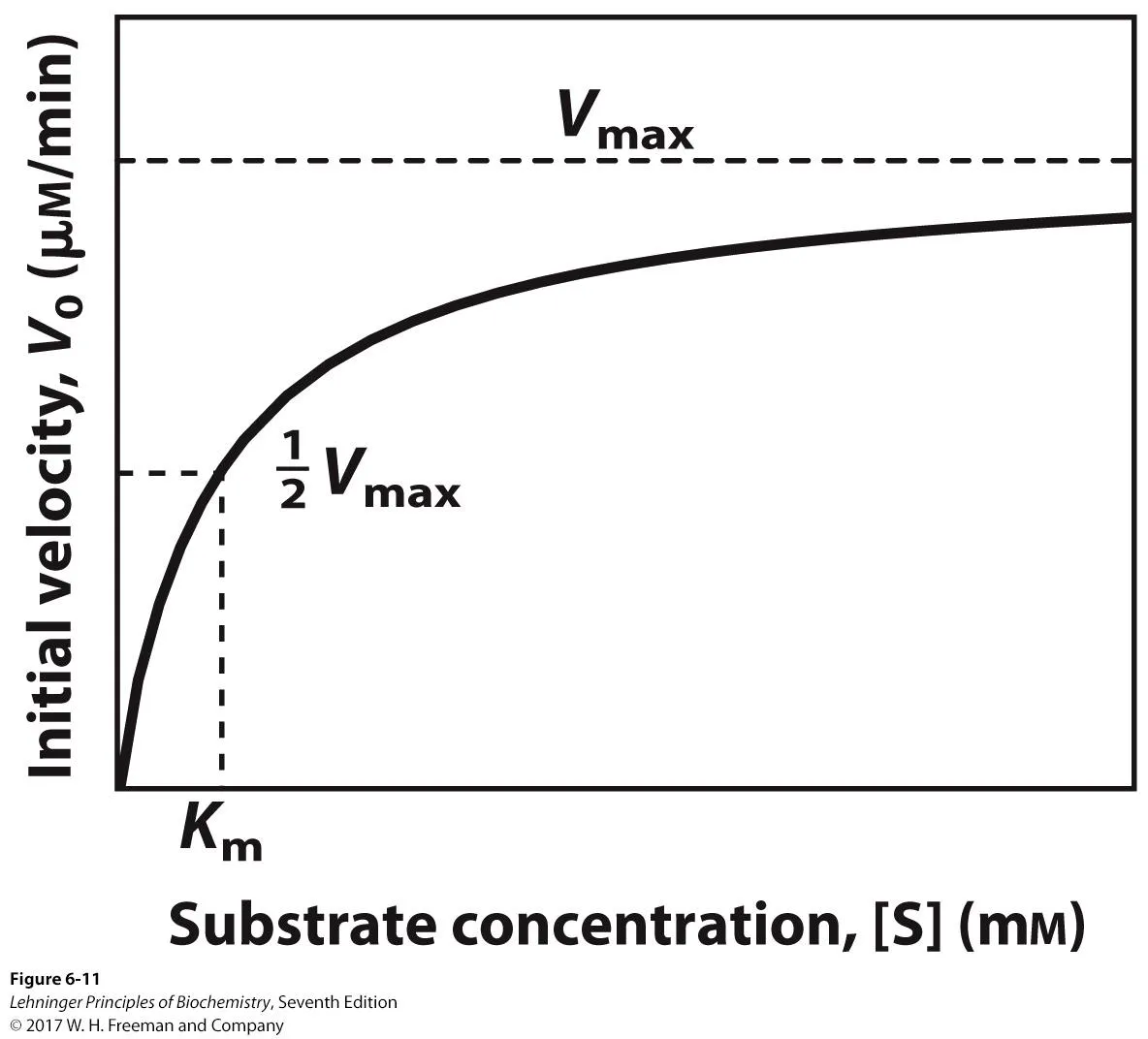
위 graph는 substrate의 농도를 서서히 올려줬을 때 initial velocity가 어떻게 달라지는지를 보여주고 있음. 이 때 결국 [S]가 엄청나게 증가하면 initial velocity는 Vmax에 수렴하게 됨. 그렇기에 만약 initial velocity가 Vmax인 상황이라면 이 때는 사실상 모든 효소가 다 ES complex를 형성하고 있을 것이므로 결국 Vmax=kcat x [Et]가 되는 것임.
이 밖에 specificity constant라는 것도 존재함. 이 constant는 kcat / Km임. 이 때 kcat은 얼마나 특정 reaction이 빨라지는지에 대한 정보이고 Km은 affinity에 대한 정보를 줌. 따라서 이 둘을 다 고려해서 얼마나 catalytic efficiency가 좋은지 정도를 표현할 수 있는 상수가 바로 specificity constant임.
한편 substrate의 양이 매우 적을때는 아래와 같은 approximation이 가능함.
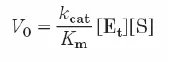
이 공식하에서의 rate constant가 바로 specificity constant임.
그렇다면 방금 전에 봤던 substrate-initial velocity graph는 어떻게 얻을 수 있을까.
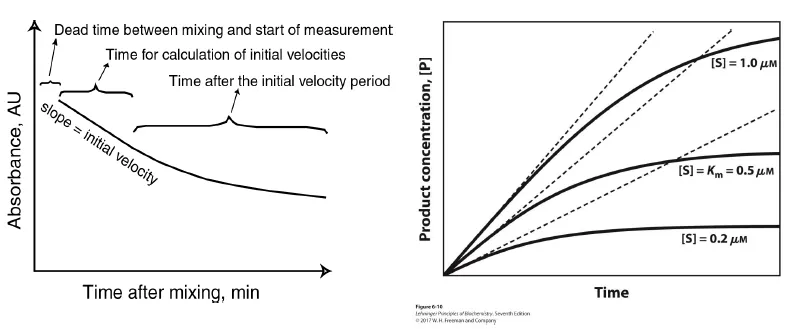
방법은 간단한데, enzyme과 substrate를 넣고 absorbance를 측정하면 됨. 이 때 줄어드는 substrate의 양을 측정할 수도 있고 늘어나는 product의 양을 측정할수도 있음.
이 때 위 그림 오른쪽 graph와 같이 S의 양을 계속 달리해가면서 실험을 수행함. 그랬을 때 얻어지는 graph의 접선의 기울기는 점차 감소하는 형태일텐데(시간이 지날수록 S의 양이 줄어들게 되므로) 그러므로 맨 처음의 초기속도를 구하기 위해 time이 0인 지점에서의 graph에 접선을 긋게 됨. 이 때의 직선 기울기가 바로 특정 S에서의 initial velocity임.
한편 이러한 측정은 실제로는 정확히 이루어질 수 없음. 이럴 수 밖에 없는 이유는 여러 가지가 있는데, 일단 그 중 하나가 위 graph에 나타나 있는 dead time임. 이 time은 실제로 반응이 시작되었을 때부터 측정을 할 때까지 기계가 소비하는, 낭비하는 시간을 의미함. 이 값의 경우 기술적 한계가 있기 때문에 줄일 수는 있어도 아예 극복하는 것은 불가능함.
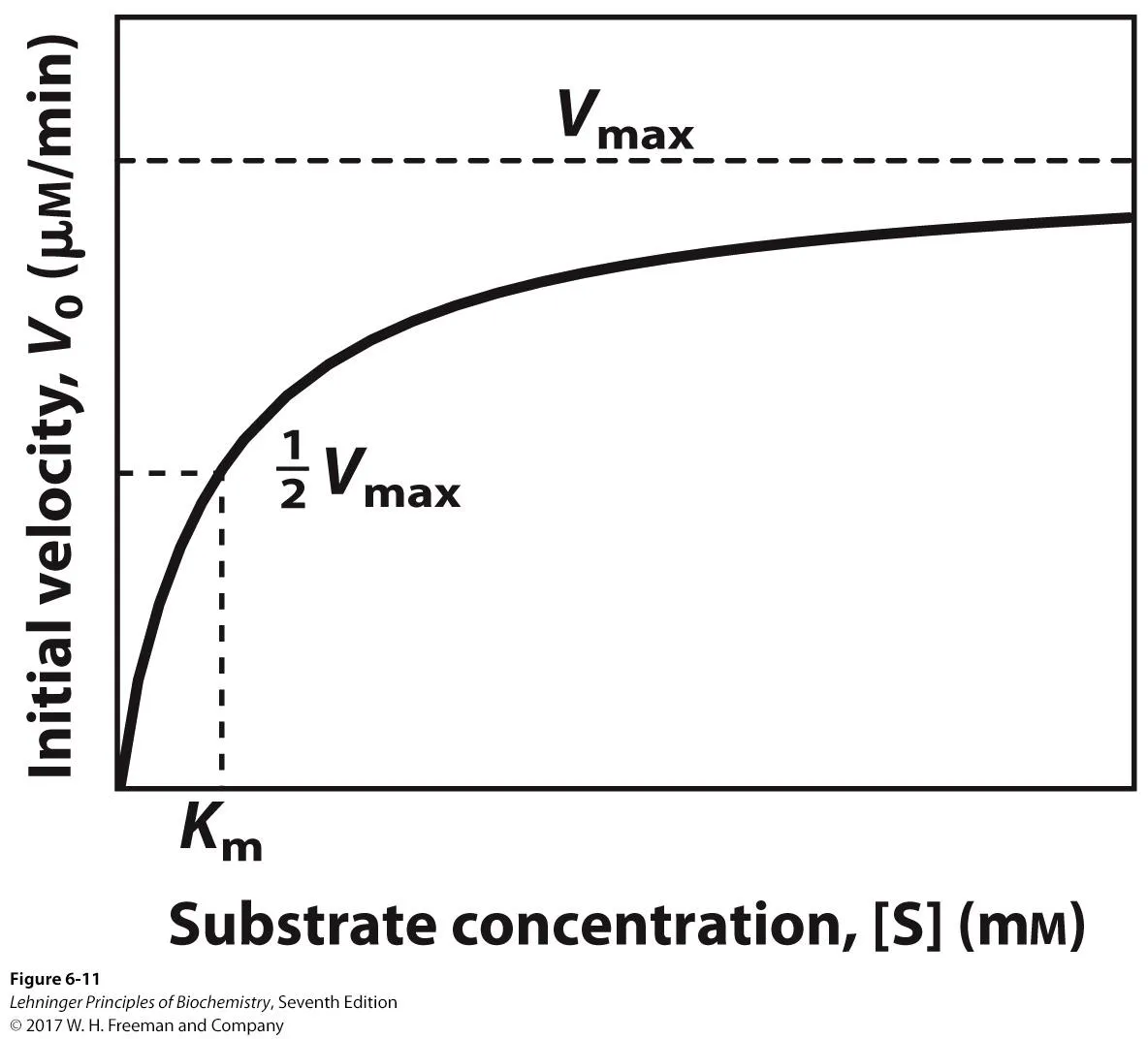
한편 위 graph에서 Km 값은 재미있는 의미를 가짐. Michaelis-Menten equation에 따르면 Km이 [S]와 같아질 때 속도가 Vmax의 절반이 된다는 것을 알 수 있음. 따라서 실제로 [S]=Km인 point에서의 initial velocity는 1/2Vmax임.
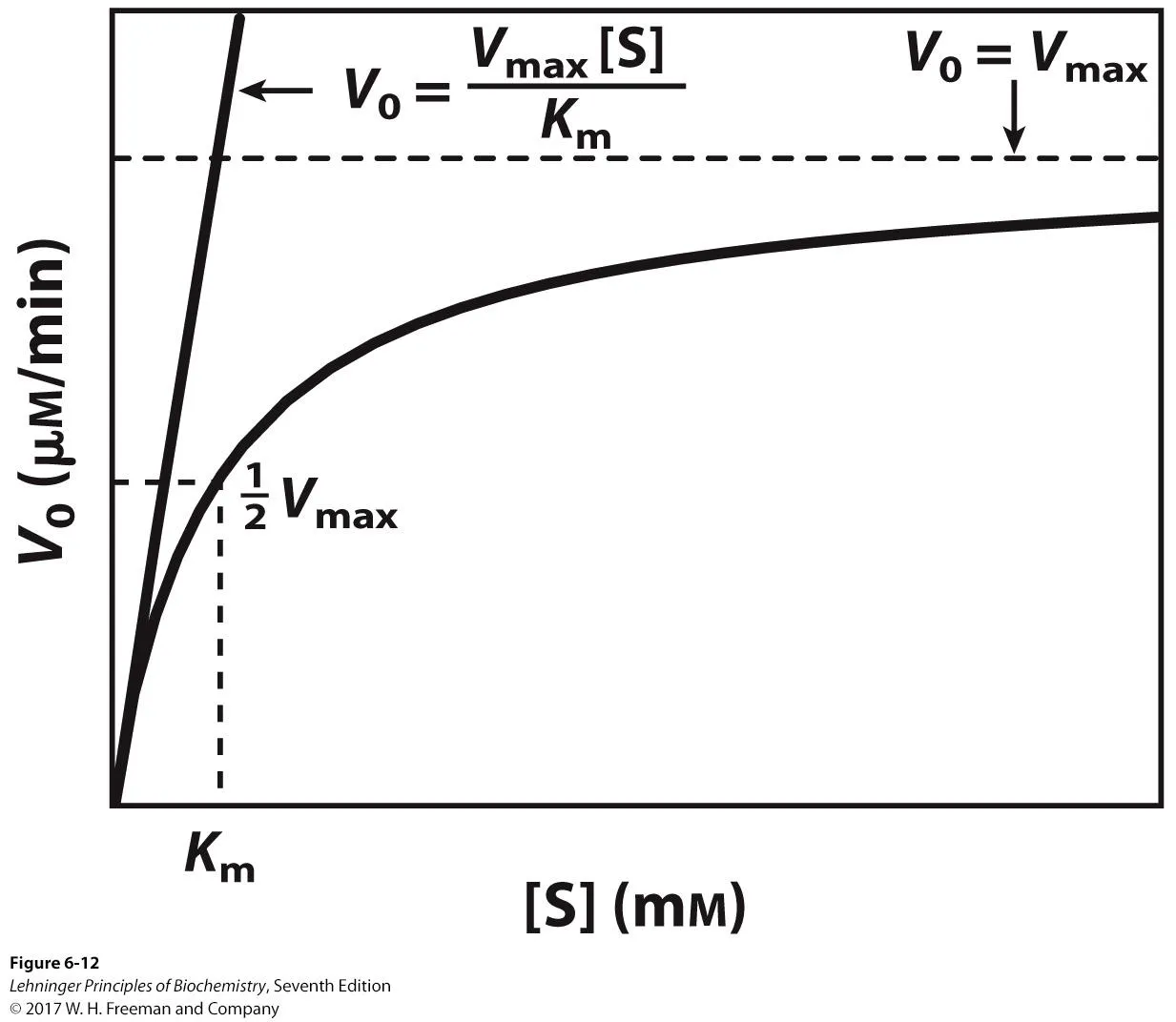
한편 위 graph에서도 나와있는 것과 같이 실제로는 [S]가 굉장히 작을 때 V0가 위 그림 위쪽에 나타나있는 식과 같아지게 됨.
그런데 이러한 graph를 통해서는 Km, Vmax를 계산해내기 어려움.
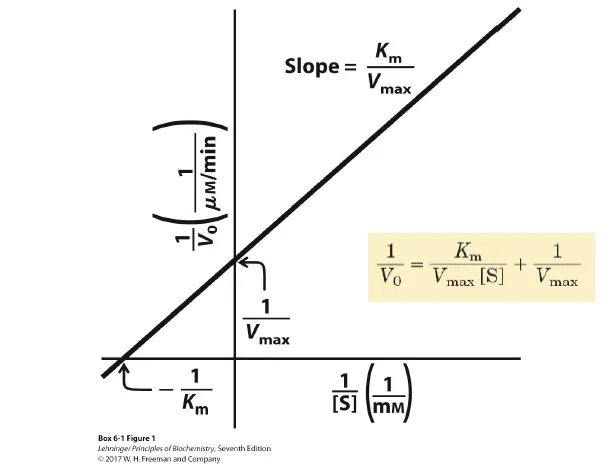
이 때 활용 가능한 것이 바로 위에 나타나 있는 Lineweaver-Burk plot(라인위버-버크 플랏)임. 이 plot의 경우 x축으로 1/[S], y축으로 1/V0으로 한 상태에서 그린 것임. 그럴 시 얻어지는 직선의 x, y절편으로부터 Km, Vmax를 쉽게 계산해낼 수 있음.
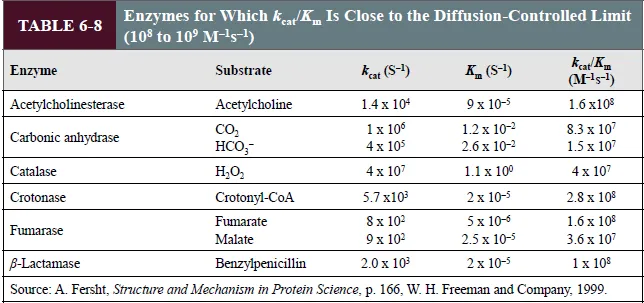
한편 위 표에는 효소의 종류에 따른 specificity constant가 나타나 있음. 이 때 이 constant가 크면 클수록 더 enzyme efficiency도 크다는 것임. 그런데 사실 enzyme, substrate가 만나서 반응하기 위해서는 diffusion을 해야 하므로 이 enzyme efficiency가 무한정 좋아질수는 없고, diffusion을 고려했을 때 최대한 커질 수 있는 specificity factor는 10^{8}-10^9M^{-1}s^{-1}임. 따라서 위 표상에서 acetylcholinesterase, Crotonase와 같은 효소는 사실상 거의 maximum에 가까운 enzyme efficiency를 내고 있다고 보아도 무방함.
다음 포스트에서는 여러 substrate가 있을때의 반응을 해석해보도록 하자.
'전공자를 위한 생물학 > 생화학' 카테고리의 다른 글
| [생화학] 5.6 : 효소 반응의 조절 (0) | 2024.11.07 |
|---|---|
| [생화학] 5.5 : 효소의 kinetics - 3 (0) | 2024.11.07 |
| [생화학] 5.5 : 효소의 kinetics - 1 (0) | 2024.11.07 |
| [생화학] 5.4 : 펩티도글리칸과 라이소자임(peptidoglycan and lysozyme) (0) | 2024.11.07 |
| [생화학] 5.3 : HIV protease, hexokinase, enolase (0) | 2024.11.07 |