이번 포스트에서는 지금까지 살펴보지 않았던 다양한 post-trasncriptional regulation 기작들에 대해 알아보자.
실제로 많은 경우 post-transcriptional control을 통해 mRNA의 stability가 조절되게 됨.

위 표를 보면 Casein mRNA의 경우 다른 mRNA보다도 특별히 prolactin이 있을 시 RNA half-life가 비약적으로 증가하는 것을 볼 수 있는데, 이것도 다 post-transcriptional control에 의해 발생하는 현상임.
post-transcriptional control과 관련해서 흥미로운 예시가 있음. 바로 transferrin receptor의 mRNA stability와 관련된 내용임. 우선 이에 대해 이해하기 전에 transferrin receptor의 역할에 대해 알아야 함.
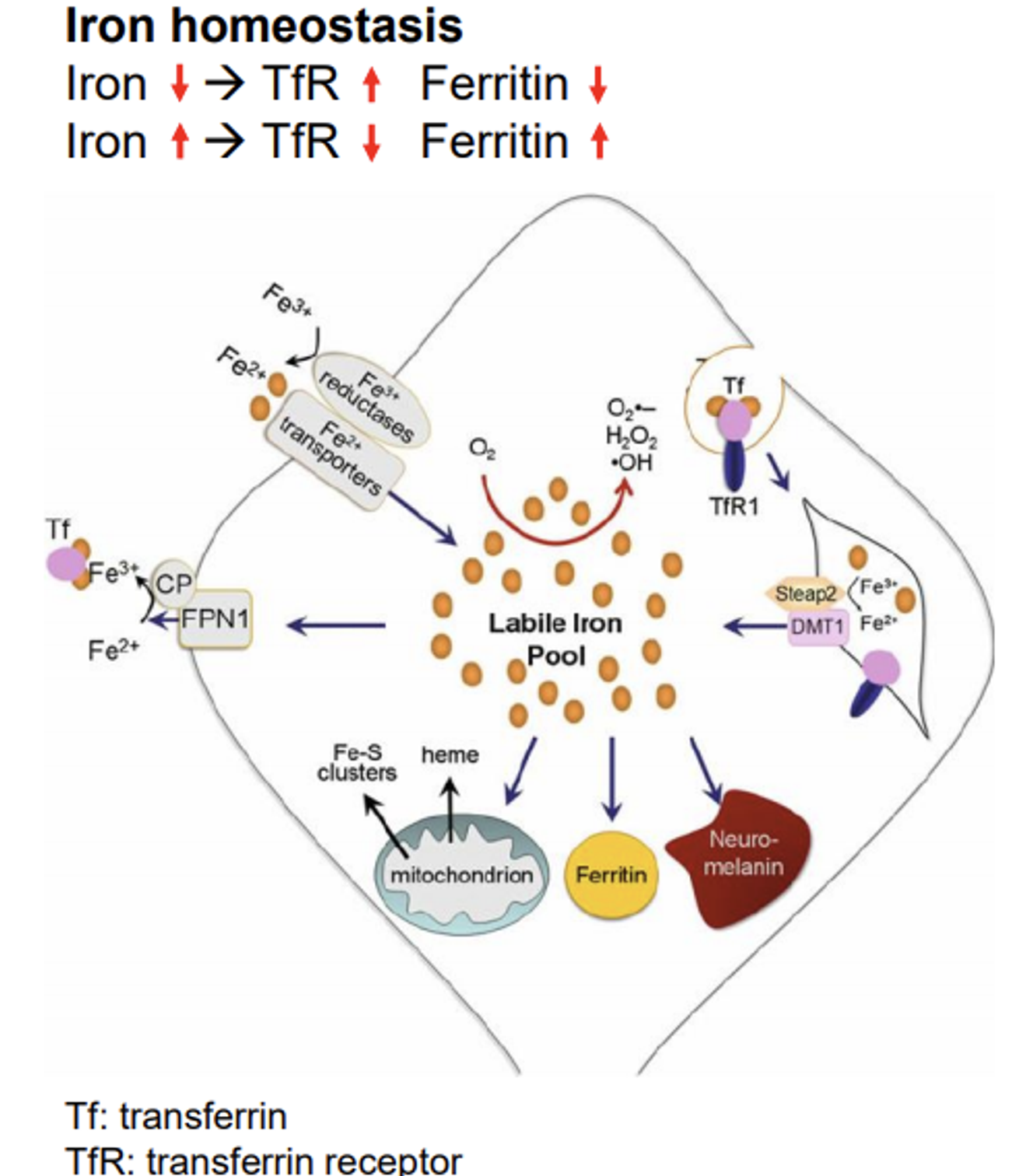
iron(철)은 세포 내에 너무 많을 시 toxic한 반면 그럼에도 불구하고 세포 내에 어느 정도의 양은 있어야 하기에 iron 농도가 정교하게 조절되어야 함. 이와 관련된 단백질이 transferrin receptor(TfR)와 ferritin임.
이 때 TfR은 iron을 세포 내로 통과시켜주는 역할을 하고, ferritin은 iron을 저장하는 역할을 함. 그렇기에 위 그림에 나와있는 것처럼 세포 내에 iron 농도가 너무 많은 경우 iron의 추가적인 출입을 막기 위해 TfR의 양은 줄어들고, 많은 iron들을 저장하기 위해 ferritin의 양은 늘어남. 한편 세포 내에 iron 농도가 너무 적은 경우에는 TfR의 양이 늘어나고 Ferritin의 양은 줄어들 것임.
그렇다면 TfR, Ferritin의 발현양이 iron 농도에 따라 어떻게 조절되는 것일까.
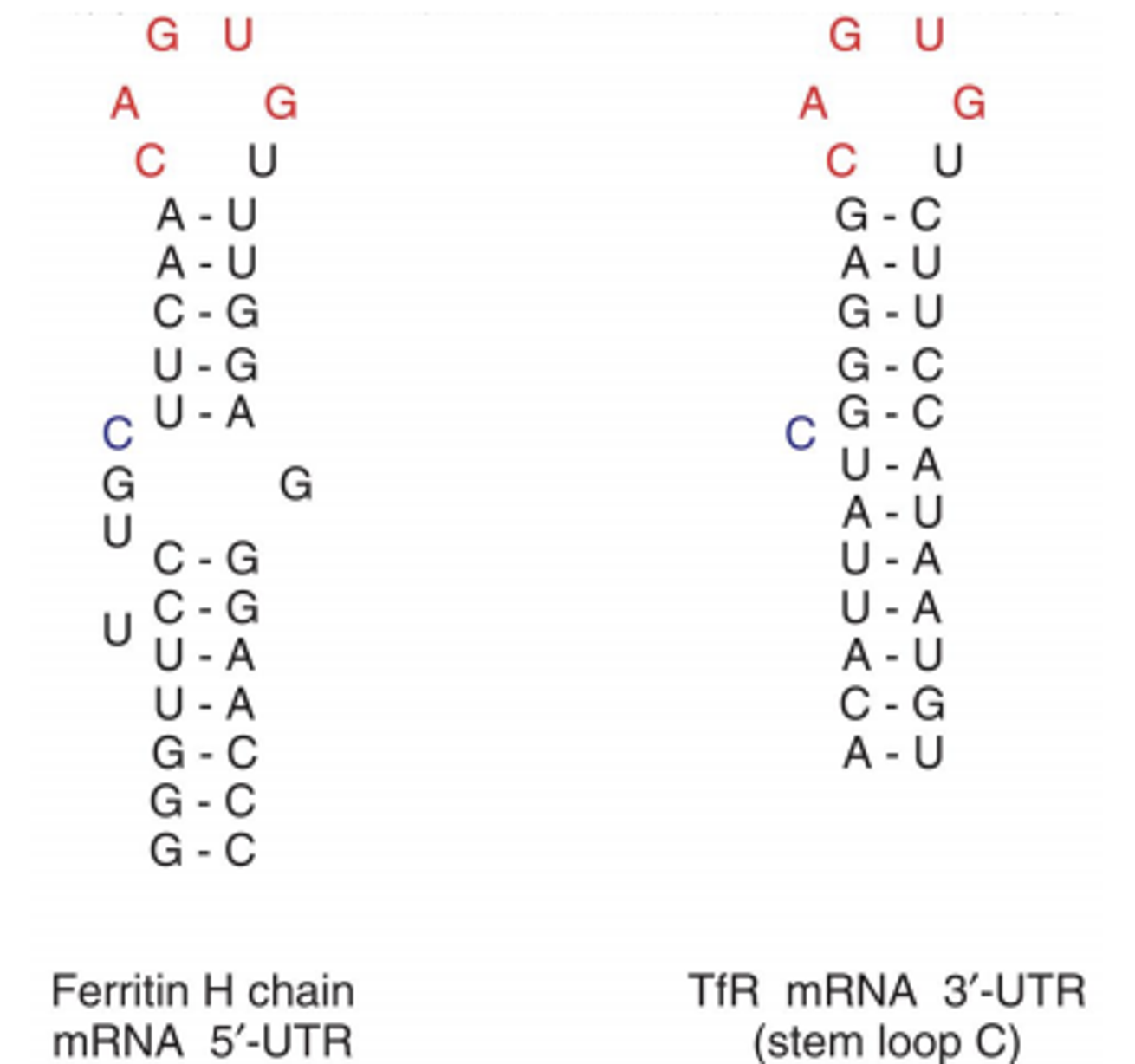
TfR, Ferritin을 encoding하고 있는 mRNA에는 위와 같은 특징적인 구조가 있음. 이 구조가 iron 농도에 따른 이들의 발현조절에 핵심적임. 우선 Ferritin의 경우 5' UTR 부분에 위 그림 왼쪽과 같은 stem loop 구조가 있음.
한편 TfR mRNA의 경우 3' UTR 부분에 위 그림 오른쪽과 같은 stem loop 구조가 있음. 이 때 Ferritin mRNA에서의 loop은 translation과 매우 깊은 관련이 있고, 이후 포스트들에서 다루어질 것임. 한편 TfR mRNA에서의 loop은 post-transcriptional control과 밀접하게 관련되어 있음. 이에 대해 조금 더 알아보자.

우선 이와 관련된 실험을 하기 위해 TfR의 3' UTR 부분을 가져옴. 이 때 전체 UTR 부분을 다 사용하기에는 너무 복잡하므로 (a)의 맨 위와 같이 별로 중요하지 않은 부분들을 다 제거해 준 상태로 실험을 수행함.
(a)의 맨 위 오른쪽에 나타난 UTR이 포함된 mRNA를 이용해 수행한 실험이 positive control임) 한편 (a)의 두번째 줄에서는 앞서 봤던 stem loop에 해당하는 B, C, D 부분을 다 제거해줘봄. 마지막으로 (a)의 세번째 줄에서는 stem loop에서 중요한 역할을 하는 것으로 알려진 nucleotide들을 바꿔봤음.
이들을 이용해 (b)와 같이 TfR biosynthesis 정도를 측정해봄. 이 때 H와 D condition을 번갈아가며 사용하였는데, H는 hemin을 의미하며 hemin이 있을 경우 hemin이 iron을 함유하고 있으므로 iron이 풍부한 condition이 됨.
반면 D는 desferrioxamine을 의미하며 desferrioxamine이 있을 때는 desferrioxamine이 iron을 킬레이팅하는 역할을 해서 결과적으로 iron이 부족한 condition이 됨.
control의 실험 결과를 먼저 보면 iron이 풍부한 H에서는 TfR이 적게 만들어진 반면 iron이 부족한 D에서는 TfR이 많이 만들어짐. (100%의 regulation) 반면 아예 stem loop을 빼 준 TRS-3나 loop의 일부 nucleotide를 바꿔준 TRS-4의 경우 H와 D에 따른 TfR 농도의 차이가 나타나지 않음.
즉, iron에 따른 response가 일어나지 않았고, 결국 생합성 조절이 일어나지 않은 것임.
그렇다면 왜 TRS-3의 경우에서는 TRS-1의 H condition보다 더 많은 TfR 발현양이 나타나는 것일까. 이는 결국 원래는 stem loop이 iron 농도가 높을 때에는 mRNA의 stability를 저해시켜주는 역할을 하기 때문임.
그렇기에 아예 loop가 없는 TRS-3에서의 TfR 발현양이 더 높게 나타나는 것.
다음으로 TRS-4의 경우에서는 왜 TRS-1의 H condition보다 더 적은 TfR 발현양이 나타나는 것일까. 이는 stem의 일부 nucleotide가 바뀔 시 원래는 loop을 인식해야 할 factor가 stem loop을 인식하지 못하게 된 결과 mRNA의 안정성이 더욱 떨어져버렸기 때문임. 이와 관련해서 이해를 도울 만한 그림이 아래에 첨부되어 있음.

위 그림은 WT에서 일어나는 일을 보여주고 있음. 우선 iron 농도가 낮은 경우에는 stem loop에 주황색 단백질(iron-regulatory protein[IRP])이 붙게 되고 그 결과 주변에 RNase가 얼씬하지 못해서 결국 mRNA의 stability가 높아진 결과 TfR 발현양이 늘어남.
한편 iron 농도가 높을 경우 IRP에 Fe가 결합하여 conformational change가 일어난 결과 IRP가 stem loop을 인지하지 못하게 됨. 그러면 결국 RNase에 의해서 poly A 부분이 잘려나가게 되고, mRNA의 stability가 떨어져 TfR 발현양이 감소하게 됨.
참고로 이 때 IRP가 결합하지 않은 상태에서 존재하고 있는 stem loop는 linear한 구조에 비해 훨씬 더 RNase에 sensitive해지기 때문에 poly A tail이 잘 잘려나감.
결국 이 mechanism이 TRS-4에서는 일어나지 못함. 즉, IRP가 애초에 loop를 인식하지 못함. 한편 stem loop는 남아 있기에 RNase에 더 sensitive해짐. 그 결과 mRNA는 RNase에 의해 많이 잘려나갈 것이고 WT보다 더 mRNA의 stability가 낮아지게 될 것임.
다음 포스트에서는 RNAi(RNA interference)에 대해 알아보도록 하자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 16.5 : piRNA, miRNA에 의한 조절 (0) | 2023.12.21 |
|---|---|
| [분자생물학] 16.4 : RNAi (RNA interference, RNA 간섭) (0) | 2023.12.21 |
| [분자생물학] 16.2 : RNA editing (0) | 2023.12.21 |
| [분자생물학] 16.1 : ribosomal, transfer RNA processing, trans-splicing (0) | 2023.12.21 |
| [분자생물학] 15.5 : mRNA processing과 전사의 연관성 - 2 (0) | 2023.12.21 |