이번 포스트에서는 eukaryote에서의 재조합 기작에 대해 살펴보자.

위 그림에 진핵생물의 meiotic recombination의 대략적인 과정이 나타나 있음.
일단 이 경우 처음에 dsDNA의 break가 일어나게 됨. 이후 5' resection이 일어나고, 이어서 3' overhang의 invasion에 의해 D-loop가 형성됨. 그 다음에는 DNA repair synthesis에 의해 새로운 가닥이 합성되고, 뒤이어 ligation이 일어난 뒤 BLM, WRN과 같은 helicase에 의해 junction migration이 일어남. 이후 resolution 과정에 의해 앞서와 동일하게 noncrossover ,혹은 crossover recombinant가 만들어지게 됨.
참고로 이 때 BLM, WRN과 같은 helicase에 이상이 생기게 되면 조로증이 발생할 수 있음.
그렇다면 위 그림상에 나타나 있는 첫 step, 즉 dsDNA가 break되는 과정은 어떻게 해서 일어나는 것일까. 이는 Spo11라는 DNA cleavage 활성을 가지고 있는 녀석에 의해 일어남.

구체적으로는 Spo11 내부에 있는 Tyr이 가지고 있는 OH가 phosphodiester bond를 공격해서 끊어주게 됨. 이 과정에서 결국 protein와 DNA사이의 crosslinking이 일어남. 이런 상태로 어느 정도 존재하다가, 결국 뒤이어 살펴볼 processing 과정에 의해서 특정 DNA 부분이 끊어지게 되면(위 그림 아래에서 두 번째를 보면 끊어진 서열을 확인 가능) 결국 끊어진 서열과 함께 Spo11도 분리되어 빠져나가게 됨. 그 결과 위 그림 맨 아래에 나와있는 것과 같이 3' protruding end가 형성되게 됨.

이와 관련된 실험 결과가 위 그림에 나타나 있음.
일단 (a)와 같이 SPO11이 인지 가능한 DSB site를 2개 함유하고 있는 DNA를 사용함. 한편 이 DNA를 표지해주기 위해 DNA의 일부와 상보적으로 결합하는 probe도 만들어줌. (이 probe는 b, c에서만 사용한 것)
이런 상태에서 우선 (b)의 경우 전체 DNA와 함께 SPO11 혹은 HA tagging이 된 SPO11을 넣었을 때 나오는 모든 DNA band를 관찰한 것임. 이 결과를 보면 site I이 잘려 생기는 band, site II가 잘려 생기는 band, parent(PstI에 의해 잘린, 전체 DNA 조각)에 의해 생기는 band가 모두 나타난다는 것을 알 수 있음.
다음으로 (b)의 경우에는 HA tag와 결합 가능한 antibody인 alpha-HA를 사용해서 IP를 수행한 다음에 Southern blot을 수행한 결과임. 보면 site I band, site II band만 관찰됨을 확인 가능함. 이를 통해 SPO11이 DSB site 부분에 결합하여 DSB site-SPO11 adduct를 형성한다는 것을 확인가능함.
마지막으로 위 그림 맨 오른쪽의 결과는 alpha-HA를 사용해 IP를 수행한 다음에 probe 대신 그냥 DNA 서열 자체를 oligo labeling한 뒤에 SDS-PAGE를 수행한 결과임. (참고로 이 때 Y135F-HA는 SPO11의 activity를 망가뜨리기 위해 유발한 mutation임) 결과를 보면 한 쪽 끝부터 site I까지의 조각, 한쪽 끝부터 site II까지의 조각 두 개가 SPO11과 함께 결합된 채로 band로 관찰된다는 것을 확인 가능함.
참고로 위 그림 아래에는 oligo labeling을 수행하기 위해 필요한 TdT(terminal deoxynucleotidyl transferase)와 labeled dATP가 나타나 있음. TdT와 함께 dATP를 넣어주게 되면 dATP는 3'에 OH가 없으므로 결국 3' 말단에 딱 labeled dATP만 들어간 채로 합성이 끝나게 됨. 그러면 oligo labeling을 수행할 수 있음.
(본 내용과는 크게 관련되어 있지 않지만 apoptosis 시 일어나는 DNA fragmentation을 확인하기 위해서도 TdT system을 사용할 수 있음. TdT system을 사용하면 결과적으로 얼마나 많은 수의 fragment가 생기는지 여부를 정량적으로 확인할 수 있으므로 apoptosis 여부와 정도를 파악할 수 있게 됨)
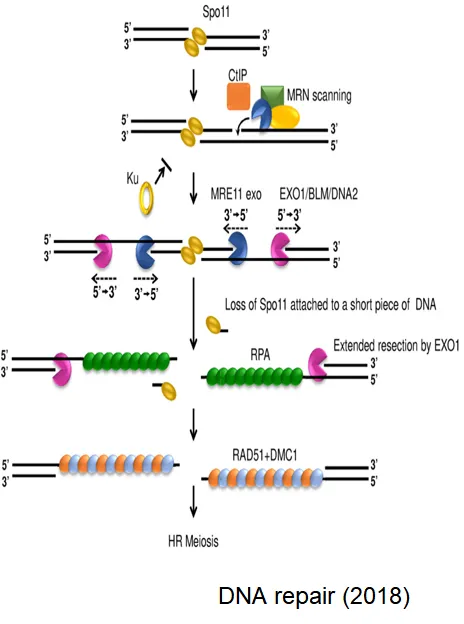
한편 위 그림에는 dsDNA breakage가 일어나고 난 뒤에 meiotic recombination이 어떤 식으로 진행되는지의 과정이 나타나 있음. 대부분의 과정은 앞서 DSB repair 과정에서 본 것과 비슷함. 다만 이 때 DNA 절단부위에 Ku가 결합하지 못한다는 것이 다른데, 절단부위 부분에 남아있는 SPO11이 Ku의 결합을 block해줌.
그리고 CtIP와 MRN scanning 과정에서 결국 nick이 만들어지게 되고, 이 부분을 기점으로 MRE11 exonuclease(3'→5') 혹은 EXO1/BLM/DNA2 exonuclease complex(5'→3') 들에 의해 끝부분이 점점 더 많이 잘려나가는 과정에서 앞서 봤던 것처럼 SPO11과 이 녀석에 결합되어있던 DNA 일부가 같이 떨어져 나가게 됨.
이후 ssDNA는 RPA에 의해 잠시 coating되었다가 이후 RAD51, DMC1과 같은 단백질들이 ssDNA에 달라붙으면서 본격적으로 displacement에 이은 D-loop의 형성이 일어나게 됨.
결론적으로 지금까지 살펴본 여러 종류의 HR 과정은 시작만 다를 뿐 후반부의 기작은 거의 비슷하다는 것을 기억해두면 됨.

이제 마지막으로 HR을 이용한 gene editing에 대해 알아보자.
위 그림에 가장 많이 쓰이는 3가지 종류의 유전자 가위가 나타나 있으며, 이 녀석들에 의해 인위적으로 특정한 위치에 DSB가 발생하게 됨. 그러면 DSB repair 기작인 HR 혹은 NHEJ에 의해 repair가 일어나게 되고, 이 과정에서 우리가 원하는 editing 효과를 얻을 수 있게 됨.
하나하나 살펴보자. 우선 1세대 가위로도 불리는 Zinc-finger nuclease의 경우 1개의 zinc-finger motif가 3개의 nucleotide를 인지하게 되고, 이들 motif들의 조합에 의해 random한 부분을 자르는 endonuclease의 일종인 Fok-1을 특정 자리로 recruit하게 됨. (즉, motif들이 Fok-1을 guide하는 역할)
다음으로 TALEN에 대해 살펴보자. 이 경우 식물에 감염되는 virus가 가지는 sequence에 착안해서 특정 nucleotide 하나를 인지할 수 있는 TAL effector들을 많이 연결해서 guide로 사용해주게 됨. (이 때도 Fok-1의 guide 역할을 해주는 녀석이 TAL effector라는 것만 빼면 전반적인 원리는 동일함)
마지막으로 CRISPR에 대해 알아보자. 이 녀석의 경우 sgRNA(single strand guide RNA)와 Cas9이 만나 이루는 complex에 의해 PAM이라고 하는 특정 sequence가 인지되고, 그 밖에 crRNA에 의해서도 특정 sequence가 인지된 결과 Cas9의 nuclease 활성에 의해서 특정 부분을 잘라 blunt end를 만들어버림.
일단 이런 유전자 가위들에 의해 DSB가 생성되고 나면 repair 과정에서 실험자가 원하는 서열을 특정 위치에 삽입하거나, 혹은 특정 부분을 deletion하는 등의 조작이 가능함.
다음 포스트부터는 transposition(전위)에 대해 알아보자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 23.2 : DNA transposon - 1 (0) | 2024.11.03 |
|---|---|
| [분자생물학] 23.1 : 전위(transposition) (0) | 2024.11.02 |
| [분자생물학] 22.2 : bacteria에서의 재조합 기작 - 2 (0) | 2024.11.01 |
| [분자생물학] 22.2 : bacteria에서의 재조합 기작 - 1 (0) | 2024.11.01 |
| [분자생물학] 22.1 : homologous recombination (상동재조합) (0) | 2024.11.01 |