이번 포스트에서는 지난 포스트에 이어 bacteria에서의 재조합 기작에 대해 자세히 알아보자.
holliday junction을 resolving해주는데 관여하는 RuvA, RuvB, RuvC에 대해 조금 더 자세히 알아보자. 일단 resolving 과정에서 RuvA, RuvB가 먼저 holliday junction 부위에 붙은 후 뒤이어 RuvC가 붙으며 resolving이 일어남.

우선 RuvA, B의 경우 DNA helicase 활성을 가지고 있어서 branch migration에 관여함. 위 그림을 보면 RuvA가 junction 부분에 결합하고 B가 A의 좌우로 결합하고 있음을 알 수 있음. 이 때 특히 RuvB가 ATPase 활성을 바탕으로 DNA를 unwinding시켜주게 되는데(즉, helicase로 작용하게 되는데), 이 과정에서 위 그림에서도 나타난 것과 같이 결과적으로 branch migration이 일어남. 이 때 B가 어떤 식으로 위치하게 되는지를 볼 필요가 있는데, (b)와 같은 position에서는 B가 다른 방향으로 움직여야 하지만, 결국 (c)처럼 중간부분의 branch를 한번 꼬아주게 되면 2개의 B가 서로 같은 방향으로 이동할 수 있게 됨. 이 때 RuvB의 이동에 의해서 junction의 migration이 일어나게 됨.

위 그림에는 RuvA, RuvB와 관련된 실험 결과가 나타나 있음.
일단 이 때 Junction으로 표시된 sample들(a, b, c, d, e, f, g, h)은 위 그림 왼쪽 아래와 같은 junction이 있는 서열을 넣어준 경우이고 Duplex로 표시된 sample들(i, j, k, l)의 경우에는 junction이 없는 단순한 형태의 duplex를 넣어준 경우임. 일단 duplex를 넣어준 경우에는 RuvA, RuvB를 넣어주나 넣어주지 않으나 별도의 gel mobility shift가 일어나지 않음. 즉, 일반적인 duplex DNA에는 RuvA, RuvB가 결합하지 않음. 다음으로 Junction이 있는 DNA sample을 넣어주었을 때의 실험 결과를 살펴보자. 일단 이 경우 RuvA만을 넣어준 b, c의 경우에서는 RuvA/DNA complex에 해당하는 shift band가 관찰됨. 즉, RuvA는 독자적으로 junction DNA에 결합 가능함.
한편 d, e에서와 같이 RuvB만을 넣어준 경우에는 shift band가 관찰되지 않음. 즉, 이를 통해 RuvB는 독자적으로 junction DNA에 binding하지 못한다는 것을 알 수 있음. 그리고 이어서 f, g를 보면 RuvA, RuvB를 다 넣어줄 경우 비로소 RuvAB/DNA complex의 형성에 따른 shift band가 관찰됨. 이를 통해서 RuvB는 RuvA가 먼저 붙고 나면 RuvA dependent하게 DNA와 binding할 수 있다는 것을 알 수 있음. 한편 h를 보면 RuvA, B와 함께 ATPgammaS를 넣어주지 않은 경우에(나머지 sample에서는 ATPgammaS가 다 들어있음. 이 때 정상적인 ATP 대신 ATPgammaS를 다른 sample들에 넣어준 이유는 migration을 stop시켜놓기 위해서임) RuvA/DNA complex의 band는 생기지만 B는 DNA와 결합하지 못하는 것을 알 수 있음. 이를 통해서 RuvB가 DNA와 binding하기 위해서는 ATP 자체가 필요하다는 것을 알 수 있음.
다음으로 RuvC의 역할에 대해 알아보자. 일단 RuvC는 endonuclease 활성을 가져서 DNA를 cutting해주는 역할을 수행하며 특히나 cut을 통해서 holliday junction을 resolve해준다고 해서 RuvC를 resolvase라고도 부름. 이 때 RuvC에 의한 resolution이 일어나기 위해서는 일단 branch migration이 선행되어야 하므로, 결과적으로 resolution 과정에는 RuvA, B, C가 다 필요하다고도 볼 수 있음. 참고로 RuvC는 5'-(A/T)TT(G/C)-3'의 서열에 preference를 가지고 있어서 이 부분을 더 잘 잘라주게 됨.

위 그림에는 RuvC와 관련된 실험 결과가 나타나 있음.
일단 위 그림 중 (b) 실험 결과의 경우에는 MgCl2 deletion, 낮은 온조의 조건에서 실험을 수행함. 이 조건에서는 결국 RuvC의 resolvase activity가 억제되게 됨. 이 때의 결과를 살펴보면 Junction DNA sample과 함께 서서히 많은 양의 RuvC를 넣어준 경우 일단 Junction DNA와 RuvC가 결합해서 생긴 shift band는 관찰됨을 알 수 있음. (complex로 표시된 부분) 그러나 RuvC 활성에 의해 잘려나간 DNA 조각은 관찰되지 않음. 즉, 이 실험 과정에서는 RuvC와 junction 부분이 결합하기는 하지만, 이후 RuvC에 의한 효소반응이 일어나지 않아 junction 부위에 RuvC가 계속 결합한 채로 존재하는 것임. 한편 (c)의 경우 MgCl2을 넣어주고 37도에서 실험을 수행함. 이 경우 앞서와 동일한 실험을 수행할 시 complex 부근의 band는 관찰되지 않고 RuvC가 작용해서 생성된 Duplex 부근에 band가 발생한다는 것을 확인할 수 있음. 즉, 이 실험과정에서는 RuvC가 junction에 붙은 후 resolvase로 기능해서 resolution이 일어나게 되고, resolution 과정을 통해 duplex DNA가 형성되면 RuvC는 다시금 떨어져나간다는 것을 알 수 있음.

실제로는 위 그림과 같이 1, 2 부분이 잘리느냐, 3, 4 부분이 잘리느냐에 따라서 서로 다른 product가 생성됨. 참고로 RuvC에 의해서 1, 2의 cut, 3, 4의 cut이 모두 일어날 수 있음.
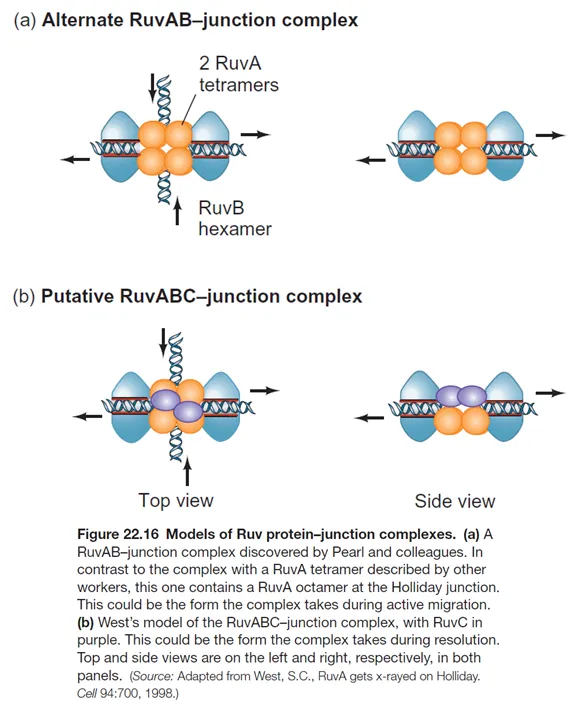
위 그림은 일종의 RuvABC 결합 과정에서의 model을 보여주고 있음. 보면 처음에 RuvA tetramer 2개(즉, octamer)가 junction 부위에 붙은 뒤에 RuvB hexamer가 결합함. 이후 RuvA에서 tetramer 하나가 떨어져 나오게 되고, 이 자리에 RuvC가 결합하여 존재하게 됨.
다음 포스트에서는 eukaryote에서의 재조합 기작에 대해 알아보자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 23.1 : 전위(transposition) (0) | 2024.11.02 |
|---|---|
| [분자생물학] 22.3 : eukaryote에서의 재조합 기작 (0) | 2024.11.01 |
| [분자생물학] 22.2 : bacteria에서의 재조합 기작 - 1 (0) | 2024.11.01 |
| [분자생물학] 22.1 : homologous recombination (상동재조합) (0) | 2024.11.01 |
| [분자생물학] 21.3 : translesion (0) | 2024.11.01 |