이번 포스트에서는 activator들간의 각종 상호작용들에 대해 알아보도록 하자.
activator들은 다양한 general transcription factor들, 또 다른 activator들, co-activator 등과 상호작용 해야 비로소 앞서 논의한 것과 같이 전사를 조절할 수 있음. 지금부터 이 interaction에 대해 알아보자.
우선 하나의 activator 안에서 일어나는 대표적인 interaction이 dimerization임. 앞에서도 언급한 것처럼 대부분의 activator들은 dimer를 이룸. 그러면 activator가 dimer를 이루었을 때 좋은 점은 무엇일까. 일단 monomer 형태로 DNA에 결합하는 것에 비해 당연히 dimer를 이루게 되면(작용 가능한 부위가 넓어지므로) affinity, 즉 결합력이 높아질 것임.
이와 더불어 protein이 인지하는 DNA 서열의 대부분은 회문 구조로 이루어져 있는 경우가 많음. 이 회문 구조를 monomer가 인식하는 것보다는, dimer가 인식하는 것이 훨씬 유리할 것임. (가운데를 중심으로 서열이 거울상대칭적이므로 dimer가 각각 한쪽씩을 인식하는 것이 가능해질 것임)
참고로 dimer를 이룰 때 어떤 activator들은 homodimer(동일한 두 monomer끼리 dimerization)를 이루는 경우도 있고 어떤 activator들은 heterodimer(서로 다른 두 monomer끼리 dimerization)를 이루는 경우도 있음.
한편 bacterial enhancer와 eukaryotic enhancer들은 position independent, orientation independent한 특징을 가지고 있음. 이 때 이 녀석들이 position independent하므로, 이 녀석들에 붙는 activator도 position independent하게 전사 촉진에 관여해야 함. 그렇다면 원거리에서 enhancer에 붙은 activator가 전사 조절에 관여하는 기작은 무엇일까. 이와 관련해서 크게 4가지 model(혹은 hypothesis)이 존재함.
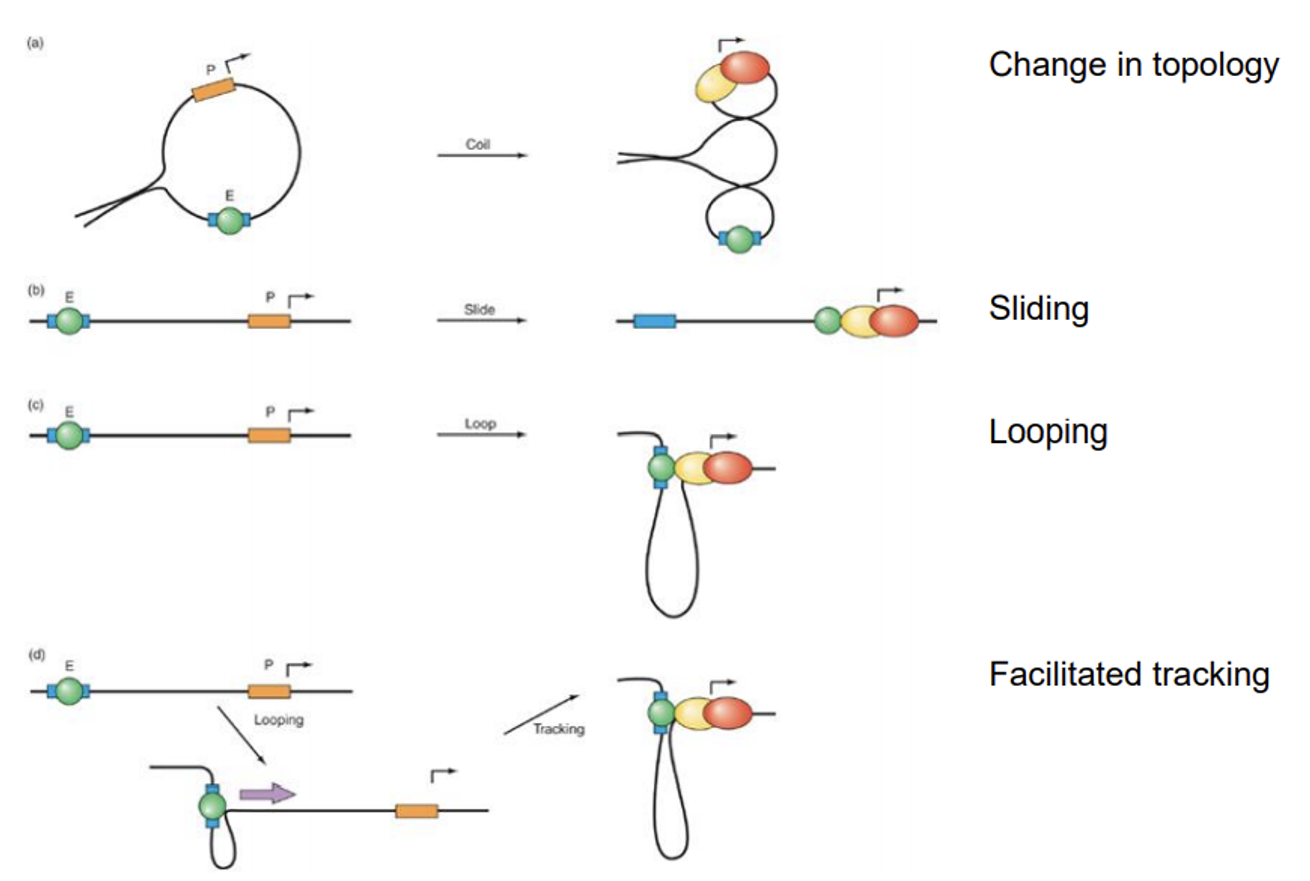
위 그림에 4가지 model이 나타나 있음.
(a)는 change in topology로, 그림에서 나타난 것처럼 enhancer에 activator가 붙으면 이 activator에 의해 DNA 전체의 topology가 바뀌어서 (위 그림에서는 DNA의 supercoil이 일어남) 바뀐 topology에 의해 전사가 물리적으로 촉진된다는 model임.
(b)는 sliding으로, enhancer에 activator가 붙고 나면 activator가 enhancer부터 promoter까지의 DNA 서열을 쭉 미끄러져 내려오다가 promoter를 만나게 되면 그곳에서 여러 전사에 관여하는 단백질들을 recruit한다는 model임.
(c)는 looping으로, enhancer에 activator가 붙고 나면 enhancer와 promoter 사이의 DNA 부분에 대해 loop가 형성되어서 물리적으로 activator와 promoter의 거리가 가까워지게 되고, 이후 recruit가 일어난다는 model임.
(d)는 facilitated tracking으로, 이 경우 enhancer에 붙은 activator가 DNA를 그림에서 나타난 것처럼 쭉 훑으면서 가다가 결과적으로 promoter 부분에 도달하면 loop를 형성한 모양새가 되어 (c)와 동일한 효과를 발생시킨다는 model임.
이 때 조금만 생각해보면 (a)와 (b)는 enhancer와 promoter가 같은 DNA molecule 상에 있어야 작용 가능함. 한편 (c)와 (d)는 enhancer와 promoter가 굳이 같은 molecule 상에 있을 필요는 없음. 이를 이용해 (a), (b) 혹은 (c), (d) 중 어느 model이 맞는지에 대한 검증을 목적으로 하는 실험을 수행할 수 있음.
이에 대한 검증을 위해 2개의 서로 독립적인 plasmid를 사용함. 그런데 이 둘을 어떻게든 가까운 거리에 고정시키는 경우를 만들어야 (a), (b)가 맞는지, (c), (d)가 맞는지를 판별할 수 있음. 이를 위해 아래 그림과 같은 catenane을 제작함.

보면 두 plasmid의 가운데가 얽혀 있는 것을 볼 수 있음. 이런 catenane을 만들면 서로 다른 DNA molecule에 있으나 promoter와 enhancer의 거리는 가까울 때 전사가 촉진되는지, 촉진되지 않는지 여부를 확인할 수 있음.

구체적인 실험 결과는 위와 같음. 하나하나 살펴보자.
우선 본격적으로 실험 결과를 살펴보기 전에 주의해야 할 점이 있음. 위 그림에서는 크게 두 종류의 plasmid를 사용하였고, 각각의 plasmid는 Ψ40, Ψ52의 서로 다른 gene을 함유하고 있음. 이 때 Ψ52는 모든 sample에서 아무런 처리를 해주지 않음. 즉, Ψ52에 의한 gene expression 정도가 일종의 reference이며, 이 reference를 바탕으로 상대적인 Ψ40의 전사 정도를 파악하게 됨. (특히나 이 경우 1, 2, 3, 4, 5, 6 sample을 각각 다른 xenopus oocyte에 넣어줬는데, oocyte 종류에 따라 gene expression 정도가 각기 다르므로 sample 끼리의 data 비교는 의미가 없고, 한 sample에서 Ψ52에 대한 상대적인 Ψ40의 발현 비율을 보는 것이 바람직함)
1부터 살펴보자. 1의 경우 두 plasmid에 아무런 처리를 가해주지 않은 상태에서 그냥 발현 정도를 비교한 것임. 결과를 보면 Ψ52가 Ψ40보다 상대적으로 더 많이 발현하고 있음을 알 수 있음. (즉, 유전자 발현 정도 관점에서 Ψ52> Ψ40임)
2에서는 Ψ40 plasmid에 (특별히 Ψ40 gene과 가까운 부분에) enhancer를 삽입한 채 실험을 진행함. 그 결과 Ψ52에 비해 압도적으로 많은 양의 Ψ40 발현이 관찰됨. 즉, enhancer에 의해 Ψ40의 activation이 관찰됨.
3에서도 Ψ40 plasmid에 enhancer를 삽입하되, 이번에는 2에서보다 조금 더 먼 거리에 enhancer를 삽입해봄. 그 결과 2보다는 덜하지만 그래도 1에 비해 Ψ52과 비교해서 꽤 많은 양의 Ψ40 발현이 이루어졌음을 확인 가능함.
4가 중요한 data인데, 4에서는 앞서 말한 catenane 형태로 Ψ40을 함유하는 plasmid와 enhancer를 함유하는 plasmid를 서로 엮어줌. 그랬더니 결과적으로 Ψ52에 비해 압도적으로 많은 양의 Ψ40 발현이 관찰됨. (2에서와 거의 identical한 효과) 즉, 서로 다른 DNA molecule에 있더라도 거리만 가까우면 enhancer가 전사를 촉진시킬 수 있음.
5에서는 그냥 Ψ40을 함유한 plasmid와 enhancer를 함유한 plasmid를 독립적으로 넣어줌. 그랬더니 1과 유사한 결과가 나타남. 즉, 이런 상태로는 enhancer가 전사를 촉진시키지 못한 것임.
이 결과를 통해 enhancer와 promoter가 비록 다른 DNA molecule에 있더라도 거리만 충분히 가까우면 enhancer에 의한 전사 activation이 일어날 수 있다는 결론을 내릴 수 있음. 따라서 결국 앞서 본 4가지 model 중에 (c), (d)가 맞는 model임을 검증 가능함.
다만 (c)와 (d) 중 어느 것이 맞는지를 검증하는 것은 꽤나 어려움. 대신 (c), (d) 둘 다에서 특징적으로 형성되는 것이 DNA loop이므로 정말 DNA loop이 형성되는지를 확인해보는 실험을 통해 (c), (d) model이 맞음을 한 번 더 검증할수는 있음.
이를 위해 chromosome conformation capture(3C) 방식을 사용함. 3C는 DNA가 특정한 구조를 가지고 있을 때 이를 detection해내는 방법임.

위 그림은 3C 방식의 원리를 나타내주고 있음. 예를 들어 위 그림과 같이 초록-노랑 단백질에 의해 서로 다른 파랑, 빨강 DNA strand가 서로 가까이에 있다고 하자. 이 때 이들이 가까이 있는지 여부를 3C를 이용하면 확인할 수 있음.
구체적으로는 위 그림처럼 우선 DNA 사이를 (포름알데히드 등을 사용하여) cross-link시켜줌. 일단 cross-link를 시켜주고 나면 deproteinize를 시켜주더라도(즉, 단백질을 없애주더라도) 서로 가까이 위치했던 DNA들은 다 여전히 붙어있게 될 것임. 이후 이들을 특정 제한효소로 잘라준 후 ligase를 사용해주면(같은 제한효소로 잘린 절단부위들은 서로 상보적인 sticky end를 가질 것이므로 ligase를 처리해줄 시 서로 붙어버릴 것임)
위 그림과 같은 연결된 loop 구조가 형성됨. 이후 이를 PCR하게 되면(특히 우리가 파란색과 빨간색 DNA가 가까이 있는지를 의심하고 있는 경우 파란색에 해당하는 primer, 빨간색에 해당하는 primer를 하나씩 사용함) 위 그림과 같이 loop 형성에 의해 생기는 빨간색-파란색 혼성체 DNA가 존재하는지 여부를 검증할 수 있음. (저런 혼성체가 생긴 경우에만 비로소 PCR 반응이 제대로 일어나 결과물이 얻어질 것임)
이런 방식을 이용하면 앞서 (c), (d) model에서 관찰한 것과 같은 loop 구조의 여부도 확인할 수 있을 것임. (loop를 형성했다는 것은 결국 특정 두 DNA strand가 protein들의 cooperative binding을 매개로 해서 가까이 위치해있다는 뜻이므로 3C로 이에 대한 실험을 수행할 수 있음)

결과적으로 위 그림과 같은 실험을 수행함. 특히 이 실험에서는 IGF2(interferon-like growth factor 2) gene의 앞쪽 부위에 대한 looping 여부를 3C를 이용해 확인함. 뒤에서 다시 한 번 더 설명이 되겠지만 이 IGF2 gene은 특이하게도 male에서만 turn-on되어 있고 female에서는 silenced되어있는 독특한 gene 중 하나임. 후에 looping 여부와 이 특성들이 matching되는 결과를 살펴볼 것임.
우선 (b), (c)는 각각 muscle cell과 liver cell에서 수행한 3C의 결과인데, 각각에서 3C 과정에서 사용한 PCR primer set이 다름. 위 그림 (a)에 작은 화살표로 표시된 1~13까지가 모두 primer의 위치인데, 그 중 muscle cell, liver cell의 enhancer 위치를 고려해서 primer set을 선택함.
보면 muscle cell에서 active한 enhancer는 붉은색으로 표시된 부분이고 liver cell에서 active한 enhancer는 노란색으로 표시된 부분이므로, 이들 각각의 위치를 고려해서 liver cell에서는 2-9, 4-9, 5-10 pair의 primer를 사용해서 이 부위들이 가까이 위치하는지 여부를 확인했으며 muscle cell에서는 1-13, 4-11, 5-12 pair의 primer를 사용해서 이 부위들이 가까이 위치하는지 여부를 확인함.
그 결과가 각각 (b), (c)의 맨 윗줄 부분에 나타나 있음. 보면 전반적으로 모두 PCR product가 관찰됨. 이를 통해 실험한 부위 모두가 loop을 형성해서 가까이에 위치하고 있었음을 확인할 수 있음. (다시 말해 이 전체 부분이 공간상에서 매우 가까이 있음을 확인함)
더 나아가서 이 실험에서는 두 가지 다른 종류의 종을 교배한 자손종을 사용함. 위 그림에서 C, D로 나타난 것들이 서로 다른 종이며, 다만 C와 D간의 교배는 가능함. C, D는 종이 다르므로 restriction fragment length polymorphism이 나타남. 즉, 3C 과정에서 restriction enzyme을 처리했을때 C, D 종에 따라 잘리는 pattern이 다르게 나타남.
한편 위 그림에서 C/D라고 표시된 것도 있고 D/C라고 표시된 것도 있는데 앞쪽에 오는 것이 female, 뒤쪽에 오는 것이 male임. 이 때 놀랍게도 3C를 통해 loop가 형성되었을 때만 signal이 나타나게끔 한 결과물을 바탕으로 전기영동 분석을 해 본 결과 모두 male의 restriction fragment length pattern으로 나타남. (위 그림에서 화살표로 표시된 D specific, C specific band pattern을 참고할 것) 따라서 loop이 형성되는 gene은 male의 것이라는 것을 알 수 있음.
결과적으로 male로부터 전해진 gene에서만 loop가 형성되고, looping의 결과로 male의 IGF2 gene만 activation되어서 활발하게 전사되는 것임. (female의 경우에는 looping이 일어나지 않고 그 결과 gene이 침묵화됨)
다음 포스트에서는 transcription factory, 그리고 enhancer와 관련된 다양한 개념들에 대해 살펴보자.
'전공자를 위한 생물학 > 분자생물학' 카테고리의 다른 글
| [분자생물학] 12.6 : insulator (0) | 2023.12.18 |
|---|---|
| [분자생물학] 12.5 : transcription factory, complex enhancers, etc (0) | 2023.12.17 |
| [분자생물학] 12.3 : activator의 기능 (0) | 2023.12.17 |
| [분자생물학] 12.2 : DNA-binding motif의 구조 - 2 (0) | 2023.12.17 |
| [분자생물학] 12.2 : DNA-binding motif의 구조 - 1 (0) | 2023.12.17 |