AlphaFold는 단백질의 아미노산 서열만 보고 3차원 구조를 예측하는 놀라운 AI 도구예요.
그런데 이게 도대체 어떻게 가능한 걸까요? 무작정 예측하는 건 아니고요, AlphaFold 내부에서는 두 개의 핵심 단계가 작동해요. 첫 번째는 진화적 정보를 해석하는 ‘Evoformer’, 두 번째는 구조를 실제로 그려내는 ‘Structural Stage’예요.
이번 글에서는 그중에서도 AlphaFold의 가장 독창적인 부분이라 불리는 Evoformer 단계에 대해 알아볼게요. 단백질의 진화적 힌트를 어떻게 모으고, 그걸 구조 예측에 어떻게 활용하는지를 함께 살펴보죠.
MSA와 Pairwise Features : 진화 정보의 단서들
AlphaFold는 아미노산 서열 하나만 보고 예측을 하진 않아요.

먼저 MSA(Multiple Sequence Alignment, 다중 서열 정렬)를 생성해요. 이건 간단히 말하면 ‘이 단백질과 비슷한 단백질 서열들을 모아서 정렬한 것’이에요. 비슷한 서열들이 어떤 아미노산 위치에서 변하지 않고 잘 보존되어 있다면, 그건 중요한 기능을 하는 위치일 가능성이 높죠.
이런 정보를 바탕으로, 각 아미노산 쌍(Residue pair) 간의 관계도 분석해요. 이를 Pairwise Features라고 해요. 어떤 아미노산 쌍이 진화적으로 함께 바뀌었다면, 실제 구조에서도 가까이 붙어 있을 가능성이 크다고 판단하는 거죠.
AlphaFold는 이 MSA와 Pairwise Features를 각각의 표현(Representation)으로 받아들이고, 그걸 반복적으로 다듬으면서 정보를 정제해요. 마치 퍼즐 조각을 맞추듯이 여러 층의 신경망을 거쳐 점점 더 정확한 단서들이 만들어지는 거예요.
Evoformer 블록 : 정보를 반복적으로 정제하는 두뇌
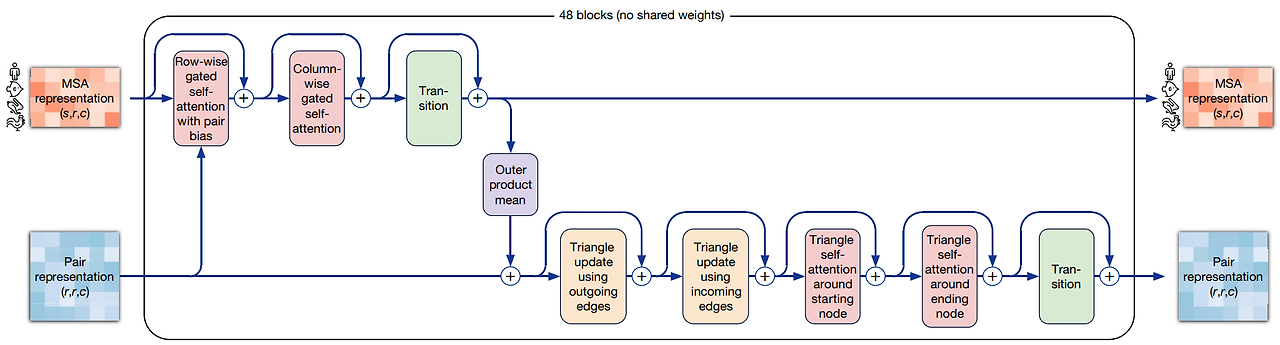
Evoformer는 이 모든 정보를 처리하는 AlphaFold 내부의 두뇌 같은 부분이에요. 여기서는 총 48번의 반복 과정이 일어나요. 각 반복에서는 MSA 표현과 Pairwise 표현이 서로 영향을 주고받으면서 정교해지죠.
먼저 MSA 표현은 Pairwise 정보를 사용해 자기 자신을 업데이트해요. 다시 말해, 아미노산 서열의 진화적 관계를 더 깊이 이해하도록 돕는 거예요. 그러고 나면, MSA가 Pairwise 표현에도 영향을 줘요. 이 과정을 통해 “이 아미노산 쌍은 구조적으로 가까워질 것 같아”라는 판단이 점점 구체화돼요.
특히 흥미로운 점은, 이 Pairwise 표현이 그래프 형태로 변환된다는 거예요. 아미노산 쌍이 각각 ‘노드’와 ‘엣지’로 표현되고, 이를 삼각형 단위로 연결해가며 구조적으로 자연스러운 형태를 만들어가요. 이 방식은 단백질 구조의 물리적 제약을 고려한 똑똑한 설계라고 할 수 있어요.
Evoformer는 단순히 데이터를 한 번 처리하고 끝내는 게 아니라, 반복적으로 수십 번 되풀이하면서 점점 더 정제된 정보를 만들어요. 이렇게 만들어진 진화 기반 정보 패키지는 다음 단계인 구조 예측으로 넘어가요.
다음 글에서는 AlphaFold가 어떻게 이 정보를 바탕으로 실제 3D 구조를 생성하는지, 그리고 Backbone, Rotation 같은 개념들이 어떤 식으로 사용되는지를 알아볼 거예요.
'전공자를 위한 생물학 > 대학원생을 위한 필수 생물학 개념들' 카테고리의 다른 글
| [알파폴드] 4편 : Google Colab으로 AlphaFold2 직접 사용해보기 - ColabFold (0) | 2025.04.20 |
|---|---|
| [알파폴드] 3편 : AlphaFold는 어떻게 작동할까 - 구조 예측 단계 (0) | 2025.04.20 |
| [알파폴드] 1편 : AlphaFold란 무엇인가? 단백질 구조 예측의 새로운 시대 (0) | 2025.04.20 |
| [TRE 시스템] 5편 : Tet 시스템의 다양한 응용법 (0) | 2025.04.19 |
| [TRE 시스템] 4편 : Tet 시스템 구축 가이드 - 플라스미드 구성부터 rtTA 선택까지 (0) | 2025.04.19 |