이번 포스트에서는 지난 포스트에 이어 구체적인 NGS 방식들에 대해 하나하나 알아보자.
우선 pyrosequencing에 대해 자세히 알아보자.

위 그림은 pyrosequencing의 원리를 나타내주고 있음. pyrosequencing은 Roche에서 2004년에 개발되었으며, 이 때의 상품명을 따 454 pyrosequencing으로도 부름.
보면 특정 종류의 dNTP를 넣었을 때 DNA가 elongation된다면, 이 때 pyrophosphate가 방출됨. 이 때 이 녀석에 ATP sulfrylase와 adenosine이 가해지게 되면 ATP로 바뀌게 됨. 이후 ATP가 input으로 들어가면서 luciferin이 oxyluciferin으로 변화하게 되면서 light이 방출됨. (이 때 luciferase라는 효소가 필요함)
그런데 이 때 signal 정보 그 자체만으로는 어떤 염기가 첨가되었는지를 알 수 없으므로 우리는 순서에 맞게 한 종류의 dNTP씩을 넣고 washing해가면서 light signal이 나오는지를 detection하는 방식으로 sequencing을 하게 됨.

위 그림은 pyrosequencing의 과정을 다시금 나타내주고 있음. 일단 보면 DNA 중합을 위해 polymerase가 필요하며, 그 밖에 앞서 발했던 것처럼 sulfurylase, luciferase와 같은 효소가 필요함. 이 밖에 Apyrase라는 효소도 필요한데, 이 효소는 한 cycle 이후 남아있는 nucleotide를 다 분해해주는, cleaning 과정에서 작용함.

위 그림은 실제로 이 방법을 통해 얻은 signal을 보여주고 있음. 보면 A, C, G, T의 순서대로 계속 반복적으로 dNTP를 넣어줘가며 light signal을 측정하게 됨.
이 때 signal이 1-mer의 2배, 3배, 혹은 그 이상으로 나오는 경우가 있는데(즉, pyrophosphate가 2배, 3배, 그 이상으로 나온다는 뜻) 이 경우 특정 염기가 연속적으로 2번, 3번, 혹은 그 이상 serial하게 나열되어 있음을 indicate함. 다만 실제로는 같은 nucleotide가 연속적으로 10개, 11개 있는 경우에는 위 방법을 이용해서 10개가 연속적으로 있는 것인지, 11개가 연속적으로 있는 것인지를 구분하기가 쉽지 않음. (signal 세기가 거의 차이 나지 않음) 이러한 단점은 뒤이어 살펴볼 ion semiconductor sequencing method도 공통적으로 가지는 단점임.
다음으로 ion semiconductor sequencing, 그 중에서도 ion torrent에 대해 알아보자.

이 경우 앞서와 동일하게 각 step마다 서로 다른 종류의 dNTP를 넣어주게 됨. 다만 이 때는 nucleotide 중합반응에서 (3' OH로부터) 방출되는 proton에 의해 미세하게 감소하는 pH를 electric signal로 바꾸어서 sensing하게 됨.
이제 마지막으로 reversible terminator sequencing method에 대해 알아보자.

위 그림에 나타나 있는 method는 reversible terminator를 사용하는 Illumina genome analyzer(HiSeq system)임. 이 platform은 위와 같이 solid support에 각각의 clone이 amplified되어 있는 상황에서 sequencing을 수행하는 방식으로 작동함. 그리고 이 경우 앞서와는 달리 4개의 nucleotide를 한번에 집어넣게 됨. (즉, cleaning이 불필요) 이 때 4개의 nucleotide는 각각 위 그림 가운데와 같이 각각 다른 색을 내게 됨. (그렇게 되도록 각 nucleotide에 각기 다른 fluorophore를 붙임)

실제로 위 그림을 보면 base마다 서로 다른 형광을 낼 수 있는 fluorophore를 붙여주게 됨. 한편 이와 동시에 이 경우 3' 위치의 OH를 아예 H로 바꿔서 ddNTP를 만들어버리는 것이 아니라 O를 별도의 protection group으로 protect하고 있는 상태로 넣어줌. 즉 이와 같이 3'이 protection된 상태로는 다음 round의 nucleotide가 들어오는 것이 불가능함.

보면 위와 같이 서로 다른 색을 가지고 있는 nucleotide를 한번에 넣어주게 됨. 이 때 각각의 strand들은 하나씩으로 표시되어 있지만 사실은 같은 sequence를 가지고 있는 DNA cluster임. 그리고 위 그림에 나타난 3개의 strand들은 모두 다른 strand들임.
보면 clone마다 어떤 nucleotide가 들어오는지는 다름. 그 결과 clone의 sequence에 따라 들어간 nucleotide가 다르고, 각 strand별로 나오는 light도 다르게 됨. 이후 다음 step에서는 fluorophore를 cleavage 후 wash한 이후에 3' 부분을 deprotection해주게 됨. 그럴 시 3' OH가 다시 regeneration되면서 결과적으로 다음 step에서 새로운 nucleotide가 들어오는 것이 가능함. 이렇듯 넣어주는 nucleotide가 reversible하게 다시 3' OH를 회복할 수 있는 녀석이기에 이 녀석을 reversible terminator라 함.
이런 방법을 이용할 시 한 step별로 하나씩의 nucleotide만 첨가된다는 확신을 가질 수 있으므로 연속해서 같은 nucleotide가 계속 나열되어 있어도 상당히 정확하게 sequencing을 하는 것이 가능함.

실제로는 위와 같이 dot 형태로 결과가 나옴. 이 때 cluster 하나(dot 하나)에 대해 cycle 진행에 따라 나오는 light을 쭉 나열할 시 sequence를 알아낼 수 있음. 이런 방법으로 sequencing을 한 결과가 위 그림 가운데에 나타나 있으므로 참고할 것.
다음으로 single-molecule next-generation sequencing에 대해 알아보자.

위 그림에는 oxford Nanopore technology가 나타나 있음. 이 경우 DNA polymerization 방식이 아니라 pore를 통과하는 긴 DNA가 pore 통과 과정에서 base에 따라서 서로 다른 signal을 유발하게 되고 이를 측정하는 방식으로 sequencing이 이루어짐. (즉, DNA를 새로 합성하면서 sequencing하는 것이 아니라 이미 합성된 DNA의 sequence를 말 그대로 read하는 것) 조금 더 구체적으로 설명하자면, 위 그림에서와 같이 이 경우 pore를 통과하는 base의 종류에 따라서 전류 변화의 미묘한 차이가 생기게 되었을 때 이를 detection하게 됨.

실제로는 위 그림 왼쪽에 나온 것과 같이 single-molecule nanopore의 가운데 구멍으로 DNA 서열을 통과시키게 됨. 이 때 위 그림 오른쪽과 같이 통과하는 base의 종류에 따라 전류 변화 양상이 각기 달라지게 됨. 이를 t축에 대해 real-time으로 측정해서 결과적으로 sequencing을 수행할 수 있게 되는 것임. 이 때 이 장비는 소형화가 가능해서 위 그림 오른쪽 아래와 같은 MinION처럼 휴대용 labtop에 연결해서 사용하는 형태로도 개발되어 있음.

다음으로 위 그림에 나타나 있는 것은 single-molecule real-time(SMRT) sequencing임. (Pacific BioSciences가 개발)
보면 이 경우 fluorophore가 leaving group인 γ phosphate에 달려있음. 그리고 3' OH는 protection되지 않은 그냥 normal한 OH임.
일단 위 그림과 같이 reaction chamber에 DNA polymerase를 embed시킴. 이 때 well의 depth가 상당히 얕음. (약 100nm) 그리고 맨 아래 바닥 부분은 유리로 되어 있음. 그런데 depth가 상당히 얕으므로 결국 polymerase에 의해 중합 반응이 일어나는, 딱 그 위치에서의 signal만이 detection될 수 있음. 이 상태에서 A, T, G, C를 각각 다른 fluorophore로 tag해서 한꺼번에 넣어주게 됨.
그럴 시 DNA polymerase에 의해 특정 DNA sequence가 합성될 때 딱 실시간으로 필요한 dNTP가 들어오게 되고, 이 때 fluoroscence가 측정될 것임. 한편 이 녀석이 중합 반응에 참여한 결과 pyrophosphate가 방출되고 그러면서 fluorophore도 위쪽으로 가버려서 금새 signal은 없어지게 됨. 이후 또 다른 nucleotide가 들어오며 fluorescence가 detection되게 됨.
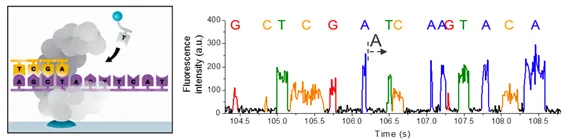
그 결과 위 그림 오른쪽과 같이 하나의 DNA polymerase에서 일어나는 한 가닥의 DNA 합성 과정에서 input으로 들어가는 nucleotide들을 실시간으로 detection할 수 있게 되는 것임. 따라서 위 그림 오른쪽과 같이 sequencing을 할 수 있게 됨.
다음 포스트부터는 DNA 관련 각종 기술들에 대해 알아보자.
'전공자를 위한 생물학 > 생화학' 카테고리의 다른 글
| [생화학] 8.1 : DNA 클로닝(cloning) - 2 (0) | 2024.11.09 |
|---|---|
| [생화학] 8.1 : DNA 클로닝(cloning) - 1 (0) | 2024.11.09 |
| [생화학] 7.10 : DNA 시퀀싱(DNA sequencing) - 2 (0) | 2024.11.09 |
| [생화학] 7.10 : DNA 시퀀싱(DNA sequencing) - 1 (0) | 2024.11.09 |
| [생화학] 7.9 : DNA 합성(DNA synthesis) (0) | 2024.11.09 |