[생화학] 8.6 : 유전자 편집(gene editing) - 2
이번 포스트에서는 CRISPR/Cas9에 대해 알아보자.

그러던 중 위 그림에 나타나 있는 것과 같이 연구자들이 bacteria의 adaptive immune system인 CRISPR를 이용해서 3세대 유전자 가위인 CRISPR/Cas9을 개발함.

위 그림에는 CRISPR biology와 관련된 발전의 계보도가 나타나 있음. 보면 CRISPR biology와 함께 genome editing과 관련된 기술도 지속적으로 발전해왔음을 알 수 있음.
CRISPR는 clustered regularly interspaced short palindromic repeats의 약자임. 이 system은 bacteria의 immune system으로 작용하며, bacteriophage의 DNA를 일정한 간격을 두고 내용적으로 저장하는 식으로 작동함.

위 그림은 실제로 bacteria가 가지고 있는 CRISPR/Cas adaptive immune system을 보여주고 있음. 보면 우선 한번 내부로 감염된 virus에 의한 viral DNA들이 위 그림 오른쪽과 같이 서로 palindromic repeat들 사이사이에 삽입되어 저장되어 있음. 그러다가 이 녀석이 mRNA로 발현되게 되고 이후 RNase III에 의해 modify되게 됨. 이렇게 해서 만들어지는 mature RNA가 바로 CRISPR RNA, 즉 crRNA가 되는 것임. 이후 이 crRNA는 bacteria 자체가 가지는 tracer RNA, Cas9 protein과 상호작용하게 되는데, 이 과정에서 crRNA가 indicate하는, viral DNA와 상보적으로 결합 가능한 viral DNA 파편들이 crRNA와 결합하게 된다면 이 결합에 의해 Cas9 protein이 crRNA에 의해 인지된 viral RNA를 파괴해버리게 됨. (즉, 이 방식은 한번 들어온 viral DNA가 이후 다시 들어왔을 때 작동하게 되는 적응면역 system임)

위 그림 왼쪽이 실제로 bacteria 내에서 작용하는 system을 보여주고 있는 것임. 보면 crispr RNA(target DNA와 binding)가 일단 tracer RNA(trans-activating crispr RNA)와 결합하고, 이어서 tracer RNA에 Cas9이 결합하게 되면서 결과적으로 Cas9이 crRNA와 상보적으로 결합하는 부분을 cut해버리게 되는 식으로 작동함.
그런데 연구자들이 이 system을 모방해서 위 그림 오른쪽과 같이 crRNA와 tracerRNA를 linker loop으로 이어버린 chimeric한 sgRNA를 만들어내게 됨. 그럴 시 실제로 5'에 있는 20nt의 서열과 상보적으로 결합하는 부위를 Cas9으로 specific하게 절단할 수 있는 system을 구현하는 것이 가능해짐.

위 그림은 Cas9이 인지하는 서열에 대한 정보를 제공해주고 있음. 보면 일단 crRNA에 해당하는 guide sequence(20bp), loop 등으로 이루어진 sgRNA가 있음. 한편 흥미롭게도 Cas9는 sgRNA를 인지할 뿐만 아니라 DNA의 PAM sequence도 인지하게 됨. 이 때 PAM이란 NGG, 즉 아무 nucleotide 다음에 GG가 오는 형태를 의미함. 이 서열이 Cas9에 의해 인지될 시 이 서열로부터 3개 nucleotide 뒤에 해당하는 부분이 blunt end로 잘려나가게 됨.

실제로 이 3세대 유전자 가위를 개발한 공로로 위 그림에 나타나 있는 것처럼 2020년에 두 명의 과학자에게 노벨 화학상이 수여됨.
그렇다면 이런 유전자 가위에 의해 DNA 절단이 일어나면 어떤 일이 일어나는가.
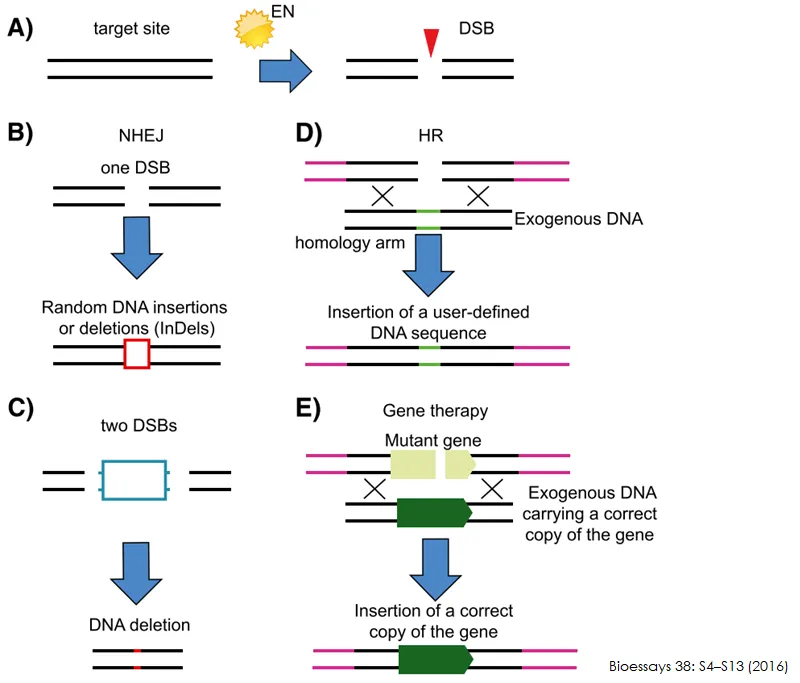
실제로 위 그림에 나타나 있는 것과 같은 repair 과정이 일어남. 그 중에서도 double strand DNA가 break되었을 경우(DSB) 위 그림에 나타나 있는 것처럼 NHEJ(non-homologous end joining), 혹은 HR(homologous recombination)에 의해 repair가 일어남. 이 중 NHEJ의 경우 processing의 과정에서 random하게 nucleotide가 손실되거나 insertion되는 과정이 존재함. 이렇게 될 시 결과적으로 잘라준 특정 부분의 서열이 이상해지게 되므로 gene 정보가 이상해져서 functional한 단백질 발현이 이루어지지 않게 됨.
한편 위 그림 오른쪽에 나타나 있는 HR의 경우 homology를 가진 exogenous DNA를 넣어주게 되면 이 서열을 참고해서 repair시킬 수 있게 됨. 따라서 위 그림상에서와 같이 초록색 유전자를 cut 부위에 삽입하고 싶다면 그 유전자가 중간에 삽입되어 있고 양쪽에 homology arm이 있는 exogenous DNA를 만들어서 유전자 가위 사용 이후 처리해주게 되면 HR 과정에 의해 이 서열이 cut 위치에 삽입되게 됨.
즉, 결과적으로 NHEJ를 통해서는 특정 부분을 deletion시키거나 이상하게 만들어버릴 수 있고 HR을 통해서는 우리가 원하는 서열을 insertion시킬 수 있음. (knock-in)
사실 최근에는 CRISPR/Cas9에서 Cas9의 nuclease activity는 turn-off시킨 상태에서 Cas9에 DNA repair와 관련된 protein을 같이 붙여줘서 DNA 서열 하나만을 다른 것으로 바꿔주는 base editing 방식도 많이 각광받고 있음.

다음으로, 위 그림에 나타나 있는 Feng Zhang은 CRISPR/Cas system을 genome wide하게 만들어주는 방식을 시도함.

그 결과 위와 같은 GeCKO library를 만들었는데, GeCKO는 genome-scale CRISPR knock-out이며, 이는 sgRNA를 조금씩 다 바꿔가며 30000여개의 gene과 상호작용할 수 있는 CRISPR/Cas system library를 만든 것임. 이 library 제작에 의해 CRISPR system의 상용화에 불이 붙음.

한편 앞서 말했던 것처럼 CRISPR system을 이용해 gene transcription을 activation시킬 수도 있음. 보면 위와 같이 작동을 못하게끔 조작한 Cas9에 VP64, P65, HSF1과 같은 transcriptional activator가 붙게 되고, 결과적으로 sgRNA와 상보적으로 결합하는 promoter 부위에 Cas9이 recruit되게 되면 transcriptional activator도 같이 recruit되어서 특정 gene의 전사를 촉진시켜줄 수 있음. 이러한 library를 SAM(synergistic activation mediators) library라 함.
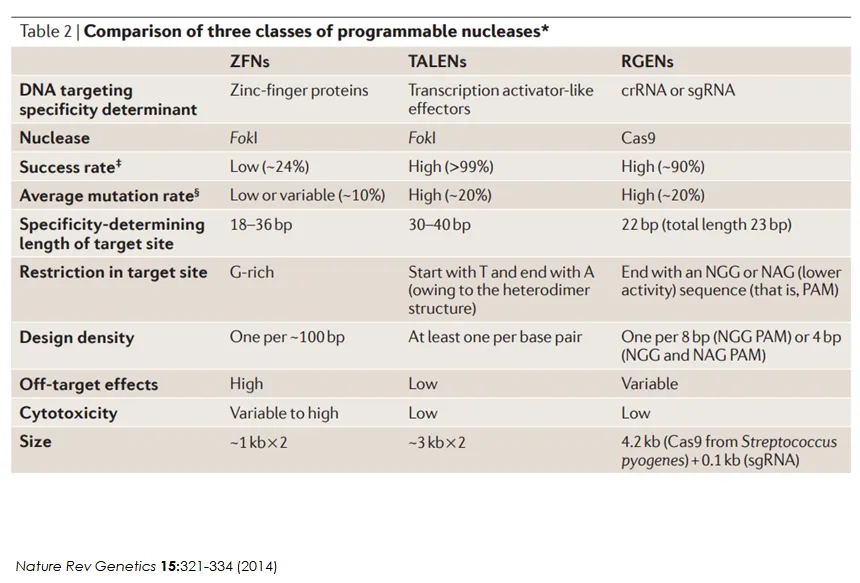
위 표는 3가지 세대의 유전자 가위를 비교해주고 있으므로 참고할 것.
다음 포스트부터는 지질(lipid)에 대해 알아보자.